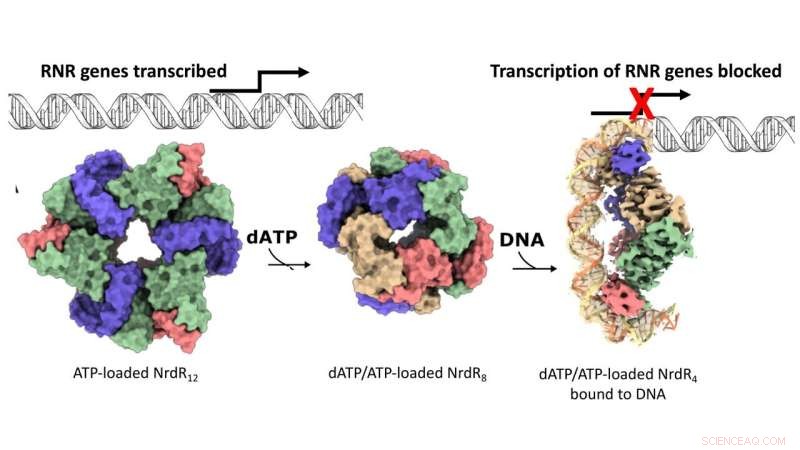
Il meccanismo d'azione del repressore specifico di RNR NrdR. Rappresentazione superficiale delle mappe crio-EM per le strutture NrdR tetrameriche dodecameriche, ottameriche e legate al DNA. I monomeri NrdR in ciascun gruppo tetramero sono colorati di beige, blu, verde e rosa. Il DNA è mostrato schematicamente, ad eccezione della parte risolta da cryo-EM in cui i due filamenti di DNA antiparallelo sono rispettivamente di colore arancione e giallo chiaro. Credito:Inna Rozman Grinberg
In un nuovo studio pubblicato su Nature Communications , i ricercatori dell'Università di Stoccolma mostrano per la prima volta come l'NrdR si lega al DNA per inibire l'RNR. Il nuovo meccanismo potrebbe aiutare gli scienziati a progettare antibiotici migliori prendendo di mira la capacità di riproduzione di un agente patogeno.
"Abbiamo scoperto la NrdR più di un decennio fa, ma il modo in cui funziona era un enigma. In questo articolo, abbiamo combinato studi biochimici e strutturali per scoprire come la NrdR si lega al DNA", afferma Inna Rozman Grinberg, ricercatrice presso il Dipartimento di Biochimica e Biofisica all'Università di Stoccolma e principale autore dello studio.
La sintesi del DNA è il processo di creazione di nuove molecole di DNA che avviene quando una cellula si divide. Praticamente in tutti gli organismi viventi, l'enzima ribonucleotide reduttasi (RNR) fornisce i mattoni per i nuovi filamenti di DNA. In molti batteri, inclusi noti agenti patogeni come il Mycobacterium tuberculosis, la sintesi di RNR è disattivata dalla proteina repressore NrdR. Tuttavia, il meccanismo alla base dell'interazione tra RNR e NrdR è rimasto sfuggente. In generale, quando le cellule batteriche si dividono, l'RNR viene attivato per fornire elementi costitutivi per la creazione di nuovo DNA. Nel frattempo, NrdR rimane spento. Quindi, quando la sintesi del DNA è completa, NrdR si lega al DNA impedendo un'ulteriore sintesi di RNR.
Ma come percepiscono le cellule quando è il momento di disattivare l'RNR? La risposta sta nel rapporto di concentrazione relativo tra due molecole chiave, vale a dire l'adenosina trifosfato (ATP) e la desossiadenosina trifosfato (dATP). I ricercatori hanno dimostrato che alcuni RNR possono spegnersi quando sono presenti livelli elevati di dATP. NrdR, d'altra parte, si lega al DNA e inibisce la sintesi di RNR solo quando i livelli di dATP sono alti. Questo meccanismo di rilevamento, utilizzato quasi esclusivamente da RNR e NrdR, è mediato da un dominio proteico chiamato "cono ATP".
"Questo meccanismo mette in evidenza un aspetto interessante dell'evoluzione delle proteine", afferma Inna Rozman Grinberg.
Secondo Inna Rozman Grinberg, poiché NrdR è abbondante nei batteri ma assente negli esseri umani, il cono ATP potrebbe essere utilizzato come bersaglio antibiotico contro agenti patogeni con un singolo RNR o contro agenti patogeni con NrdR per controllare tutti i loro RNR. "A nostra conoscenza, questo è l'unico esempio di un dominio proteico mobile evolutivo che controlla sia l'espressione che l'attività di un enzima". + Esplora ulteriormente