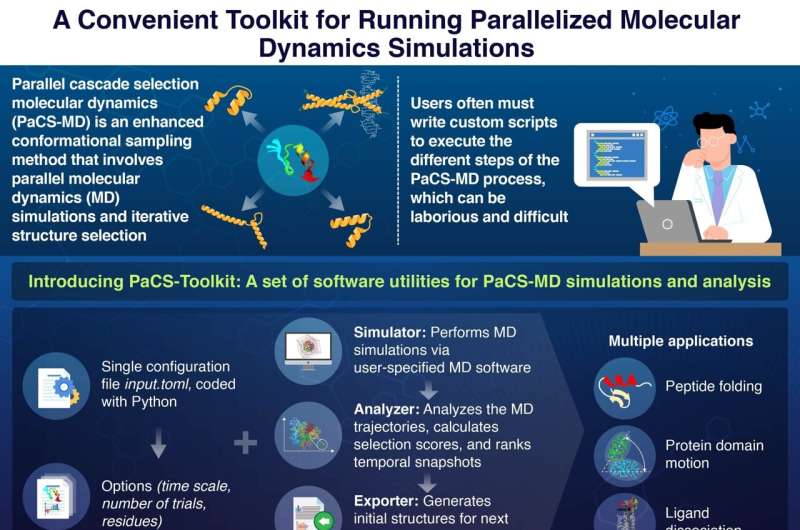
Le simulazioni di dinamica molecolare (MD) sono diventate uno strumento potente nei campi in continua crescita della biologia molecolare e dello sviluppo di farmaci. Sebbene esistano molte tecniche di simulazione MD, la MD con selezione a cascata parallela (PaCS-MD) è particolarmente utile quando si studia il ripiegamento (o "conformazione") delle proteine o le interazioni tra proteine e ligandi.
Il punto cruciale di questo metodo consiste nell'eseguire più simulazioni MD in parallelo, esplorando così simultaneamente diverse possibili conformazioni. Utilizzando criteri di selezione attentamente elaborati, le conformazioni promettenti possono essere rilevate automaticamente in "istantanee temporali" e studiate ulteriormente. Questa strategia accelera notevolmente la scoperta di interazioni molecolari chiave e processi dinamici che aiutano gli scienziati a comprendere i movimenti funzionali delle proteine.
Tuttavia, uno degli ostacoli di PaCS-MD è che gli utenti devono scrivere script personalizzati per eseguire le simulazioni MD desiderate. In questi script, devono specificare le condizioni iniziali e le caratteristiche target, selezionare il software MD da utilizzare, implementare una procedura di classificazione delle istantanee e preparare le strutture iniziali per il successivo ciclo di simulazione. Questo processo, che può essere piuttosto complesso e soggetto a errori, crea una notevole barriera all'ingresso per gli scienziati interessati a utilizzare PaCS-MD.
Fortunatamente, un team di ricercatori della School of Life Science and Technology del Tokyo Institute of Technology, in Giappone, ha recentemente deciso di affrontare questo problema. Nel loro ultimo studio, pubblicato su The Journal of Physical Chemistry B e guidato dal professor Akio Kitao, il team ha sviluppato un pacchetto software chiamato PaCS-Toolkit per rendere PaCS-MD più accessibile e comodo da usare.
Un notevole vantaggio di PaCS-Toolkit è che l'intero processo di simulazione viene impostato tramite un unico file di configurazione. In questo file, gli utenti specificano parametri importanti per le simulazioni, incluso il tipo di PaCS-MD, il numero di simulazioni MD da eseguire in parallelo e i residui proteici o gli atomi di cui tenere traccia come criteri di selezione per i rami paralleli.
PaCS-Toolkit prende questo file di configurazione, insieme ai file di input MD standard, ed esegue simulazioni PaCS-MD in base al software MD specificato. Vale la pena notare che, poiché il pacchetto è un software aperto scritto in Python, un popolare linguaggio di programmazione, gli utenti possono contribuire a migliorare PaCS-Toolkit ed estenderne le funzionalità.
"Il nostro toolkit mantiene la flessibilità in modo che nuove funzionalità, librerie e software MD possano essere aggiunti introducendo classi responsabili in Python. Gli utenti che possono programmare in questo linguaggio dovrebbero essere in grado di modificare il codice di PaCS-Toolkit e implementare nuovi metodi secondo necessità, " afferma il Prof. Kitao.
Un altro vantaggio cruciale di PaCS-Toolkit risiede nella sua ottimizzazione e compatibilità con diversi ambienti informatici, che si tratti di una serie di supercomputer con un'interfaccia di passaggio di messaggi (MPI), server dotati di più schede grafiche (GPU) o personal computer come laptop.
"PaCS-Toolkit incorpora fortemente la parallelizzazione utilizzando MPI, GPU e il pacchetto multiprocessing di Python, consentendo l'ottimizzazione del tempo di calcolo a seconda delle risorse computazionali disponibili", spiega il prof. Kitao.
Per mostrare il potenziale del loro toolkit, i ricercatori hanno condotto simulazioni PaCS-MD su misura per tre diverse applicazioni. Questi includevano il ripiegamento della mini-proteina chignolina, il movimento dei domini proteici in un enzima SARS-CoV-2 e la dissociazione del ligando da un importante recettore dell'adenosina.
"Presi insieme, i nostri risultati indicano che PaCS-Toolkit può essere facilmente utilizzato per simulare un'ampia varietà di dinamiche per diversi tipi di sistemi molecolari", conclude il prof. Kitao.
Questo kit di strumenti potrebbe aiutare a sbloccare il vero potenziale delle simulazioni PaCS-MD in vari campi, consentendo ai ricercatori interessati di far luce su processi molecolari complessi e accelerare la scoperta di farmaci.
Ulteriori informazioni: Shinji Ikizawa et al, PaCS-Toolkit:Utilità software ottimizzate per simulazioni di dinamica molecolare di selezione in cascata parallela (PaCS-MD) e analisi successive, The Journal of Physical Chemistry B (2024). DOI:10.1021/acs.jpcb.4c01271
Informazioni sul giornale: Giornale di chimica fisica B
Fornito dal Tokyo Institute of Technology