Gli scienziati associati al progetto software internazionale MZmine, guidati dal Dr. Robin Schmid e dal Dr. Tomáš Pluskal dell'Istituto di chimica organica e biochimica dell'Accademia ceca delle scienze, hanno ideato un nuovo software che accelera e semplifica notevolmente l'identificazione di sostanze chimiche nei tessuti.
Consente ai ricercatori di riconoscere con sicurezza le sostanze chimiche e di visualizzarne la distribuzione negli organi. Rispetto ai flussi di lavoro consolidati, la nuova pipeline richiede meno lavoro in laboratorio per ottenere informazioni simili sulla chimica che avviene, ad esempio, all’interno dei tumori e delle lesioni infiammatorie. L'articolo che presenta questo software alla comunità scientifica globale è stato appena pubblicato sulla rivista Nature Communications .
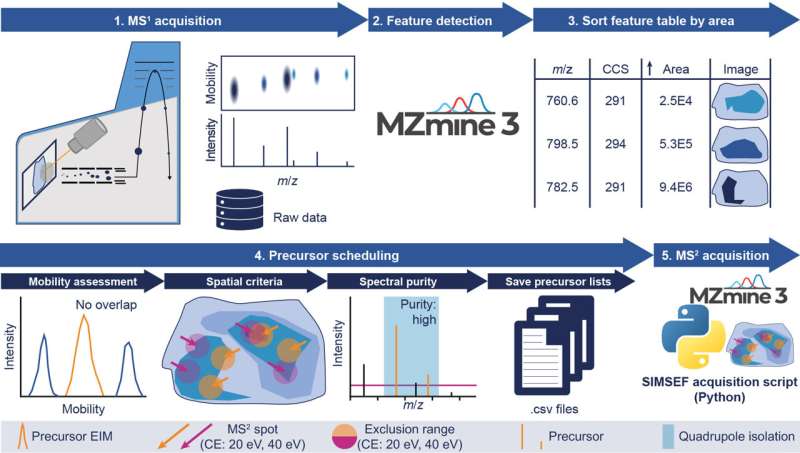
Presso l'IOCB di Praga, il Dr. Robin Schmid è principalmente coinvolto nello sviluppo del software SIMSEF (spatial ion mobilità-schedulata esaustiva frammentazione) insieme ai suoi colleghi del gruppo del Prof. Dr. Karst presso l'Università di Münster, Germania, dove ha lavorato in precedenza basato.
Il lavoro viene svolto in collaborazione con gli sviluppatori dello spettrometro di massa all'avanguardia timsTOF fleX di Bruker Daltonics, che consente la delucidazione di alto livello della composizione delle molecole misurando la mobilità degli ioni.
"Fino ad ora, gli scienziati potevano dire con uno spettrometro di massa che questa era la formula della molecola che stavano guardando, ma poi quando hanno guardato il database e hanno cercato di identificare questa sostanza, è stato molto difficile. Il motivo è che la biologia i campioni possono contenere numerosi lipidi distinti e combinazioni di molecole che spesso sono biologicamente molto diverse."
"Ora, grazie al nuovo algoritmo, è possibile guardare all'interno della molecola, scoprire da cosa è composta e persino confrontare le immagini tra loro", spiega Robin Schmid. Ciò può fornire informazioni importanti, ad esempio che una parte specifica del cervello contiene un tipo diverso di lipidi rispetto a un'altra parte di questo organo essenziale e che questo particolare lipide è assente in altri tessuti.
L'algoritmo SIMSEF è stato sviluppato in collaborazione con esperti medici di università tedesche e svizzere. Un aspetto fondamentale per i medici è che anche il rilevamento di biomarcatori clinici a fini diagnostici viene notevolmente accelerato. Imparare molto rapidamente, ad esempio, se si ha a che fare con un lipide o un metabolita che si trova solo nel tessuto canceroso, gioca un ruolo fondamentale nel decidere un ulteriore trattamento. Inutile dire che ciò ha grandi implicazioni per il suo esito.
Il nuovo algoritmo fa parte del pacchetto software open source MZmine, che dal 2005 aiuta gli esperti di tutto il mondo ad analizzare i dati della spettrometria di massa. Steffen Heuckeroth del team MZmine e dell'Istituto di chimica inorganica e analitica dell'Università di Münster è stato il responsabile sviluppatore principale dell'algoritmo SIMSEF ed è il primo autore dell'articolo ora pubblicato su Nature Communications .
E aggiunge:"L'articolo in questione non contiene alcuna scoperta specifica nel campo della biologia. Tuttavia, grazie al nostro nuovo metodo, molti scienziati possono migliorare e accelerare il loro lavoro, il che apre spazio a nuove scoperte."
"Penso sempre che sia fantastico quando permettiamo ad altre persone di migliorare le condizioni di lavoro per la loro ricerca e anche di comunicarla ad altri scienziati. Grazie al feedback della comunità scientifica che utilizza MZmine, il programma stesso spinge direttamente avanti la loro ricerca", aggiunge Robin Schmid del CIOB Praga.
Il direttore dell'IOCB di Praga, Prof. Jan Konvalinka, sottolinea:"Oggigiorno tutti parlano di machine learning e intelligenza artificiale. I nostri colleghi del team del Dr. Pluskal lo utilizzano per risolvere problemi altamente specifici nella ricerca così come nella pratica clinica."
La terza generazione di MZmine, riportata nella primavera del 2023 nella rivista Nature Biotechnology , può elaborare migliaia di campioni all'ora. Gli algoritmi recentemente sviluppati da questo team internazionale migliorano quindi ulteriormente le capacità esistenti di collegare diversi tipi di dati, inclusa la combinazione di dati provenienti da metodi analitici e di imaging, cosa che fino ad ora non era stata possibile con nessun software accademico o commerciale.
Ulteriori informazioni: Steffen Heuckeroth et al, Bioimaging MALDI-TIMS-MS2 dipendente dal set di dati sui tessuti, Nature Communications (2023). DOI:10.1038/s41467-023-43298-9
Informazioni sul giornale: Comunicazioni sulla natura , Biotecnologie naturali
Fornito dall'Istituto di chimica organica e biochimica dell'Accademia ceca delle scienze (IOCB Praga)