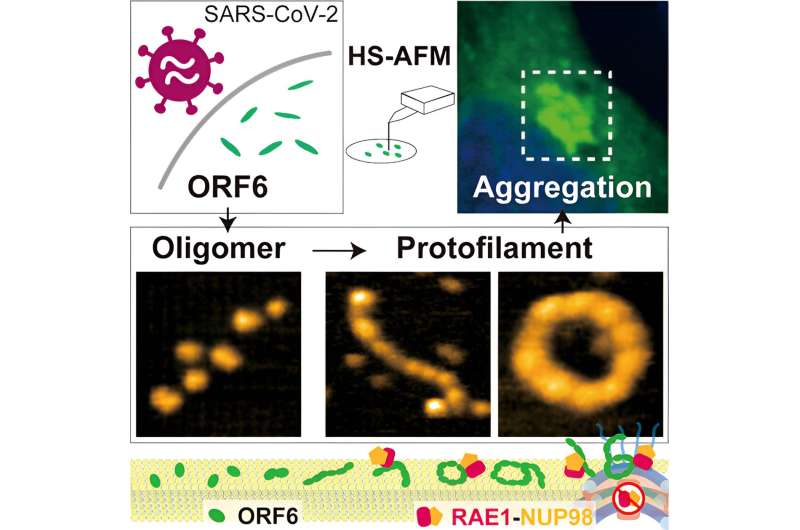
I ricercatori dell'Università di Kanazawa riferiscono nel Journal of Physical Chemistry Letters studi di microscopia a forza atomica ad alta velocità che fanno luce sul possibile ruolo della proteina open reading frame 6 (ORF6) nei sintomi del COVID-19.
Mentre molti paesi in tutto il mondo stanno vivendo una tregua dall'intensa diffusione delle infezioni da SARS-CoV-2 che ha portato a tragici livelli di malattia e a molteplici blocchi nazionali all'inizio del decennio, i casi di infezione persistono.
Una migliore comprensione dei meccanismi che sostengono il virus nel corpo potrebbe aiutare a trovare trattamenti più efficaci contro le malattie causate dalla malattia, oltre a prepararsi contro future epidemie di infezioni simili. Tenendo questo in mente, c'è stato molto interesse per le proteine accessorie prodotte dal virus per aiutarlo a prosperare nel corpo.
"Simile ad altri virus, SARS-CoV-2 esprime una serie di proteine accessorie per riprogrammare l'ambiente ospite per favorirne la replicazione e la sopravvivenza", spiegano Richard Wong dell'Università di Kanazawa e Noritaka Nishida dell'Università di Chiba e i loro colleghi in quest'ultimo rapporto. Tra queste proteine accessorie c'è l'ORF6.
Precedenti studi hanno suggerito che ORF6 interferisce con la funzione dell’interferone 1 (IFN-I), un particolare tipo di piccola proteina utilizzata nel sistema immunitario, il che potrebbe spiegare i casi di infezione asintomatica da SARS-CoV2. Esistono anche prove che l'ORF6 provoca la ritenzione di alcune proteine nel citoplasma interrompendo il trasporto dell'mRNA dalla cellula, il che potrebbe essere un mezzo per inibire la segnalazione dell'IFN-I. Tuttavia, il meccanismo alla base di questa ritenzione proteica e dell'interruzione del trasporto non era chiaro.
Per far luce su questi meccanismi, i ricercatori hanno prima esaminato quali indizi potrebbero fornire vari programmi software sulla struttura di ORF6. Questi indicavano la probabile presenza di diverse regioni intrinsecamente disordinate. Anche le misurazioni della risonanza magnetica nucleare hanno confermato la presenza di un segmento disordinato molto flessibile.
Sebbene l’algoritmo di apprendimento automatico AlphaFold2 si sia rivelato molto utile per determinare come le proteine si ripiegano, la presenza di queste regioni intrinsecamente disordinate ne limita l’uso per stabilire la struttura di ORF6, quindi i ricercatori hanno utilizzato la microscopia a forza atomica ad alta velocità (HS-AFM), che è in grado di identificare le strutture rilevando la topografia dei campioni nello stesso modo in cui la puntina di un giradischi attraversa i solchi del vinile.
Utilizzando HS-AFM i ricercatori hanno stabilito che ORF 6 è principalmente sotto forma di filamenti ellissoidali di oligomeri:stringhe di unità molecolari ripetitive ma più corte dei polimeri. La lunghezza e la circonferenza di questi filamenti erano massime a 37°C e minime a 4°C, quindi la presenza di febbre potrebbe essere utile per produrre filamenti più grandi. Anche i substrati costituiti da lipidi, ovvero composti grassi, incoraggiano la formazione di oligomeri più grandi.
Poiché HS-AFM cattura le immagini così rapidamente, è stato possibile cogliere non solo le strutture ma anche alcune dinamiche del comportamento dell'ORF6, tra cui il movimento circolare, l'assemblaggio e il ribaltamento delle proteine. Inoltre, ulteriori analisi computerizzate hanno anche rivelato che i filamenti erano inclini ad aggregarsi in amiloidi, come riscontrato in alcune malattie neurodegenerative, e che possono portare a complicazioni nei sintomi di COVID-19. Come sottolineano i ricercatori, questa aggregazione funziona "per sequestrare efficacemente un vasto numero di proteine ospiti, in particolare i fattori di trascrizione coinvolti nella segnalazione dell'IFN-I."
Poiché questi filamenti si rompono in presenza di alcuni alcoli, urea o sodio dodecil solfato, Wong, Nishida e i loro colleghi concludono che la proteina è in gran parte tenuta insieme da interazioni idrofobiche. "I potenziali candidati farmacologici che dissociano gli aggregati ORF6 interrompendo le interazioni idrofobiche dovrebbero essere considerati e testati nel prossimo futuro per valutare il loro valore terapeutico nella gestione e nel trattamento del COVID-19", affermano i ricercatori.
Ulteriori informazioni: Goro Nishide et al, Delucidazione nanoscopica dell'autoassemblaggio spontaneo della sindrome respiratoria acuta grave del coronavirus 2 (SARS-CoV-2) Open Reading Frame 6 (ORF6) Protein, The Journal of Physical Chemistry Letters (2023). DOI:10.1021/acs.jpclett.3c01440
Informazioni sul giornale: Giornale di lettere di chimica fisica
Fornito dall'Università di Kanazawa