La nuova tecnologia di sequenziamento delle proteine con sensibilità e produttività migliorate porterà una rivoluzione nella proteomica e nella diagnostica clinica.
In uno studio pubblicato su Nature Methods , un gruppo di ricerca guidato dal Prof. Wu Haichen dell'Istituto di Chimica dell'Accademia Cinese delle Scienze (CAS) e dal Prof. Liu Lei dell'Istituto di Fisica delle Alte Energie della CAS, insieme ai loro collaboratori, hanno sviluppato un nuovo metodo per il sequenziamento di peptidi basato sul rilevamento di nanopori assistito dall'interazione ospite-ospite.
La storia del sequenziamento delle proteine potrebbe essere fatta risalire alla determinazione della sequenza aminoacidica completa dell'insulina da parte di Sanger negli anni '50. Tuttavia, finora, esistono solo due metodi principali per il sequenziamento delle proteine, ovvero la spettrometria di massa e la degradazione di Edman.
Negli ultimi decenni, il rilevamento dei nanopori è emerso come l’ultima tecnica “dirompente” a singola molecola e ha ottenuto un grande successo nella nuova generazione di sviluppo del sequenziamento del DNA. Ciò ha ispirato gli scienziati a trapiantare la tecnologia nel sequenziamento proteico di singole molecole. Tuttavia, il sequenziamento delle proteine tramite nanopori deve affrontare enormi sfide, come la realizzazione di un trasporto unidirezionale di catene peptidiche caricate in modo eterogeneo attraverso un nanoporo e l'identificazione elettrica di 20 singoli amminoacidi o delle loro combinazioni.
In questo studio, i ricercatori hanno proposto una strategia di sequenziamento alternativa basata su una tecnica di rilevamento dei nanopori assistita dall'interazione ospite-ospite migliorata.
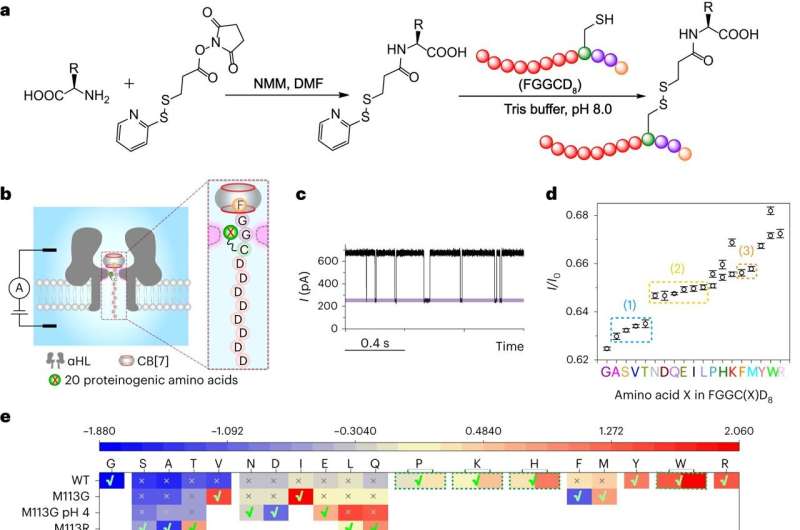
I peptidi modello sono stati prima digeriti con carbossipeptidasi per ottenere una miscela di amminoacidi. Il passaggio chiave successivo è stato quello di accoppiare gli amminoacidi rilasciati a una sonda peptidica FGGCD8⊂CB[7] tramite un linker covalente e quindi sottoporre il complesso a esperimenti di traslocazione attraverso l'α-emolisina di tipo selvatico o i suoi mutanti.
Infine, l'attuale blocco di ciascun peptide FGGC(X)D8⊂CB[7] è stato utilizzato per identificare l'amminoacido X e la loro abbondanza relativa è stata utilizzata per determinare l'ordine della scissione enzimatica, ovvero la sequenza del peptide.
Questo studio serve come dimostrazione di prova di un nuovo metodo in grado di determinare con precisione la sequenza aminoacidica di un peptide. Anche se persistono notevoli limitazioni, ciò segna un progresso significativo e svela una strada promettente per il futuro del sequenziamento delle proteine.
Ulteriori informazioni: Yun Zhang et al, Sequenziamento di peptidi basato sul rilevamento di nanopori assistito dall'interazione ospite-ospite, Metodi naturali (2023). DOI:10.1038/s41592-023-02095-4
Informazioni sul giornale: Metodi naturali
Fornito dall'Accademia cinese delle scienze