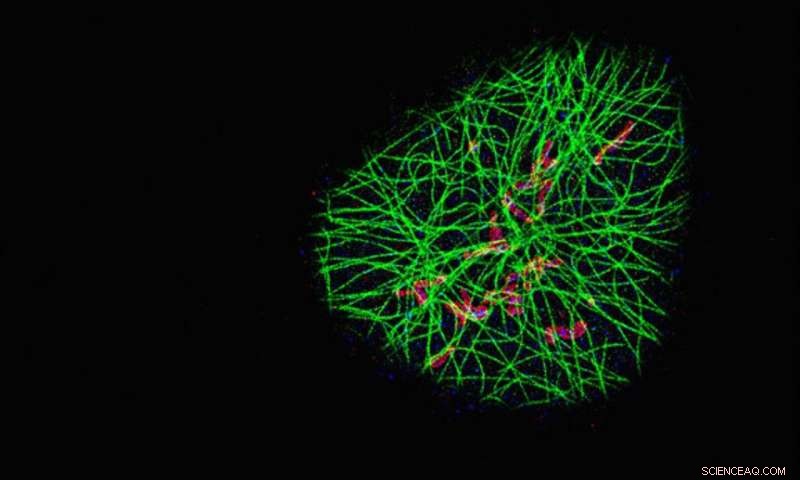
I ricercatori hanno utilizzato il loro metodo SDC-PAINT per visualizzare la rete di filamenti di microtubuli citoscheletrici (verde) e la loro vicinanza con due proteine aggiuntive chiamate TOM20 (rosso) e HSP60 (blu). Ogni immagine mostra le proteine in un piano diverso della cellula partendo dall'alto, e le immagini ingrandite in basso confrontano la risoluzione ottenuta con SDC-PAINT (a sinistra) con quella possibile con la microscopia confocale convenzionale (a destra). Credito:Florian Schueder, MPI/LMU
I biologi cellulari usano tradizionalmente coloranti fluorescenti per etichettare e visualizzare le cellule e le molecole al loro interno al microscopio. Con diversi metodi di microscopia a super risoluzione, possono anche illuminare singole molecole e le loro complesse interazioni tra loro. Però, l'hardware di microscopia che consente loro di farlo è altamente specializzato e costoso e quindi, relativamente raro nei laboratori di tutto il mondo, e il funzionamento di tali microscopi è scoraggiante, in quanto richiede abilità uniche.
Ralf Jungmann, dottorato di ricerca, un alunno del Wyss Institute for Biologically Inspired Engineering di Harvard e attualmente professore alla Ludwig Maximilian University (LMU) e al Max Planck Institute (MPI) di biochimica in Germania e membro della facoltà del Wyss Institute Core Peng Yin, dottorato di ricerca, hanno sviluppato DNA-PAINT, una potente tecnologia di imaging molecolare che coinvolge interazioni transitorie DNA-DNA per localizzare accuratamente i coloranti fluorescenti con una super risoluzione. Però, sebbene i ricercatori abbiano dimostrato il potenziale di DNA-PAINT visualizzando singole biomolecole, come proteine, in celle fisse a distanza ravvicinata fissa, la tecnologia non poteva ancora essere applicata per studiare le molecole in profondità all'interno delle cellule.
Ora, I team di Jungmann e Yin segnalano congiuntamente una soluzione per superare questa limitazione. Nel loro nuovo studio, hanno adattato la tecnologia DNA-PAINT ai microscopi diffusi tra i laboratori di biologia cellulare, chiamati microscopi confocali, e che vengono utilizzati dai ricercatori per visualizzare cellule intere e tessuti più spessi a una risoluzione inferiore. Il team dell'MPI/Wyss Institute dimostra che il metodo può visualizzare una varietà di molecole diverse, comprese le combinazioni di diverse proteine, RNA e DNA in tutta la profondità di intere cellule a super-risoluzione. Pubblicato in Comunicazioni sulla natura , l'approccio potrebbe aprire la porta a studi dettagliati sulla localizzazione di singole molecole in molte aree della ricerca cellulare.
L'approccio DNA-PAINT attacca un "filo di ancoraggio" di DNA alla molecola di interesse. Quindi un "filo di imager" di DNA marcato con colorante con una sequenza complementare si attacca transitoriamente all'ancora e produce un segnale fluorescente, che si verifica come un evento di lampeggio definito in singoli siti molecolari. Poiché "lampeggiante" è sintonizzabile con precisione, si possono distinguere molecole che distano solo nanometri l'una dall'altra, alla fine della risoluzione più alta della super-risoluzione.
"Il nostro nuovo approccio, SDC-PITTURA, integra le versatili capacità di super-risoluzione di DNA-PAINT con le caratteristiche di sezionamento ottico dei microscopi confocali. Abbiamo così creato i mezzi per esplorare l'intera profondità di una cellula, e per visualizzare le molecole al suo interno su scala nanometrica, " ha detto Jungmann. Il team ha mappato la presenza di diverse combinazioni di proteine all'interno di cellule intere e poi è andato oltre. " Diversificando i nostri approcci di etichettatura, abbiamo anche visualizzato diversi tipi di singole biomolecole nel nucleo contenente cromosomi, comprese le sequenze nel DNA, proteine legate al DNA o alla membrana che racchiude il nucleo, così come gli RNA nucleari, "aggiunge Yin, che è anche co-leader della Molecular Robotics Initiative del Wyss Institute, e professore di biologia dei sistemi alla Harvard Medical School. .
In linea di principio, i microscopi confocali utilizzano i cosiddetti fori stenopeici per eliminare la fluorescenza fuori fuoco indesiderata dai piani dell'immagine sopra e sotto il piano focale. Scansionando il campione, aereo dopo aereo, i ricercatori possono raccogliere i segnali di fluorescenza emessi dai coloranti legati alla molecola su tutta la profondità. Nello specifico, il team dell'MPI/Wyss Institute ha sviluppato la tecnica per microscopi "Spinning Disk Confocal" (SDC) che rilevano i segnali di fluorescenza da un intero piano contemporaneamente rilevandoli attraverso un disco rotante con più fori di spillo. Inoltre, "per ottenere una super risoluzione 3D, abbiamo posizionato una lente aggiuntiva nel percorso di rilevamento, che ci consente di archiviare la risoluzione limitata dalla sub-diffrazione nella terza dimensione", ha affermato il primo autore Florian Schueder, uno studente laureato che lavora con Jungmann che ha anche lavorato con il team del Wyss Institute di Yin come parte della sua tesi di laurea.
"Questa aggiunta può essere facilmente personalizzata dai produttori di microscopi SDC; quindi fondamentalmente implementiamo la microscopia a super risoluzione senza complesse modifiche hardware ai microscopi che sono generalmente disponibili per i biologi cellulari di tutte le sedi della ricerca biomedica. L'approccio ha quindi il potenziale per democratizzare il super -imaging ad alta risoluzione su cellule e tessuti interi, " disse Jungmann.
"Con questo importante passo avanti, microscopia a super risoluzione e DNA-PAINT potrebbero diventare più accessibili ai ricercatori biomedici, accelerando le nostre intuizioni sulla funzione delle singole molecole e sui processi che controllano all'interno delle cellule, ", ha affermato il direttore fondatore del Wyss Institute, Donald Ingber, M.D., dottorato di ricerca, che è anche Judah Folkman Professor of Vascular Biology presso HMS e Vascular Biology Program presso il Boston Children's Hospital, nonché Professore di Bioingegneria presso SEAS.