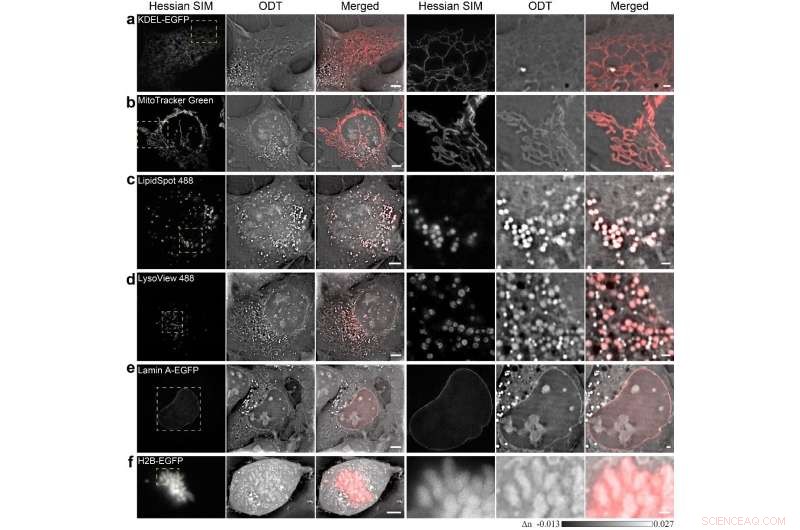
Le regioni racchiuse dai riquadri gialli tratteggiati a sinistra vengono ingrandite a destra. Barre della scala, 5 micron (sinistra) e 1 micron (destra). Credito:di Dashan Dong, Xiaoshuai Huang, Liuju Li, Heng Mao, Yanquan Mo, Guangyi Zhang, Zhe Zhang, Jiayu Shen, Wei Liu, Zeming Wu, Guanghui Liu, Yanmei Liu, Hong Yang, Qihuang Gong, Kebin Shi, Liangyi Chen
L'emergere della microscopia a fluorescenza a superrisoluzione (SR) ha ringiovanito la ricerca di nuove sottostrutture cellulari e intermedi dinamici. Però, limitato dall'ampio spettro di emissione dei fluorofori e dall'eccessiva fototossicità, La microscopia a fluorescenza SR può essere utilizzata solo per evidenziare contemporaneamente una manciata di biomolecole e non è in grado di fornire una mappa olistica dell'ambiente e del paesaggio cellulare.
In un nuovo articolo pubblicato su Scienza e applicazioni della luce , scienziati dello State Key Laboratory for Mesoscopic Physics and Frontiers Science Center for Nano-optoelectronics, Università di Pechino, Cina, Laboratorio statale chiave di biologia delle membrane, Laboratorio chiave di Pechino di medicina molecolare cardiometabolica, Istituto di Medicina Molecolare, Università di Pechino, La Cina e i suoi collaboratori hanno sviluppato la tomografia computazionale a diffrazione assistita da fluorescenza SR (SR-FACT), che combina la tomografia a diffrazione ottica tridimensionale (ODT) senza etichetta con la microscopia a illuminazione strutturata dell'Assia a fluorescenza bidimensionale. Il modulo ODT è in grado di risolvere i mitocondri, goccioline lipidiche, la membrana nucleare, cromosomi, reticolo endoplasmatico tubulare e lisosomi. Utilizzando l'imaging di cellule vive correlate a doppia modalità per un periodo di tempo prolungato, hanno osservato nuove strutture subcellulari denominate corpi di vacuoli oscuri (DB), la maggior parte dei quali proviene da regioni perinucleari densamente popolate, e interagiscono intensamente con organelli come i mitocondri e la membrana nucleare prima di collassare definitivamente nella membrana plasmatica. Questo lavoro dimostra le capacità uniche di SR-FACT, il che suggerisce la sua ampia applicabilità nella biologia cellulare in generale.
SR-FACT visualizza sia il panorama cellulare che l'identità molecolare delle cellule vive. È stato sviluppato un nuovo algoritmo denominato algoritmo di ricerca iterativa vettoriale (VISA) per ridurre al minimo gli errori di ricostruzione dell'imaging 3D in uno schema di scansione tomografica a frequenza di kHz ad alta velocità. Di conseguenza, SR-FACT può utilizzare contemporaneamente una velocità di imaging massima per catturare le dinamiche nelle cellule vive e mantenere un flusso di fotoni sufficiente per la massima sensibilità. Nel sistema SR-FACT, il modulo ODT ha raggiunto una risoluzione laterale di ~200 nm a una velocità di imaging volumetrico di 0,8 Hz (40×40×20 m 3 ). 2-D-SIM dell'Assia, che consente l'imaging SR a una frazione della dose di fotoni utilizzata dalle SIM convenzionali, è stato utilizzato per guidare l'interpretazione delle strutture osservate dal modulo ODT. Eseguendo l'imaging correlato a doppia modalità nelle cellule COS-7, hanno risolto sei organelli noti senza etichettatura:il reticolo endoplasmatico tubulare (ER), mitocondri, endosomi/lisosomi tardivi (LEs/LYs), LD, la membrana nucleare e i cromosomi. Tutti questi dati evidenziano il vantaggio unico di SR-FACT nello studio dell'interattoma degli organelli. Inoltre, hanno anche osservato strutture vacuolate con pH neutro che contenevano prevalentemente liquido nel lume. L'imaging SR di cellule vive trascorse un'ora in combinazione con l'analisi quantitativa rivela le rotte del traffico non convenzionale e i ruoli indispensabili dei vacuoli nell'organizzazione dell'interattoma dell'organello, tutto ciò suggerisce che rappresentano organelli precedentemente non apprezzati.