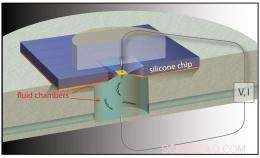
Un team di ricercatori guidati dall'ingegnere biomedico della Boston University Amit Meller sta usando campi elettrici per disegnare in modo efficiente lunghi filamenti di DNA attraverso sensori a nanopori, riducendo drasticamente il numero di copie di DNA necessarie per un'analisi ad alto rendimento. Figura diritto d'autore, Nanotecnologia della natura, 2009
(PhysOrg.com) -- Gli ingegneri biomedici della Boston University hanno ideato un metodo per rendere il futuro sequenziamento del genoma più veloce ed economico riducendo drasticamente la quantità di DNA richiesta, eliminando così il costoso, fase di amplificazione del DNA che richiede tempo e che è soggetta a errori.
In uno studio pubblicato nell'edizione online del 20 dicembre di Nature Nanotechnology, un team guidato dal professore associato di ingegneria biomedica dell'Università di Boston, Amit Meller, descrive in dettaglio il lavoro pionieristico nel rilevamento delle molecole di DNA mentre passano attraverso i nanopori di silicio. La tecnica utilizza campi elettrici per alimentare lunghi filamenti di DNA attraverso pori larghi quattro nanometri, proprio come infilare un ago. Il metodo utilizza misurazioni sensibili della corrente elettrica per rilevare singole molecole di DNA mentre passano attraverso i nanopori.
"L'attuale studio mostra che possiamo rilevare una quantità molto più piccola di campione di DNA rispetto a quanto riportato in precedenza, " ha detto Meller. "Quando le persone iniziano a implementare il sequenziamento del genoma o il profilo del genoma utilizzando i nanopori, potrebbero utilizzare il nostro approccio di cattura dei nanopori per ridurre notevolmente il numero di copie utilizzate in tali misurazioni".
Attualmente, il sequenziamento del genoma utilizza l'amplificazione del DNA per creare miliardi di copie molecolari al fine di produrre un campione sufficientemente grande da essere analizzato. Oltre ai tempi e ai costi che l'amplificazione del DNA comporta, alcune molecole - come fotocopie di fotocopie - risultano meno che perfette. Meller e i suoi colleghi della BU, La New York University e la Bar-Ilan University in Israele hanno sfruttato i campi elettrici che circondano le bocche dei nanopori per attirare lunghi, filamenti di DNA caricati negativamente e farli scorrere attraverso il nanoporo dove può essere rilevata la sequenza del DNA. Poiché il DNA viene attratto dai nanopori a distanza, sono necessarie molte meno copie della molecola.
Prima di creare questo nuovo metodo, il team ha dovuto sviluppare una comprensione dell'elettrofisica su scala nanometrica, dove le regole che governano il mondo più ampio non si applicano necessariamente. Hanno fatto una scoperta controintuitiva:più lungo è il filamento di DNA, più rapidamente trovava l'apertura dei pori.
"È davvero sorprendente, "Meller ha detto. "Ci si aspetterebbe che se si dispone di un più lungo 'spaghetti, ' allora trovare la fine sarebbe molto più difficile. Allo stesso tempo, questa scoperta significa che il sistema dei nanopori è ottimizzato per il rilevamento di lunghi filamenti di DNA - decine di migliaia di paia di basi, o anche di più. Ciò potrebbe accelerare notevolmente il futuro sequenziamento genomico consentendo l'analisi di un lungo filamento di DNA in un colpo solo, piuttosto che dover assemblare i risultati da molti brevi frammenti.
"Le tecnologie di amplificazione del DNA limitano la lunghezza della molecola del DNA a meno di mille paia di basi, " Ha aggiunto Meller. "Poiché il nostro metodo evita l'amplificazione, non solo riduce il costo, tempo e tasso di errore delle tecniche di replicazione del DNA, ma consente anche l'analisi di filamenti di DNA molto lunghi, molto più lungo dei limiti attuali."
Con questa conoscenza in mano, Meller e il suo team hanno deciso di ottimizzare l'effetto. Hanno usato gradienti salini per alterare il campo elettrico intorno ai pori, che ha aumentato la velocità con cui le molecole di DNA sono state catturate e accorciato il tempo di ritardo tra le molecole, riducendo così la quantità di DNA necessaria per misurazioni accurate. Invece di fluttuare finché non si sono imbattuti in un nanoporo, I filamenti di DNA sono stati incanalati nelle aperture.
Aumentando i tassi di cattura di alcuni ordini di grandezza, e riducendo il volume della camera del campione i ricercatori hanno ridotto il numero di molecole di DNA richieste di un fattore 10, 000 - da circa 1 miliardo di molecole campione a 100, 000.