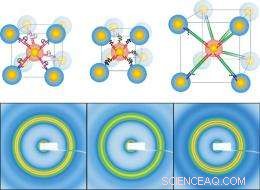
Queste illustrazioni mostrano come un cristallo 3-D fatto da nanoparticelle cambia tra due stati distinti tramite una struttura intermedia (riga superiore, al centro) quando vengono utilizzate catene di DNA a doppio filamento ad anello (a sinistra) rispetto a quelle non ad anello (a destra) per collegare le particelle. Gli scienziati sono stati in grado di misurare la distanza tra le particelle in ciascuna struttura registrando i modelli di diffusione dei raggi X (riga in basso). Il passaggio dal DNA ad anello a quello non ad anello ha aumentato la distanza interparticellare di circa 6 nanometri.
(PhysOrg.com) -- Gli scienziati del Brookhaven National Laboratory del Dipartimento dell'Energia degli Stati Uniti hanno trovato un nuovo modo per utilizzare una forma sintetica di DNA per controllare l'assemblaggio delle nanoparticelle, questa volta risultando commutabile, strutture tridimensionali e a piccoli cluster che potrebbero essere utili, Per esempio, come biosensori, nelle celle solari, e come nuovi materiali per l'archiviazione dei dati. Il lavoro è descritto in Nanotecnologia della natura, pubblicato online il 20 dicembre 2009.
La squadra di Brookhaven, guidato dal fisico Oleg Gang, ha perfezionato le tecniche per utilizzare filamenti di DNA artificiale come un tipo altamente specifico di velcro o colla per collegare le nanoparticelle. Tale autoassemblaggio basato sul DNA è promettente per la progettazione razionale di una gamma di nuovi materiali per applicazioni nella separazione molecolare, elettronica, conversione di energia, e altri campi. Ma nessuna di queste strutture ha avuto la capacità di cambiare in modo programmabile in risposta a stimoli molecolari, fino ad ora.
"Ora stiamo usando un tipo speciale di dispositivo di collegamento del DNA - una sorta di "colla intelligente" - che influenza il modo in cui le particelle si connettono per creare strutture che sono commutabili tra diverse configurazioni, "dice Gang. Questo affidabile, la commutazione reversibile potrebbe essere utilizzata per regolare le proprietà funzionali, ad esempio la fluorescenza di un materiale e le proprietà di trasferimento di energia - per creare nuovi materiali che rispondono alle mutevoli condizioni, o per modificare le loro funzioni su richiesta.
Tale reattività ai cambiamenti delle condizioni ambientali e la capacità di adottare nuove forme sono i tratti distintivi dei sistemi viventi. In quel modo, questi nuovi nanomateriali imitano più da vicino i sistemi biologici rispetto a qualsiasi precedente nanostruttura. Benché lontana da ogni forma di vera “vita artificiale, ” questi materiali potrebbero portare alla progettazione di macchine su scala nanometrica che, a un livello molto semplice, imitare processi cellulari come convertire la luce solare in energia utile, o percependo la presenza di altre molecole. I materiali reattivi avrebbero anche vantaggi nel campo dell'ottica o per produrre materiali porosi regolati per separazioni molecolari, Gang dice.
Gli scienziati hanno raggiunto l'obiettivo della reattività creando strutture in cui la distanza tra le nanoparticelle poteva essere controllata attentamente con precisione nanometrica.
“Molte caratteristiche fisiche dei nanomateriali, quali proprietà ottiche e magnetiche, sono fortemente dipendenti dalla distanza tra le nanoparticelle, "Gang spiega.
Nei loro studi precedenti, gli scienziati hanno utilizzato singoli filamenti di DNA attaccati a singole nanoparticelle come molecole di collegamento. Quando le estremità libere di questi filamenti di DNA avevano un codice genetico complementare, si legherebbero per attaccare le particelle. Vincolare le interazioni ancorando alcune delle particelle su una superficie ha permesso agli scienziati di formare in modo affidabile una varietà di strutture da cluster di due particelle (chiamati dimeri) a cristalli di nanoparticelle 3-D più complessi.
Nel nuovo lavoro, gli scienziati hanno aggiunto strutture di DNA a doppio filamento più complicate. A differenza dei singoli fili, che si arrotolano in modi incontrollabili, queste strutture a doppio filamento sono più rigide e quindi vincolano le distanze tra le particelle.
Inoltre, alcuni dei filamenti che compongono le molecole di DNA a doppio filamento hanno strutture complicate come anse, che avvicinano le particelle legate rispetto a quando entrambi i filamenti sono esattamente paralleli. Variando il tipo di dispositivo del DNA, tra fili ad anello e fili non snodati, e misurare le distanze tra le particelle utilizzando tecniche di precisione presso la National Synchrotron Light Source (NSLS) di Brookhaven e presso il Center for Functional Nanomaterials (CFN), gli scienziati hanno dimostrato di poter controllare efficacemente la distanza tra le particelle e far passare il sistema da uno stato all'altro a piacimento.
L'approccio ha portato a due configurazioni, sistemi commutabili sia in dimeri che in nanocristalli, con una variazione di distanza di circa 6 nanometri, circa il 25% della distanza interparticellare. Confrontando la cinetica nei due sistemi, hanno scoperto che il passaggio da uno stato all'altro è più veloce nel più semplice, sistema a due particelle. I dimeri mantengono anche la loro capacità di tornare al loro stato iniziale in modo più preciso rispetto ai cristalli 3-D, suggerendo che l'affollamento molecolare può essere un problema da approfondire nei materiali 3-D.
“La nostra speranza è che la capacità di indurre la riorganizzazione post-assemblaggio di queste strutture aggiungendo DNA o altre molecole come stimoli esterni, e la nostra capacità di osservare questi cambiamenti con una risoluzione nanometrica, ci aiuterà a comprendere questi processi e a trovare il modo di applicarli in nuovi tipi di nanomacchine in cui la funzionalità del sistema è determinata dalle nanoparticelle e dalla loro relativa organizzazione, "dice Gang.
Gli studi futuri si avvarranno di precise capacità di imaging, come strumenti avanzati di microscopia elettronica presso il CFN e tecniche a raggi X ad alta risoluzione che saranno disponibili presso la nuova fonte di luce di Brookhaven, NSLS-II, ora in costruzione.