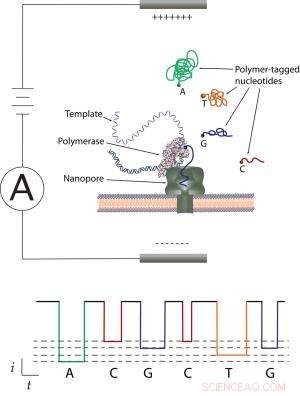
Schema di una membrana artificiale, attraverso il quale una tensione forza un fluido ionizzato attraverso il nanoporo. I nucleotidi su un filamento di DNA vengono prima etichettati con polimeri di dimensioni diverse, e poi il filo viene passato vicino all'apertura del nanoporo, dove una polimerasi scinde i polimeri e li fa passare uno ad uno attraverso il nanoporo. Mentre passano, il poro produce una firma unica di blocco della corrente ionica a causa della struttura chimica distinta del tag, determinando così la sequenza del DNA. Credito:NIST
(Phys.org)—Rapido, un accurato sequenziamento genetico potrebbe presto essere alla portata di ogni studio medico se le recenti ricerche del National Institute of Standards and Technology (NIST) e della School of Engineering and Applied Science della Columbia University potranno essere commercializzate in modo efficace. Il team ha dimostrato un potenziale a basso costo, modo affidabile per ottenere le sequenze complete di DNA di qualsiasi individuo utilizzando una sorta di lettore di nastri magnetici molecolari, potenzialmente consentendo un facile rilevamento di marcatori di malattia nel DNA di un paziente.
Mentre il sequenziamento del genoma di una specie animale per la prima volta è così comune che difficilmente fa più notizia, è meno noto che il sequenziamento del DNA di ogni singolo individuo è un affare costoso, che costa molte migliaia di dollari utilizzando la tecnologia odierna. Il genoma di un individuo porta marcatori che possono fornire un avvertimento anticipato del rischio di malattia, ma hai bisogno di un digiuno, modo affidabile ed economico di sequenziare i geni di ogni paziente per trarne il massimo vantaggio. Altrettanto importante è la necessità di sequenziare continuamente il DNA di un individuo nel corso della sua vita, perché il codice genetico può essere modificato da molti fattori.
Il nuovo metodo determina le sequenze di DNA attaccando distinti "tag" molecolari a ciascuno dei quattro elementi costitutivi chimici, o "basi, " che comprendono le informazioni genetiche in un filamento di DNA, abbreviato come A, G, C e T. Ciascuno di questi tag polimerici può quindi essere tagliato dal filo e passato, uno per uno, attraverso un foro nanometrico in una membrana. Un flusso costante di fluido e ioni scorre attraverso questo "nanoporo, " che è abbastanza grande da contenere un solo tag alla volta. Poiché i tag polimerici sono di dimensioni diverse, la variazione della corrente elettrica causata dall'alterazione del flusso del fluido mostra quale delle quattro basi si trova in ciascun punto del filamento di DNA.
I nanopori e la loro interazione con le molecole polimeriche sono stati a lungo un obiettivo di ricerca dello scienziato del NIST John Kasianowicz. Il suo gruppo ha collaborato con un team guidato da Jingyue Ju, direttore del Centro per la tecnologia del genoma e l'ingegneria biomolecolare della Columbia, che ha avuto l'idea di etichettare i blocchi costitutivi del DNA per il sequenziamento di singole molecole mediante rilevamento di nanopori. La capacità di discriminare tra i tag polimerici è stata dimostrata da Kasianowicz, il suo collega del NIST Joseph Robertson, e altri. La Columbia University ha richiesto brevetti per la commercializzazione della tecnologia.
Kasianowicz stima che la tecnica potrebbe identificare un elemento costitutivo del DNA con un'accuratezza estremamente elevata con un tasso di errore inferiore a uno su 500 milioni, e l'attrezzatura necessaria sarebbe alla portata di qualsiasi fornitore di servizi medici. "Il cuore del sequencer sarebbe un amplificatore operazionale che costerebbe molto meno di $ 1, 000 per un acquisto una tantum, " lui dice, "e il costo dei materiali e del software dovrebbe essere banale".
Kasianowicz aggiunge che un'azienda privata potrebbe creare una vasta gamma di nanopori in grado di analizzare il genoma di un singolo individuo tagliato in molti brevi filamenti di DNA, ognuno dei quali potrebbe essere sequenziato rapidamente. Un tale array potrebbe potenzialmente fornire il sequenziamento a basso costo necessario per l'uso medico di routine.