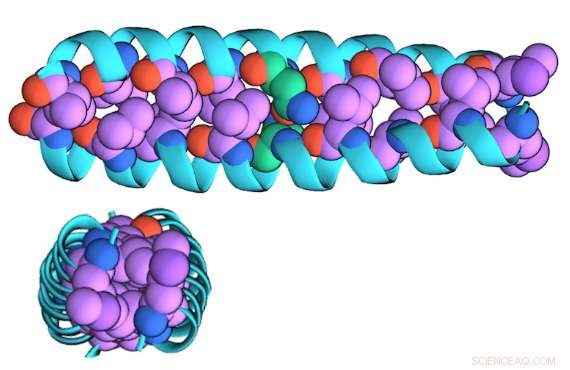
Un'illustrazione della forma dimero "zippata" di GCN4-p1 vista di lato (in alto) e sopra (in basso)
I ricercatori dell'Università della Pennsylvania hanno fatto passi da gigante verso un nuovo metodo di sequenziamento genico:un filamento di basi del DNA viene letto mentre viene infilato attraverso un foro nanoscopico.
In un nuovo studio, hanno dimostrato che questa tecnica può essere applicata anche alle proteine come modo per saperne di più sulla loro struttura.
I metodi esistenti per questo tipo di analisi richiedono molta manodopera, tipicamente comportando la raccolta di grandi quantità della proteina. Spesso richiedono anche la modifica della proteina, limitare l'utilità di questi metodi per comprendere il comportamento della proteina nel suo stato naturale.
La tecnica di traslocazione dei ricercatori Penn consente lo studio delle singole proteine senza modificarle. I campioni prelevati da un singolo individuo potrebbero essere analizzati in questo modo, apertura di applicazioni per la diagnostica e la ricerca delle malattie.
Lo studio è stato condotto da Marija Drndić, un professore nel Dipartimento di Fisica e Astronomia della School of Arts &Sciences; David Niedzwiecki, un ricercatore post-dottorato nel suo laboratorio; e Jeffery G. Saven, un professore nel Dipartimento di Chimica della Penn Arts &Sciences.
È stato pubblicato sulla rivista ACS Nano .
La tecnica del team Penn deriva dal lavoro di Drndić sul sequenziamento dei geni dei nanopori, che mira a distinguere le basi in un filamento di DNA per la diversa percentuale dell'apertura che ciascuna blocca mentre passano attraverso un poro nanoscopico. Sagome diverse consentono il passaggio di quantità diverse di un liquido ionico. Il cambiamento nel flusso di ioni è misurato dall'elettronica che circonda il poro; i picchi e le valli di quel segnale possono essere correlati a ciascuna base.
Mentre i ricercatori lavorano per aumentare la precisione di queste letture a livelli utili, Drndić e i suoi colleghi hanno sperimentato l'applicazione della tecnica ad altre molecole biologiche e strutture su nanoscala.
Collaborando con il gruppo di Saven, hanno deciso di testare i loro pori su molecole biologiche ancora più difficili.
"Ci sono molte proteine che sono molto più piccole e più difficili da manipolare di un filamento di DNA che vorremmo studiare, "Saven ha detto. "Siamo interessati a conoscere la struttura di una determinata proteina, come se esiste come monomero, o combinato con un'altra copia in un dimero, o un aggregato di più copie noto come oligomero."
Anche il rilevamento è spesso un limite.
"Non ci sono modi per amplificare peptidi e proteine come ci sono per il DNA, " ha detto Drndić. "Se vuoi studiare le proteine da una fonte particolare, sei bloccato con campioni molto piccoli. Con questo metodo, però, puoi semplicemente raccogliere la quantità di dati di cui hai bisogno e il numero di proteine che vuoi far passare attraverso il poro e poi studiarle una alla volta poiché esistono naturalmente nel corpo".
Utilizzando i nanopori di nitruro di silicio del gruppo Drndić, che può essere forato a diametri personalizzati, il team di ricerca ha deciso di testare la propria tecnica su GCN4-p1, una proteina selezionata perché contiene un motivo strutturale comune che si trova nei fattori di trascrizione e nei recettori intracellulari.
"La versione dimer è 'zippata' insieme, " Niedzwiecki ha detto, "È una 'bobina arrotolata' di eliche intercalate che è approssimativamente cilindrica. La versione monomerica è decompressa e probabilmente non è elicoidale; probabilmente è più simile a una corda".
I ricercatori hanno messo diversi rapporti di versioni zippate e non zippate della proteina in un fluido ionico e le hanno fatte passare attraverso i pori. Pur non essendo in grado di distinguere le singole proteine, i ricercatori potrebbero eseguire questa analisi sulle popolazioni della molecola.
"La forma dimero e monomero della proteina blocca un diverso numero di ioni, quindi vediamo un diverso calo di corrente quando passano attraverso il poro, " ha detto Niedzwiecki. "Ma otteniamo una gamma di valori per entrambi, poiché non tutti gli eventi di traslocazione molecolare sono uguali".
Determinare se un campione specifico di questi tipi di proteine si sta aggregando o meno potrebbe essere utilizzato per comprendere meglio la progressione della malattia.
"Molti ricercatori, "Salvato ha detto, "hanno osservato questi lunghi grovigli di peptidi e proteine aggregati in malattie come l'Alzheimer e il Parkinson, ma c'è un numero crescente di prove che suggeriscono che questi grovigli si stanno verificando dopo il fatto, che ciò che sta realmente causando il problema sono gli assemblaggi proteici più piccoli. Capire cosa sono queste assemblee e quanto sono grandi è attualmente davvero difficile da fare, quindi questo potrebbe essere un modo per risolvere il problema."