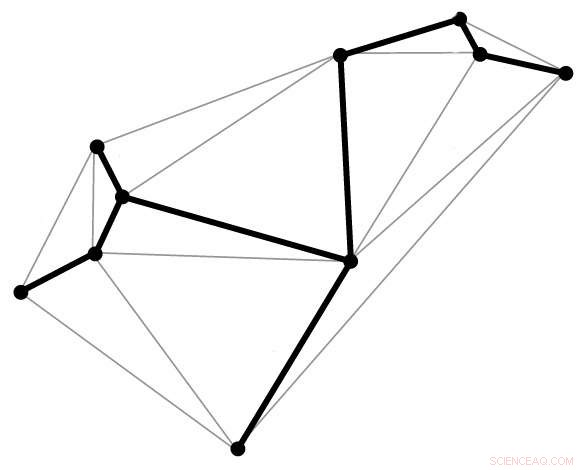
La linea in grassetto, conosciuto come un albero ricoprente, segue la forma geometrica desiderata, toccando ogni vertice solo una volta. Un algoritmo spanning tree viene utilizzato nel nuovo metodo origami del DNA per mappare il percorso di instradamento corretto per il filamento di DNA. Credito:dominio pubblico
Tra i preziosi fondi della Wellcome Library di Londra c'è un rozzo schizzo a matita realizzato nel 1953 da Francis Crick. Il disegno è uno dei primi a mostrare la struttura a doppia elica del DNA:il progetto della Natura per la progettazione delle lumache di mare, esseri umani, e ogni altra forma vivente sulla terra.
Pochi avrebbero potuto prevedere, tuttavia, le semplici proprietà di autoassemblaggio del DNA, e la sua versatile capacità di trasporto di informazioni, potrebbe essere utilizzato per molti usi mai immaginati da Watson e Crick, (o anzi, dalla Natura stessa).
In una nuova ricerca che appare nell'edizione online anticipata della rivista Scienza , Marco Bath, insieme ai colleghi Hao Yan dell'ASU e Wah Chiu del Baylor College of Medicine descrivono un nuovo metodo per progettare forme geometriche costruite dal DNA. Presentano una nuova variante di una tecnica nota come DNA origami, in cui le proprietà di accoppiamento delle basi del DNA vengono sfruttate per la costruzione di minuscole strutture in 2 e 3 dimensioni.
"Una sfida importante nel campo della nanotecnologia del DNA è progettare qualsiasi struttura desiderabile in modo top-down, senza molto input umano riguardo ai dettagli dei percorsi di piegatura del filamento di DNA, " dice Yan.
I suoi collaboratori al MIT, guidato da Mark Bathe, ha sviluppato un algoritmo informatico per progettare nanostrutture di DNA inserendo solo una forma bersaglio. Hanno progettato una piattaforma software in grado di calcolare e produrre i filamenti di DNA necessari per formare architetture di design. La formazione di queste strutture è stata poi sistematicamente caratterizzata e confermata sperimentalmente presso i tre istituti. "Questo mostra davvero la scienza collaborativa interdisciplinare in tutto il paese, "Yan dice
Yan dirige il Biodesign Center for Molecular Design and Biomimetics presso l'Arizona State University.
Altri mondi
Il team progetta strutture utili su una scala sorprendentemente ridotta. (Un nanometro è un miliardesimo di metro o circa le dimensioni di una molecola di zucchero.) Tecniche di imaging specializzate, inclusa la microscopia a forza atomica e crioelettronica vengono utilizzati per visualizzare le forme risultanti.
La tecnica semplificata descritta promette di estendere in modo significativo l'uso dell'origami del DNA oltre la comunità specialistica e di ampliare la gamma di possibili applicazioni nella scienza biomolecolare e nelle nanotecnologie. Questi includono l'uso di nanoparticelle per la somministrazione di farmaci e il targeting cellulare, costruzione di robot su scala nanometrica in grado di svolgere diverse attività nel campo della medicina e dell'industria e la progettazione di dispositivi ottici su misura.
Una delle innovazioni più entusiasmanti all'orizzonte riguarda l'uso del DNA come supporto di memorizzazione, che vanta tempi di conservazione di milioni di anni. (Un singolo grammo di DNA può contenere circa 700 terabyte di informazioni, una quantità equivalente a 14, 000 dischi Blu-ray da 50 gigabyte. Ulteriore, tale memoria di acido nucleico potrebbe potenzialmente essere utilizzata con una frazione dell'energia richiesta per altre opzioni di memorizzazione delle informazioni.)
Muta forma
Il nuovo metodo di progettazione, che può produrre praticamente qualsiasi forma poliedrica, si basa su una strategia top-down, che inizia con uno schema della forma desiderata e lavora a ritroso per fasi per definire la sequenza di DNA richiesta che si piegherà correttamente per formare il prodotto finito.
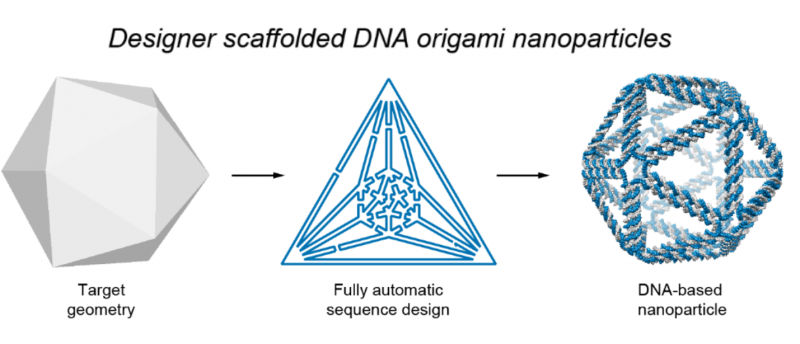
L'illustrazione mostra il processo di base utilizzato per progettare nanostrutture di origami di DNA. Primo, viene realizzato un wireframe del progetto di destinazione previsto. Il software quindi traduce questo in un piano per l'instradamento dell'impalcatura del DNA e dei filamenti di graffette, che si assemblano per formare la forma desiderata. Credito:Istituto di Biodesign
Il processo autonomo viene eseguito utilizzando un programma software progettato dagli autori. Conosciuto come DAEDALUS (per DNA Origami Sequence Design Algorithm for User-Defined Structures), il programma esegue la progettazione inversa di nanoforme di origami di DNA arbitrarie, basato su una mesh wireframe di input, (una rappresentazione visiva del chiuso, superficie geometrica tridimensionale).
Il programma non è solo user-friendly, ma altamente versatile, producendo forme non limitate alla topologia sferica, (cioè., Chiuso, strutture a due lati senza confini o buchi). Una volta che la forma di destinazione è stata descritta come una rete di nodi e archi, I filamenti dell'impalcatura del DNA di lunghezza e sequenza personalizzate vengono generati utilizzando una tecnologia nota come reazione a catena della polimerasi asimmetrica.
Il nuovo studio descrive la fabbricazione di una varietà di oggetti di DNA geometrici, tra cui 35 forme poliedriche (platonico, Archimede, solidi Johnson e Catalan) 6 strutture asimmetriche, e quattro poliedri con topologia non sferica, utilizzando principi di progettazione inversa. Il metodo può produrre nanoforme con alta fedeltà e stabilità senza il normale processo laborioso di progettazione manuale di coppie di basi per formare la struttura di destinazione prevista.
Entrando nell'ovile
Il DNA-origami porta l'antico metodo giapponese di piegare la carta su scala molecolare. Le basi sono semplici:prendi un pezzo di DNA a singolo filamento e guidalo nella forma desiderata, fissaggio della struttura insieme utilizzando i cosiddetti trefoli più corti, che si legano in punti strategici lungo la lunghezza maggiore del DNA. Il metodo si basa sul fatto che le quattro lettere nucleotidiche del DNA:A, T, C, &G si uniscono in modo coerente; Come sempre in coppia con Ts e Cs con Gs.
La molecola di DNA nella sua caratteristica forma a doppio filamento è abbastanza rigida, rispetto al DNA a filamento singolo, che è flessibile. Per questa ragione, il DNA a singolo filamento costituisce un materiale ideale per impalcature simili a pizzi. Ulteriore, le sue proprietà di accoppiamento sono prevedibili e coerenti, (a differenza dell'RNA, che è considerato promiscuo, a causa di accoppiamenti di basi che potrebbero essere inaspettati).
La tecnica si è dimostrata di grande successo nel creare una miriade di forme in 2 e 3 dimensioni, che convenientemente si autoassemblano quando le sequenze di DNA progettate vengono mescolate insieme. La parte difficile è preparare la corretta sequenza di DNA e il design del percorso per impalcature e filamenti di punti metallici al fine di ottenere la struttura target desiderata. Tipicamente, questo è un lavoro scrupoloso che deve essere eseguito manualmente.
Con la nuova tecnica, la struttura di destinazione viene prima descritta in termini di una rete metallica costituita da poliedri. Da questa, viene generato un algoritmo di spanning tree. Questa è fondamentalmente una mappa che guiderà automaticamente il percorso del filamento dell'impalcatura del DNA attraverso l'intera struttura dell'origami, toccando ogni vertice nella forma geometrica, una volta. Vengono quindi assegnati fili di graffette complementari e la forma finale si autoassembla.
Per testare il metodo, forme più semplici note come solidi platonici furono fabbricate per la prima volta, seguito da strutture sempre più complesse. Questi includevano oggetti con topologie non sferiche e dettagli interni insoliti, che non era mai stato realizzato sperimentalmente prima.
I progetti completati hanno dimostrato la capacità della tecnica top-down di generare automaticamente scaffold e percorsi di graffette per una vasta gamma di nanoforme, basata esclusivamente sulla geometria della superficie. Cryo-EM è stato utilizzato per confermare la fedeltà strutturale e la stabilità delle strutture di origami assemblate.
Ulteriori esperimenti hanno confermato che le strutture di DNA prodotte erano potenzialmente adatte per applicazioni biologiche poiché mostravano stabilità a lungo termine in condizioni sieriche ea basso contenuto di sale.
La ricerca apre la strada allo sviluppo di sistemi progettati su nanoscala che imitano le proprietà dei virus, organismi fotosintetici e altri prodotti sofisticati dell'evoluzione naturale.
Oltre alla sua nomina all'Istituto di Biodesign, Hao Yan è l'illustre professore di Milton D. Glick, Collegio delle Arti e delle Scienze Liberali, Scuola di Scienze Molecolari dell'ASU.