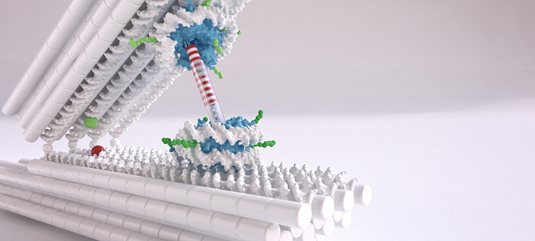
Credito:Chris Hohmann (NIM); Laboratorio Dietz (TUM)
La modalità di confezionamento del DNA genomico nel nucleo cellulare determina i modelli di espressione genica. I ricercatori di Monaco hanno utilizzato nano-pinzette basate sul DNA per misurare le forze tra nucleosomi, le unità di imballaggio di base del DNA nucleare.
Ogni cellula umana contiene circa due metri di acido desossiribonucleico (DNA), che codifica l'informazione genetica che specifica le strutture e le funzioni cellulari. Inoltre, questo DNA "genomico" è impacchettato nel nucleo della cellula, che ha un diametro inferiore a 10 micrometri. Ciò significa che il DNA nucleare deve essere imballato, principalmente interagendo con proteine specifiche. L'unità di imballaggio di base è una particella costituita da proteine chiamate istoni, intorno al quale è avvolto il DNA. Paragonabile a piccole bobine, queste strutture sono chiamate nucleosomi. I nucleosomi a loro volta sono collegati tra loro da segmenti di DNA che si estendono tra le particelle del nucleo e non sono avvolti attorno ad esse. Visto al microscopio elettronico, il DNA confezionato in nucleosomi assomiglia a perline su una corda.
Il livello successivo di confezionamento prevede l'interazione reciproca di nucleosomi, e le strutture di ordine superiore risultanti non sono ancora state completamente caratterizzate. Un team di scienziati guidati da Hendrik Dietz dell'Università tecnica di Monaco e Philipp Korber del Centro biomedico della LMU ha compiuto un passo sostanziale verso la risoluzione di questo enigma:per la prima volta in assoluto, sono riusciti a misurare direttamente le forze attrattive che agiscono tra i nucleosomi. I loro risultati appaiono sui giornali Progressi scientifici e Nano lettere .
Origami del DNA:integrazione dei nucleosomi nelle pinzette
Dietz, titolare della Cattedra di Biofisica Sperimentale presso il TUM, utilizza il DNA come materiale da costruzione per costruire strutture molecolari, una tecnologia denominata origami del DNA. Lui e il suo team hanno ora utilizzato il metodo per creare strutture costituite da due barre rigide di DNA collegate da un giunto flessibile che funge da molla. Questi possono essere usati come pinzette per misurare la forza delle interazioni tra i nucleosomi. Un nucleosoma è attaccato a ciascun braccio delle pinzette. "Possiamo controllare la posizione e l'orientamento dei nucleosomi nelle pinzette del DNA con un grado di precisione molto elevato, " dice Dietz. "Questo è molto importante quando si tratta di essere realmente in grado di misurare le interazioni".
I ricercatori della LMU si sono assunti il compito di sviluppare strutture di nucleosomi che possono essere integrate nelle pinzette. Philipp Korber, Privatdozent e Group Leader presso la Cattedra di Biologia Molecolare del BMC, spiega:"Normalmente le due estremità a doppio filamento del DNA avvolto attorno al nucleosoma sono molto vicine l'una all'altra. Ma ciò di cui avevamo bisogno erano due filamenti singoli sporgenti, più vicino al centro. Questo era un problema significativo, poiché una tale configurazione può destabilizzare l'intera struttura. Il nostro membro del team Corinna Lieleg è comunque riuscita a trovare i punti giusti per queste maniglie."
I ricercatori sono stati in grado di misurare un'interazione molto debole tra nucleosomi integrati, equivalente a una forza di attrazione di 1,6 kcal/mol, a una distanza di circa 6 nanometri (nm). Si è scoperto che gli orientamenti dei nucleosomi l'uno rispetto all'altro non hanno quasi alcun effetto. Però, particolari modificazioni chimiche nelle proteine istoniche hanno ulteriormente indebolito le interazioni.
Il problema della fibra a 30 nm
Il risultato potrebbe aiutare a risolvere un'attuale controversia scientifica. Secondo la teoria attuale, i nucleosomi formano un tipo di superspirale con un diametro di 30 nanometri, la cosiddetta fibra a 30 nm. Finora, però, questa struttura di ordine superiore a 30 nm non è mai stata osservata nelle cellule viventi. Che la cromatina assuma davvero o meno la forma di una tale super spirale è ancora molto controverso. Infatti, le minuscole forze attrattive tra i nucleosomi, che i ricercatori hanno ora misurato con successo, sembrano contraddire la teoria. "I nostri dati indicano strutture molto morbide che sono facilmente deformabili da influenze esterne, "dice Dietz.
Il modo in cui i nucleosomi sono organizzati in strutture di ordine superiore è una questione di fondamentale importanza, poiché ha profonde implicazioni per il controllo dell'espressione genica. Solo quei geni che si trovano all'interno della cromatina relativamente non compatta sono accessibili all'"attivazione", che consente alle proteine che codificano di essere prodotte dal macchinario cellulare.
La regolazione genica attraverso il confezionamento del DNA va storta nelle cellule tumorali
"Negli ultimi dieci anni è diventato chiaro che molti dei cambiamenti e delle mutazioni che trasformano le cellule in cellule cancerose avvengono a questo livello, " dice Korber. In una cellula cancerosa, i normali meccanismi che determinano quali geni sono attivi e quali sono inattivi vengono interrotti. Le regioni genomiche che non dovrebbero essere accessibili vengono lasciate aperte e viceversa. "Però, se solo l'imballaggio è difettoso, e non il gene stesso, in linea di principio dovrebbe essere possibile ripristinare nuovamente l'imballaggio adeguato".
I ricercatori intendono utilizzare la tecnica delle pinzette molecolari per studiare altre strutture. "In biologia l'orientamento delle strutture l'una rispetto all'altra è sempre importante, " dice Korber. "Ora abbiamo una sorta di morsetto molecolare che possiamo usare per controllare in modo specifico l'orientamento spaziale delle strutture l'una rispetto all'altra".