L'acido ribonucleico (RNA) viene utilizzato per creare circuiti logici in grado di eseguire vari calcoli. In nuovi esperimenti, Green e i suoi colleghi hanno incorporato le porte logiche dell'RNA nelle cellule batteriche viventi, che si comportano come piccoli computer. Credito:Jason Drees per il Biodesign Institute
Il nesso interdisciplinare tra biologia e ingegneria, nota come biologia sintetica, sta crescendo rapidamente, aprendo nuovi orizzonti che fino a poco tempo fa difficilmente si potevano immaginare.
In una nuova ricerca, Alex Verde, un professore all'Istituto di Biodesign dell'ASU, dimostra come le cellule viventi possono essere indotte a eseguire calcoli alla maniera di minuscoli robot o computer.
I risultati del nuovo studio hanno implicazioni significative per la progettazione intelligente dei farmaci e la somministrazione intelligente dei farmaci, produzione di energia verde, tecnologie diagnostiche a basso costo e persino lo sviluppo di futuristiche nanomacchine in grado di dare la caccia alle cellule cancerose o spegnere geni aberranti.
"Stiamo usando interazioni RNA-RNA molto prevedibili e programmabili per definire cosa possono fare questi circuiti, " dice Green. "Ciò significa che possiamo usare software per computer per progettare sequenze di RNA che si comportano nel modo in cui vogliamo che si comportino in una cellula. Rende il processo di progettazione molto più veloce."
Lo studio appare nell'edizione online anticipata della rivista Natura .
Designer RNA
L'approccio descritto utilizza circuiti composti da acido ribonucleico o RNA. Questi progetti di circuiti, che assomigliano ai circuiti elettronici convenzionali, autoassemblarsi nelle cellule batteriche, consentendo loro di percepire i messaggi in arrivo e rispondere ad essi producendo un particolare output computazionale, (in questo caso, una proteina).
Nel nuovo studio, circuiti specializzati noti come porte logiche sono stati progettati in laboratorio, poi incorporati nelle cellule viventi. I minuscoli interruttori del circuito scattano quando i messaggi (sotto forma di frammenti di RNA) si attaccano alle loro sequenze di RNA complementari nel circuito cellulare, attivando la porta logica e producendo un'uscita desiderata.
Gli interruttori RNA possono essere combinati in vari modi per produrre porte logiche più complesse in grado di valutare e rispondere a più ingressi, così come un semplice computer può prendere più variabili ed eseguire operazioni sequenziali come addizioni e sottrazioni per raggiungere un risultato finale.
Il nuovo studio migliora notevolmente la facilità con cui è possibile eseguire il calcolo cellulare. L'approccio del solo RNA alla produzione di nanodispositivi cellulari è un progresso significativo, poiché gli sforzi precedenti richiedevano l'uso di intermediari complessi, come le proteine. Ora, le parti di ribocalcolo necessarie possono essere facilmente progettate su computer. Le semplici proprietà di accoppiamento delle basi delle quattro lettere nucleotidiche dell'RNA (A, C, G e U) garantiscono il prevedibile autoassemblaggio e funzionamento di queste parti all'interno di una cellula vivente.
Il lavoro di Green in questo settore è iniziato presso il Wyss Institute di Harvard, dove ha contribuito a sviluppare il componente centrale utilizzato nei circuiti cellulari, noto come interruttore di presa dell'RNA. Il lavoro è stato svolto mentre Green era un post-doc che lavorava con l'esperto di nanotecnologie Peng Yin, insieme ai biologi sintetici James Collins e Pamela Silver, che sono tutti coautori del nuovo articolo. "I primi esperimenti sono stati nel 2012, " dice Green. "Fondamentalmente, i toehold si sono comportati così bene che volevamo trovare un modo per sfruttarli al meglio per le applicazioni cellulari."
Dopo essere arrivato all'ASU, Il primo studente di Green Duo Ma ha lavorato su esperimenti presso il Biodesign Institute, mentre un altro postdoc, Jongmin Kim ha continuato un lavoro simile al Wyss Institute. Entrambi sono anche coautori del nuovo studio.
Chip Pentium della natura
La possibilità di utilizzare DNA e RNA, le molecole della vita, eseguire calcoli simili a computer è stato dimostrato per la prima volta nel 1994 da Leonard Adleman della University of Southern California. Da allora, rapidi progressi hanno fatto avanzare considerevolmente il campo, e recentemente, tale calcolo molecolare è stato realizzato all'interno di cellule viventi. (Le cellule batteriche sono solitamente impiegate per questo scopo in quanto sono più semplici e facili da manipolare.)
La tecnica descritta nel nuovo articolo sfrutta il fatto che l'RNA, a differenza del DNA, è a singolo filamento quando è prodotto nelle cellule. Ciò consente ai ricercatori di progettare circuiti di RNA che possono essere attivati quando un filamento di RNA complementare si lega a una sequenza di RNA esposta nel circuito progettato. Questo legame di filamenti complementari è regolare e prevedibile, con A nucleotidi che si accoppiano sempre con U e C che si accoppiano sempre con G.
Con tutti gli elementi di elaborazione del circuito realizzati utilizzando RNA, che può assumere un numero astronomico di sequenze potenziali, il vero potere del metodo appena descritto risiede nella sua capacità di eseguire molte operazioni contemporaneamente. Questa capacità di elaborazione parallela consente calcoli più veloci e sofisticati, sfruttando al tempo stesso in modo efficiente le limitate risorse della cella.
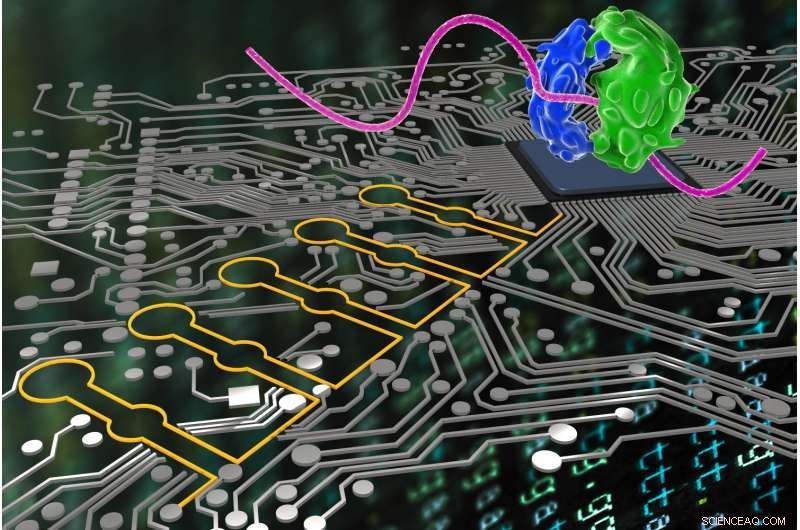
Simile a come gli scienziati informatici usano il linguaggio logico per fare in modo che i loro programmi eseguano AND accurati, O e NON decisioni verso un obiettivo finale, I "dispositivi di ribocomputing" (stilizzati qui in giallo) sviluppati da un team del Wyss Institute possono ora essere utilizzati dai biologi sintetici per rilevare e interpretare più segnali nelle cellule e istruire logicamente i loro ribosomi (stilizzati in blu e verde) per produrre proteine diverse. Credito:Wyss Institute presso l'Università di Harvard
Risultati logici
Nel nuovo studio, porte logiche note come AND, OR e NOT sono stati progettati. Una porta AND produce un output nella cella solo quando sono presenti due messaggi RNA A AND B. Una porta OR risponde a A OR B, mentre una porta NOT bloccherà l'output se è presente un dato input RNA. La combinazione di queste porte può produrre una logica complessa in grado di rispondere a più ingressi.
Usando gli interruttori di presa dell'RNA, i ricercatori hanno prodotto i primi dispositivi di ribocalcolo capaci di quattro ingressi AND, OR a sei ingressi e un dispositivo a 12 ingressi in grado di eseguire una complessa combinazione di AND, OR e NOT logica nota come espressione disgiuntiva in forma normale. Quando la porta logica incontra le corrette sequenze di legame dell'RNA che portano all'attivazione, si apre un interruttore di presa e ha luogo il processo di traduzione in proteina. Tutte queste funzioni di rilevamento del circuito e di uscita possono essere integrate nella stessa molecola, rendendo i sistemi compatti e più facili da implementare in una cella.
La ricerca rappresenta la fase successiva del lavoro in corso utilizzando gli interruttori di presa RNA altamente versatili. In lavori precedenti, Green e i suoi colleghi hanno dimostrato che un prodotto economico, una serie cartacea di interruttori di presa dell'RNA potrebbe fungere da piattaforma altamente accurata per la diagnosi del virus Zika. Il rilevamento dell'RNA virale da parte dell'array ha attivato gli interruttori di presa, innescare la produzione di una proteina, che è stato registrato come un cambiamento di colore sull'array.
Il principio di base dell'utilizzo di dispositivi basati su RNA per regolare la produzione di proteine può essere applicato praticamente a qualsiasi input di RNA, inaugurando una nuova generazione di accurati, diagnostica a basso costo per un'ampia gamma di malattie. L'approccio cell-free è particolarmente adatto per le minacce emergenti e durante le epidemie di malattie nei paesi in via di sviluppo, dove le risorse mediche e il personale possono essere limitate.
Il computer dentro
Secondo Verde, la fase successiva della ricerca si concentrerà sull'uso della tecnologia dell'RNA toehold per produrre le cosiddette reti neurali all'interno delle cellule viventi, circuiti in grado di analizzare una serie di input eccitatori e inibitori, mediandoli e producendo un output una volta raggiunta una determinata soglia di attività, allo stesso modo in cui un neurone calcola la media dei segnali in arrivo da altri neuroni. In definitiva, i ricercatori sperano di indurre le cellule a comunicare tra loro tramite segnali molecolari programmabili, formando un vero e proprio interattivo, rete simile al cervello.
"Perché stiamo usando l'RNA, una molecola universale di vita, sappiamo che queste interazioni possono funzionare anche in altre cellule, quindi il nostro metodo fornisce una strategia generale che potrebbe essere trasferita ad altri organismi, "Il verde dice, alludendo a un futuro in cui le cellule umane diventano entità completamente programmabili con ampie capacità biologiche.