Credito:American Chemical Society
Scienziati di tutto il mondo si stanno affrettando a trovare inibitori di SARS-CoV-2, il nuovo coronavirus dietro la pandemia di COVID-19. Alcuni utilizzano simulazioni al computer per identificare composti promettenti prima di condurre esperimenti reali in laboratorio. Ora, ricercatori che riferiscono in ACS Nano hanno utilizzato la modellazione al computer per valutare quattro peptidi che imitano il dominio di legame del virus della proteina umana che consente a SARS-CoV-2 di entrare nelle cellule.
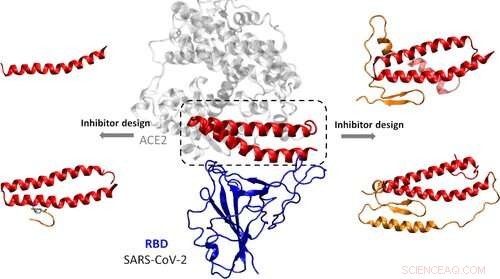
Per infettare le cellule, SARS-CoV-2 utilizza la sua proteina spike per legarsi al recettore ACE2, una proteina sulla superficie di alcune cellule umane. Questo attaccamento consente al virus di fondersi con la membrana della cellula ospite e di entrare. Molti ricercatori hanno cercato di trovare composti che bloccano le regioni chiave della proteina spike, impedendo al virus di infettare le cellule. Yanxiao Han e Petr Král volevano utilizzare la modellazione al computer per progettare composti che imitassero il bersaglio naturale della proteina spike, ACE2.
Fare così, i ricercatori hanno esaminato la struttura cristallina a raggi X recentemente pubblicata del dominio di legame al recettore di SARS-CoV-2 quando è legato ad ACE2. Hanno identificato 15 amminoacidi da ACE2 che interagiscono direttamente con la proteina virale. Quindi, i ricercatori hanno progettato quattro inibitori che contengono la maggior parte o tutti questi amminoacidi, con sequenze aggiuntive che pensavano avrebbero stabilizzato le strutture. Attraverso simulazioni al computer, il team ha studiato come gli inibitori potrebbero legarsi alla proteina spike nel corpo e le energie necessarie per il legame. Uno dei composti ha mostrato un adattamento particolarmente buono con la proteina virale. Il peptide deve ancora essere testato in laboratorio e sui pazienti, ma essere in grado di restringere i candidati ai farmaci sul computer potrebbe aiutare ad accelerare questo processo, dice la squadra.