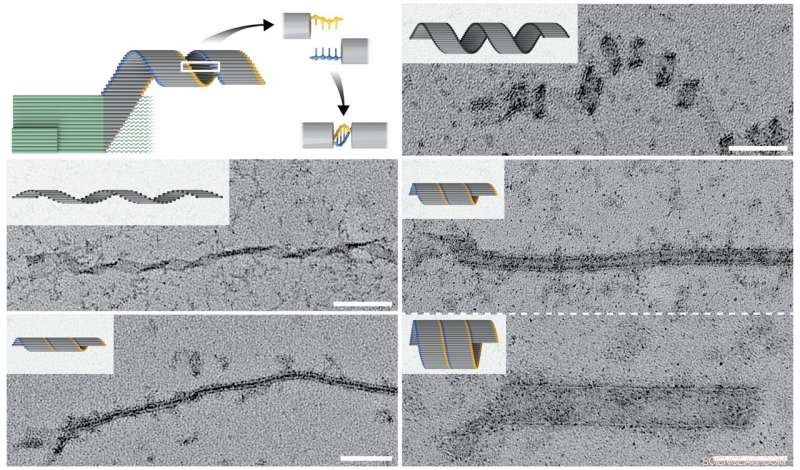
La polimerizzazione incrociata strettamente dipendente dal seme (verde) consente la formazione di tubi di forma diversa e nastri arrotolati (grigio), per cui nastri allungati con diametri diversi sono chiusi in diversi modelli da brevi sporgenze di DNA a singolo filamento complementari (giallo e blu). Questa serie di immagini TEM mostra una varietà di nanocostrutti allungati con la barra della scala che misura 100 nanometri. Credito:Wyss Institute presso l'Università di Harvard
Un team di nanobiotecnologi presso il Wyss Institute for Biologically Inspired Engineering di Harvard e il Dana-Farber Cancer Institute (DFCI) guidato da William Shih, membro della Wyss Founding Core Faculty, dottorato di ricerca, ha ideato una strategia di autoassemblaggio del DNA programmabile che risolve la sfida chiave di un robusto controllo della nucleazione e apre la strada ad applicazioni come il rilevamento di biomarcatori diagnostici ultrasensibili e la fabbricazione scalabile di strutture di dimensioni micrometriche con caratteristiche di dimensioni nanometriche.
Utilizzando il metodo, chiamata "polimerizzazione incrociata", i ricercatori possono iniziare la tessitura di nanonastri da singoli filamenti allungati di DNA (denominati "lamelle") mediante un evento di nucleazione strettamente dipendente dal seme. Lo studio è pubblicato su Comunicazioni sulla natura .
Le nanostrutture di DNA hanno un grande potenziale per risolvere vari problemi diagnostici, terapeutico, e sfide di fabbricazione a causa della loro elevata biocompatibilità e programmabilità. Per far funzionare dispositivi diagnostici efficaci, Per esempio, una nanostruttura di DNA potrebbe dover rispondere in modo specifico alla presenza di una molecola bersaglio innescando una lettura amplificata compatibile con strumenti a basso costo accessibili in ambienti point-of-care o clinico-laboratorio.
La maggior parte delle nanostrutture di DNA vengono assemblate utilizzando una delle due strategie principali, ciascuna con i propri punti di forza e i propri limiti. Gli "origami di DNA" sono formati da un lungo filamento di impalcatura a singolo filamento che è stabilizzato in una configurazione bidimensionale o tridimensionale da numerosi filamenti di fiocco più corti. Il loro assemblaggio è strettamente dipendente dal trefolo dell'impalcatura, portando a una robusta piegatura tutto o niente. Sebbene possano essere formati con elevata purezza in un'ampia gamma di condizioni, la loro dimensione massima è limitata. I "mattoni DNA" d'altra parte possono assemblare strutture molto più grandi da una moltitudine di brevi filamenti modulari. Però, il loro assemblaggio richiede condizioni ambientali strettamente controllate, può essere falsamente iniziata in assenza di un seme, e produce una proporzione significativa di strutture incomplete che devono essere purificate.
"L'introduzione dell'origami del DNA è stato il singolo progresso di maggior impatto nel campo della nanotecnologia del DNA negli ultimi due decenni. L'approccio di polimerizzazione incrociata che abbiamo sviluppato in questo studio si basa su questo e su altri fondamenti per estendere l'autoassemblaggio controllato del DNA a molto scale di lunghezza maggiore, " disse Shih, che co-guida l'iniziativa di robotica molecolare di Wyss, ed è anche professore alla Harvard Medical School e DFCI. "Prevediamo che la polimerizzazione incrociata consentirà ampiamente la formazione del tutto o niente di microstrutture bidimensionali e tridimensionali con caratteristiche indirizzabili su nanoscala, autoassemblaggio algoritmico, e amplificazione del segnale a sfondo zero in applicazioni diagnostiche che richiedono estrema sensibilità."
Piantare un seme
Avendo sperimentato i limiti degli origami di DNA e delle nanostrutture di mattoni di DNA, il team ha iniziato chiedendo se fosse possibile combinare l'assoluta dipendenza dal seme dell'assemblaggio di origami di DNA con le dimensioni illimitate delle costruzioni in mattoni di DNA in un terzo tipo di nanostruttura di DNA che cresce rapidamente e costantemente fino a raggiungere grandi dimensioni.
"Abbiamo sostenuto che l'assemblaggio tutto o niente di strutture di DNA su scala micron potrebbe essere ottenuto progettando un sistema che abbia un'alta barriera di energia libera all'assemblaggio spontaneo. La barriera può essere aggirata solo con un seme che si lega e organizza un insieme di lamelle "nucleate" per la cattura congiunta delle lamelle "di crescita". Ciò avvia una reazione a catena di aggiunte di lamelle di crescita che si traduce in lunghi nastri di DNA, " ha detto il co-primo autore Dionis Minev, dottorato di ricerca, che è un borsista post-dottorato nella squadra di Shih.
"Questo tipo di attività altamente cooperativa, la nucleazione strettamente dipendente dal seme segue alcuni degli stessi principi che regolano l'inizio e la crescita dell'actina citoscheletrica o dei filamenti dei microtubuli nelle cellule." a sua volta è necessario per il legame di quello successivo." La polimerizzazione incrociata porta questa strategia al livello successivo, consentendo ai vicini non più vicini di essere richiesti per il reclutamento dei monomeri in entrata. Il risultante livello estremo di coordinazione è la salsa segreta, " disse Minev.
Dal concetto alla struttura effettiva
Mettendo in pratica il loro concetto, il team ha progettato e convalidato un sistema in cui una minuscola struttura di semi offre un'elevata concentrazione iniziale di siti di legame preformati sotto forma di singoli filamenti di DNA sporgenti. Questi possono essere rilevati da lamelle di DNA con sei (o in un sistema incrociato alternativo otto) siti di legame disponibili, ciascuno si lega a uno dei sei (o otto) filamenti di ssDNA sporgenti adiacenti in uno schema incrociato, e le successive stecche di DNA vengono quindi aggiunte continuamente alla struttura in allungamento.
"Il nostro design è notevole perché abbiamo ottenuto una rapida crescita di enormi strutture di DNA, ma con un controllo della nucleazione che è maggiore di ordini di grandezza rispetto ad altri approcci. È come avere la tua torta e mangiarla anche tu, perché creavamo prontamente assemblee su larga scala e lo facevamo solo dove e quando lo desideravamo, " ha detto il co-primo autore Chris Wintersinger, un dottorato di ricerca studente del gruppo di Shih che ha collaborato al progetto con Minev. "Il controllo che abbiamo ottenuto con incrociato supera di gran lunga quello osservato per i metodi esistenti sul DNA in cui la nucleazione può essere diretta solo all'interno di una finestra ristretta di condizioni in cui la crescita è estremamente lenta".
Utilizzando la polimerizzazione incrociata, Il team di Shih ha generato nastri di DNA che si sono autoassemblati come risultato di un singolo evento di semina specifico in strutture che misuravano fino a decine di micrometri di lunghezza, con una massa quasi cento volte più grande di un tipico origami di DNA. Inoltre, sfruttando l'elevata programmabilità delle conformazioni e interazioni delle lamelle, i ricercatori hanno creato nastri con curve e colpi di scena distinti, con conseguente struttura a spirale e tubolare.
Negli studi futuri, questo potrebbe essere sfruttato per creare strutture funzionalizzate che possono beneficiare di compartimenti spazialmente separati. "Un'applicazione immediata per il nostro metodo di nanocostruzione incrociata è come strategia di amplificazione nei test diagnostici a seguito della formazione di nanosemi da biomarcatori specifici e rari, " ha detto la co-autrice Anastasia Ershova, che è anche un Ph.D. studente guidato da Shih.
"Lo sviluppo di questo nuovo metodo di nanofabbricazione è un esempio lampante di come la Molecular Robotics Initiative del Wyss Institute continui ad essere ispirata dai sistemi biologici, in questo caso, filamenti citoscheletrici in crescita, e continua ad espandere le possibilità in questo entusiasmante campo. Questo progresso avvicina il potenziale della nanotecnologia del DNA alla soluzione di pressanti sfide diagnostiche per le quali attualmente non esistono soluzioni, ", ha affermato il direttore fondatore di Wyss, Donald Ingber, M.D., dottorato di ricerca, che è anche Judah Folkman Professor of Vascular Biology presso la Harvard Medical School e il Boston Children's Hospital, e Professore di Bioingegneria presso la Harvard John A. Paulson School of Engineering and Applied Sciences.