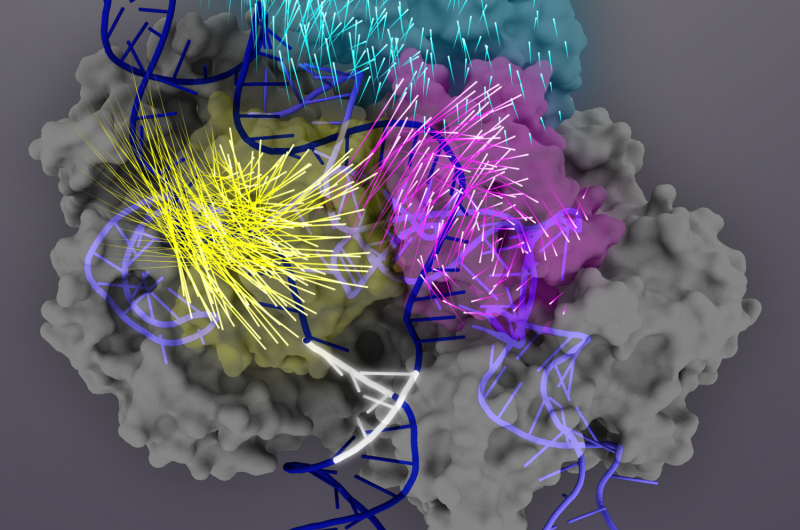
La proteina Cas9 (grigia) è una nucleasi guidata da RNA che può essere programmata per legare e tagliare qualsiasi sequenza di DNA corrispondente (doppia elica blu scuro), rendendolo un potente strumento per l'ingegneria genomica. Dopo il legame del bersaglio, I domini proteici Cas9 subiscono riarrangiamenti conformazionali (i movimenti dei singoli amminoacidi sono rappresentati dalle code dei razzi) per attivare il complesso Cas9-sgRNA per la scissione del bersaglio. Il dominio REC3 (verde acqua) è responsabile del rilevamento del bersaglio, che segnala la rotazione verso l'esterno del dominio REC2 (magenta) per aprire un percorso per il dominio nucleasi HNH (giallo). Questa conformazione attiva di Cas9 è quindi in grado di innescare la scissione concertata di entrambi i filamenti del DNA bersaglio. Credito:Janet Iwasa grafica per Doudna Lab
Scienziati dell'Università della California, Berkeley e il Massachusetts General Hospital hanno identificato una regione chiave all'interno della proteina Cas9 che regola l'accuratezza con cui CRISPR-Cas9 si posiziona su una sequenza di DNA bersaglio, e l'abbiamo ottimizzato per produrre un editor genetico iper-accurato con il livello più basso di taglio fuori bersaglio fino ad oggi.
Il dominio proteico che i ricercatori hanno identificato come controllore principale del taglio del DNA è un obiettivo ovvio per la riprogettazione per migliorare ulteriormente la precisione, dicono i ricercatori. Questo approccio dovrebbe aiutare gli scienziati a personalizzare le varianti di Cas9, la proteina che lega e taglia il DNA, per ridurre al minimo la possibilità che CRISPR-Cas9 modifichi il DNA nel posto sbagliato, una considerazione chiave quando si esegue la terapia genica negli esseri umani.
Una strategia per ottenere una maggiore precisione consiste nel creare mutazioni nel dominio proteico di governo, chiamato REC3, e vedere quali migliorano la precisione senza influire sull'efficienza del taglio sul bersaglio.
"Abbiamo scoperto che anche piccole alterazioni nel dominio REC3 di Cas9 influenzano il differenziale tra l'editing on e off-target, il che suggerisce che questo dominio è un candidato ovvio per una mutagenesi approfondita per migliorare la specificità del targeting. Come estensione di questo lavoro, si potrebbe eseguire una mutagenesi più imparziale all'interno di REC3 rispetto alle mutazioni mirate che abbiamo fatto, " ha detto la co-autrice Janice Chen, uno studente laureato nel laboratorio di Jennifer Doudna, che ha co-inventato lo strumento di modifica genetica CRISPR-Cas9.
Co-primi autori Chen, Yavuz Dagdas e Benjamin Kleinstiver, e i loro colleghi dell'Università di Berkeley, Il Massachusetts General Hospital e l'Università di Harvard riportano i loro risultati online oggi prima della pubblicazione sulla rivista Natura .
Cas9 Hyper iper-preciso
Dal 2012, quando Doudna, un professore di biologia molecolare e cellulare e un investigatore dell'Howard Hughes Medical Institute presso l'UC Berkeley, e la collega Emmanuelle Charpentier del Max Planck Institute for Infection Biology hanno riproposto la proteina Cas9 per creare un editor genetico preciso e facile da usare, i ricercatori hanno cercato di ridurre le possibilità di editing fuori bersaglio. Mentre una migliore fedeltà va a vantaggio della ricerca di base, è assolutamente fondamentale quando si modificano i geni per applicazioni cliniche, poiché qualsiasi taglio del DNA fuori bersaglio potrebbe disabilitare i geni chiave e portare a permanenti, effetti collaterali imprevisti.
Negli ultimi due anni, due team hanno progettato proteine Cas9 altamente accurate - una a specificità potenziata chiamata eSpCas9(1.1) e una ad alta fedeltà chiamata SpCas9-HF1 - e Chen e Doudna hanno cercato di capire perché tagliassero con una specificità maggiore rispetto alla proteina Cas9 wild-type di Streptococcus pyogenes oggi ampiamente utilizzato.
Attualmente, i ricercatori che utilizzano CRISPR-Cas9 creano un RNA a guida singola (sgRNA) - una molecola di RNA che include una catena di 20 acidi ribonucleici che integra una specifica sequenza di DNA di 20 acidi nucleici che vogliono prendere di mira e la attaccano a Cas9. Questa guida RNA consente a Cas9 di fissarsi sul DNA complementare, legare ad esso e tagliare l'elica a doppio filamento. Ma il complesso Cas9-sgRNA può anche legarsi al DNA che non corrisponde esattamente, portando a un taglio fuori bersaglio indesiderato.
Nel 2015, Il laboratorio di Doudna ha scoperto un interruttore conformazionale di Cas9 che viene attivato quando la guida dell'RNA e il bersaglio del DNA corrispondono. Hanno scoperto che solo quando l'RNA e il DNA corrispondono strettamente la struttura 3D di Cas9, in particolare la conformazione del dominio nucleasico HNH, cambia e attiva le forbici di Cas9. Però, il processo responsabile del rilevamento degli acidi nucleici a monte dell'interruttore conformazionale è rimasto sconosciuto.
Nello studio attuale, Chen e Dagdas hanno utilizzato una tecnica chiamata FRET a singola molecola (trasferimento di energia di risonanza di Förster) per misurare con precisione come i vari domini proteici nel complesso proteico Cas9-sgRNA - in particolare REC3, REC2 e HNH - si muovono quando il complesso si lega al DNA.
In primo luogo hanno determinato che i benefici di specificità conferiti da eSpCas9 (1.1) e SpCas9-HF1 potrebbero essere spiegati dal fatto che la soglia per l'interruttore conformazionale HNH era molto più alta per queste varianti Cas9 rispetto alla proteina Cas9 wild-type, rendendo le varianti eSpCas9(1.1) e SpCas9-HF1 meno propense ad attivare le forbici quando legate a una sequenza fuori bersaglio.
Prossimo, hanno scoperto che il dominio REC3 è responsabile del rilevamento dell'accuratezza del legame del bersaglio, che quindi segnala la rotazione verso l'esterno del dominio REC2 per aprire un percorso per il dominio della nucleasi HNH, attivando le forbici. Questa conformazione attiva di Cas9 è quindi in grado di scindere entrambi i filamenti del DNA bersaglio.
Chen, Dagdas e Kleinstiver hanno poi dimostrato che mutando parti di REC3, è possibile modificare la specificità della proteina Cas9 in modo che la nucleasi HNH non venga attivata a meno che l'RNA guida e il DNA target siano molto vicini. Sono stati in grado di progettare un Cas9 iper-preciso migliorato, soprannominato HypaCas9, che mantiene la sua efficienza sul bersaglio ma è leggermente migliore nel discriminare tra siti on e off-target nelle cellule umane.
"Se si mutano alcuni residui di amminoacidi in REC3, puoi modificare l'equilibrio tra l'attività sul bersaglio di Cas9 e una migliore specificità; siamo stati in grado di trovare il punto debole in cui c'è un'attività sufficiente al target previsto ma anche una grande riduzione degli eventi fuori target, " disse Chen.
Continuando ad esplorare le relazioni tra struttura, funzione e dinamica di Cas9, Doudna e il suo team sperano di progettare ulteriormente la proteina con una sensibilità squisita per eseguire in modo affidabile ed efficiente una varietà di alterazioni genetiche.