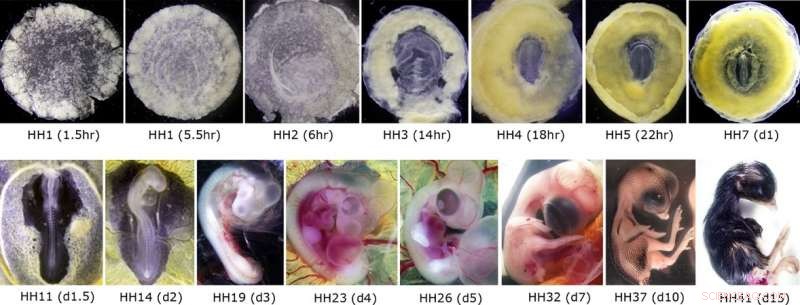
Immagini rappresentative di embrioni di pollo utilizzati per la preparazione dell'RNA e il successivo sequenziamento CAGE. I pannelli superiori mostrano gli embrioni nel primo giorno di sviluppo embrionale (21 giorni in totale), equivalente alle prime cinque settimane di gestazione umana (prime tre settimane di sviluppo umano post-fecondazione). I pannelli inferiori mostrano gli embrioni dalla costituzione del sistema circolatorio a circa una settimana prima della schiusa, simile all'inizio del terzo trimestre nello sviluppo umano. Credito:Professor Guojun Sheng
La ricerca sullo sviluppo precoce sugli embrioni umani è impossibile a causa di considerazioni etiche, ed è tecnicamente difficile da eseguire in altri mammiferi, rendendo le risorse per questo tipo di ricerca estremamente rare. Per esempio, il progetto Functional Annotation of Mammalian Genome (FANTOM) 5 con sede in Giappone, fondata nel 2000, è un progetto collaborativo mondiale con l'obiettivo di identificare tutti gli elementi funzionali nei genomi dei mammiferi, ma non ha dati sul sito di inizio della trascrizione (TSS) basati sulla Cap Analysis of Gene Expression (CAGE) per embrioni umani o per embrioni murini che rappresentano le prime fasi dello sviluppo.
Per aggirare questa scarsità di informazioni, una collaborazione internazionale di ricercatori dal Giappone (Kumamoto University &RIKEN), Russia (Università Federale di Kazan), Spagna (Università della Cantabria), e l'Australia (University of Western Australia) ha messo insieme il primo set al mondo di TSS aviari per l'intero genoma. Questi siti segnano il punto di partenza per la trascrizione e sono estremamente importanti per la differenziazione cellulare e lo sviluppo embrionale fino alla nascita, o, nel caso di una specie aviaria, fino alla schiusa. Mentre gli uccelli sembrano molto diversi dagli umani, le prime fasi dello sviluppo sono molto simili, ed è stato dimostrato che circa il 60% dei geni codificanti proteine umane ha una corrispondenza biunivoca con gli ortologhi di pollo (geni che si sono evoluti da un antenato comune).
Utilizzando la tecnologia CAGE, un metodo altamente affidabile per trovare TSS ed elementi cis-regolatori (quei blocchi di sequenza che regolano la posizione e la robustezza della trascrizione) nel genoma durante l'intero periodo di sviluppo del pulcino, i collaboratori sono stati in grado di mappare il 60% di tutti i TSS di pollo in via di sviluppo al genoma di pollo più recente, con il restante 40% probabilmente costituito da promotori alternativi ancora non caratterizzati o geni RNA non codificanti. Il genoma aggiornato è stato aggiunto al programma interattivo di RIKEN, piattaforma online ZENBU (la parola giapponese "zenbu" si traduce in "tutti" in inglese.) con il nome "Chicken-ZENBU" ed è gratuita per tutti.
I ricercatori hanno anche dimostrato che la loro mappatura TSS dell'intero genoma potrebbe essere applicata alla tecnologia CRISPR-on per attivare geni specifici durante lo sviluppo attivando con successo il gene T per la codifica della proteina Brachyury nella fase di sviluppo HH10 (circa 1,5 giorni di sviluppo dopo la deposizione). Questo risultato è stato seguito da diverse attivazioni geniche di maggior successo, mostrando così l'efficacia di CRISPR-on quando combinato con la mappatura TSS.
"Nel processo di sviluppo del nostro metodo di profilazione TSS per lo sviluppo basato su CAGE, abbiamo identificato nuovi fattori di trascrizione e i loro moduli regolatori, così come diversi nuovi geni domestici, " ha affermato il leader del progetto, il professor Guojun Sheng dell'IRCMS dell'Università di Kumamoto. "Ci aspettiamo che questo lavoro e i dati che abbiamo aggiunto al database online ZENBU facciano avanzare notevolmente la ricerca sullo sviluppo sia negli uccelli che nei mammiferi".