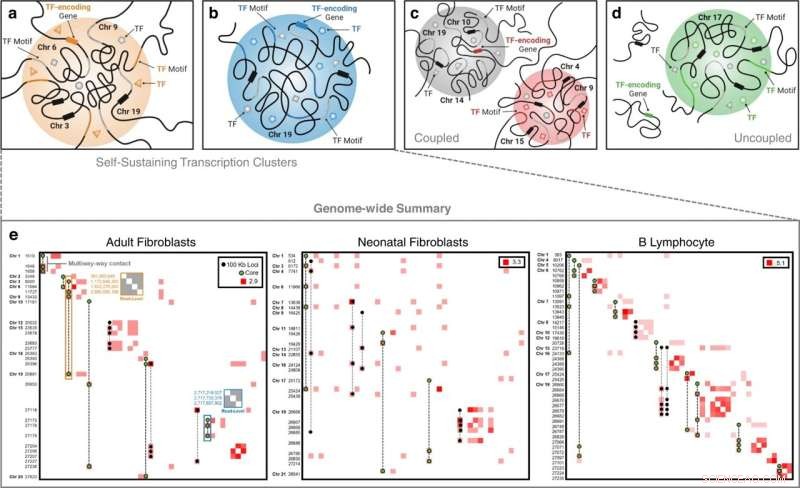
Classi di cluster di trascrizione. In un cluster di trascrizione autosufficiente, sono presenti un TF e il gene che codifica per quel TF. Gli esempi inter- e intra-cromosomici in (a) e (b), rispettivamente, illustrano questo fenomeno dove in a vediamo il TF di interesse (triangolo arancione) che circola al grappolo, il suo motivo di legame presente sulla cromatina (porzione arancione ), e il suo gene corrispondente espresso (rettangolo arancione sul cromosoma 6). Le forme grigie rappresentano ulteriori TF con motivi vincolanti (porzione grigia della cromatina) nel cluster. I rettangoli neri sui cromosomi 3, 9 e 19 rappresentano geni aggiuntivi presenti nel cluster. c Una classe indipendente dall'analogo di cluster di trascrizione in cui osserviamo un legame TF (quadrato rosso) a un cluster di trascrizione (cluster rosso) e il suo gene corrispondente espresso in un cluster di trascrizione separato (cluster grigio), ma non nello stesso cluster. d Una classe indipendente dall'analogo di cluster di trascrizione in cui osserviamo un legame TF (cerchio verde) a un cluster di trascrizione (cluster verde) e il suo gene corrispondente espresso ma non all'interno di un cluster di trascrizione. e Cluster di trascrizione autosufficienti specifici del tipo di cellula dell'intero genoma estratti da dati di contatto a più vie e decomposti in matrici di contatto Hi-C con una risoluzione di 100 kb. Le frequenze di contatto vengono trasformate in log per una migliore visualizzazione. Le frequenze lungo la diagonale indicano l'interazione tra due o più loci a più vie univoci che rientrano nello stesso bin di 100 kb. Le etichette degli assi sono coordinate bin 100 kb non contigue in ordine cromosomico. I contatti multidirezionali che costituiscono i cluster di trascrizione autosufficienti sono sovrapposti. I contatti a più vie con loci di colore verde rappresentano i cluster di trascrizione "core" - cluster di trascrizione contenenti un regolatore principale e il suo analogo del gene. Un esempio di mappa di contatto a livello di lettura per il cluster di trascrizione autosufficiente intercromosomica FOXO3 è indicata dalla casella evidenziata in arancione nella matrice di contatto di fibroblasti adulti e una mappa di contatto a livello di lettura per il cluster di trascrizione autosostenibile ZNF320 intra-cromosomica è indicato dalla casella evidenziata in blu. I valori lungo l'asse sinistro di queste matrici di contatto a livello di lettura sono le posizioni delle coppie di basi dei loci di contatto nel genoma. Credito:Comunicazioni sulla natura (2022). DOI:10.1038/s41467-022-32980-z
La terza legge dello scrittore di fantascienza Arthur C. Clarke afferma che "qualsiasi tecnologia sufficientemente avanzata è indistinguibile dalla magia".
Indika Rajapakse, Ph.D., è una credente. L'ingegnere e matematico ora si ritrova un biologo. E crede che la bellezza di fondere queste tre discipline sia fondamentale per svelare come funzionano le cellule.
Il suo ultimo sviluppo è una nuova tecnica matematica per iniziare a capire come è organizzato il nucleo di una cellula. La tecnica, che Rajapakse e collaboratori hanno testato su diversi tipi di cellule, ha rivelato ciò che i ricercatori hanno definito cluster di trascrizione autosufficienti, un sottoinsieme di proteine che svolgono un ruolo chiave nel mantenimento dell'identità cellulare.
Sperano che questa comprensione esponga vulnerabilità che possono essere prese di mira per riprogrammare una cellula per fermare il cancro o altre malattie.
"Sempre più biologi del cancro pensano che l'organizzazione del genoma svolga un ruolo enorme nella comprensione della divisione cellulare incontrollabile e se possiamo riprogrammare una cellula cancerosa. Ciò significa che dobbiamo comprendere più dettagli su ciò che sta accadendo nel nucleo", ha affermato Rajapakse, professore associato di computazionale medicina e bioinformatica, matematica e ingegneria biomedica presso l'Università del Michigan. È anche membro dell'UM Rogel Cancer Center.
Rajapakse è l'autore senior del documento, pubblicato su Nature Communications . Il progetto è stato guidato da un trio di studenti laureati con un team interdisciplinare di ricercatori.
Il team ha migliorato una vecchia tecnologia per esaminare la cromatina, chiamata Hi-C, che mappa quali pezzi del genoma sono vicini tra loro. Può identificare le traslocazioni cromosomiche, come quelle che si verificano in alcuni tumori. La sua limitazione, tuttavia, è che vede solo queste regioni genomiche adiacenti.
La nuova tecnologia, chiamata Pore-C, utilizza molti più dati per visualizzare come interagiscono tutti i pezzi all'interno del nucleo di una cellula. I ricercatori hanno utilizzato una tecnica matematica chiamata ipergrafi. Pensa:diagramma di Venn tridimensionale. Consente ai ricercatori di vedere non solo coppie di regioni genomiche che interagiscono, ma la totalità delle complesse e sovrapposte relazioni genomiche all'interno delle cellule.
"Questa relazione multidimensionale possiamo capirla in modo inequivocabile. Ci offre un modo più dettagliato per comprendere i principi organizzativi all'interno del nucleo. Se lo capisci, puoi anche capire dove deviano questi principi organizzativi, come nel cancro", ha detto Rajapakse. "È come mettere insieme tre mondi - tecnologia, matematica e biologia - per studiare più dettagli all'interno del nucleo".
I ricercatori hanno testato il loro approccio su fibroblasti neonatali, fibroblasti adulti sottoposti a biopsia e linfociti B. Hanno identificato organizzazioni di cluster di trascrizione specifici per ogni tipo di cellula. Hanno anche trovato quelli che chiamavano cluster di trascrizione autosufficienti, che fungono da firme trascrizionali chiave per un tipo di cellula.
Rajapakse descrive questo come il primo passo in un quadro più ampio.
"Il mio obiettivo è costruire questo tipo di immagine sul ciclo cellulare per capire come una cellula attraversa diverse fasi. Il cancro è una divisione cellulare incontrollabile", ha detto Rajapakse. "Se capiamo come una cellula normale cambia nel tempo, possiamo iniziare a esaminare i sistemi controllati e non controllati e trovare modi per riprogrammare quel sistema". + Esplora ulteriormente