L'interferenza dell'RNA (RNAi) è un affascinante processo biologico in vermi, piante, funghi e metazoi che si è rivelato uno strumento prezioso per studiare la funzione genetica e come agente terapeutico.
Nella Caenorhabditis elegans, la proteina transmembrana multipass, la proteina difettosa di interferenza sistemica dell'RNA 1 (SID-1), svolge un ruolo indispensabile nell'assorbimento e nel rilascio dell'RNA a doppio filamento (dsRNA) tra cellule e tessuti, portando all'RNAi sistemico.
Inoltre, due omologhi umani di SID-1, il membro 1 della famiglia transmembrana SID1 (SIDT1) e SIDT2, sono stati implicati nel trasporto dell'RNA. Tuttavia, i meccanismi molecolari alla base del modo in cui SID-1 distingue specificamente il dsRNA dall'RNA a filamento singolo (ssRNA) e dal DNA e facilita il successivo trasporto del dsRNA tra le cellule rimangono sconosciuti.
Le risposte a queste domande sono importanti per comprendere l'RNAi sistemico e per facilitare le applicazioni relative all'RNA.
Il Dr. Zhang Jiangtao del gruppo del Prof. Jiang Daohua dell'Istituto di Fisica dell'Accademia Cinese delle Scienze, ha dimostrato come SID-1 riconosca specificamente il dsRNA e ha fornito importanti approfondimenti sull'internalizzazione del dsRNA da parte del SID-1 combinando cryo-EM, esperimenti in vitro e in vivo. Il lavoro è pubblicato sulla rivista Nature Structural &Molecular Biology .
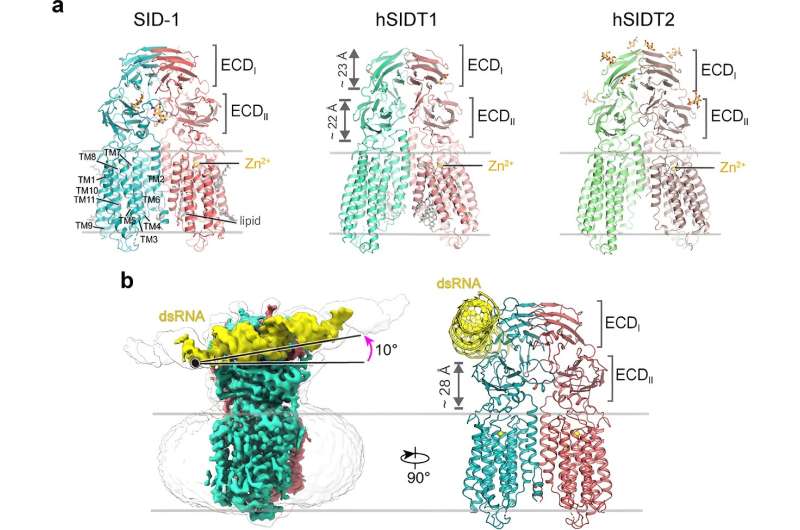
Per più di due decenni, si è pensato che SID-1 funzionasse come un canale dsRNA. Qui, i ricercatori hanno risolto le strutture crio-EM ad alta risoluzione del SID-1 e degli omologhi del SID-1 umano SIDT1 e SIDT2, rivelando l'architettura conservata di C. elegans e degli omologhi del SID-1 umano.
Gli omologhi SID-1 sono organizzati in modo omodimerico. Sorprendentemente, il dimero SID-1 non mostra un poro evidente all'interno del dominio transmembrana, suggerendo che SID-1 potrebbe non funzionare come canale dsRNA. I test di legame MST hanno confermato che SID-1 può legarsi in modo potente e specifico al dsRNA ma non al dsDNA.
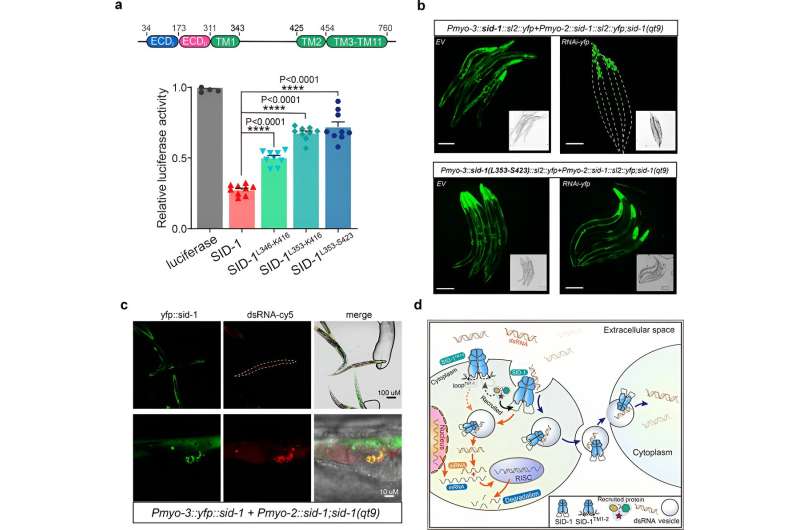
Successivamente, i ricercatori hanno ottenuto la struttura crio-EM del complesso SID-1-dsRNA, dimostrando la modalità dettagliata di legame del dsRNA e i determinanti molecolari di come SID-1 distingue il dsRNA dallo ssRNA e dal DNA.
È interessante notare che tali determinanti non sono presenti nel SIDT1 o nel SIDT2 umano. I risultati strutturali sono stati supportati da studi di mutagenesi utilizzando test di legame MST, assorbimento di dsRNA nelle cellule S2 e test di RNAi sistemici in vivo.
Infine, i ricercatori mostrano che la rimozione delle lunghe eliche transmembrana del ciclo intracellulare 1 e 2 non ha influenzato la dimerizzazione del SID-1, la localizzazione cellulare o il legame del dsRNA, ma ha compromesso significativamente l’assorbimento del dsRNA nelle cellule S2 e l’RNAi sistemico in C. elegans.
Inoltre, la co-localizzazione ha rivelato che SID-1 e dsRNA si localizzano in organelli subcellulari simili a vescicole. Sulla base di questi risultati, i ricercatori propongono che SID-1 funzioni come un recettore del dsRNA e faciliti la successiva internalizzazione del dsRNA reclutando proteine correlate agli endociti attraverso il lungo ciclo.
Ulteriori informazioni: Jiangtao Zhang et al, Approfondimenti strutturali sul riconoscimento e il trasporto dell'RNA a doppio filamento da parte di SID-1, Nature Structural &Molecular Biology (2024). DOI:10.1038/s41594-024-01276-9
Informazioni sul giornale: Biologia strutturale e molecolare della natura
Fornito dall'Accademia cinese delle scienze