Identificazione strutturale in vivo della proteina mitocondriale, utilizzando perossidasi e destiobiotina-fenolo. Credito:UNIST
Le proteine di membrana costituiscono circa un quarto di tutti i prodotti genetici e sono l'obiettivo di oltre il 50 percento di tutti i moderni farmaci farmaceutici. Il proteoma della membrana mitocondriale interna (IMM) svolge un ruolo centrale nel mantenimento della fisiologia mitocondriale e del metabolismo cellulare. Nonostante la loro importanza, non c'è stato alcun metodo per rivelare la topologia delle proteine della membrana mitocondriale nelle cellule vive, fino ad ora.
Uno studio recente, affiliato con UNIST ha sviluppato una nuova tecnica per comprendere le corrette architetture delle proteine IMM, utilizzando strumenti chimici speciali. Risolvendo la fase più difficile dello sviluppo di nuovi farmaci, il loro lavoro aiuterà ad accelerare lo sviluppo di nuove terapie e cure.
Questa ricerca è stata condotta dal team del Professor Hyun-Woo Rhee di Chimica presso l'UNIST in collaborazione con il Professor Jong-Seo Kim presso il Center of the Center for RNA Research, all'interno dell'Institute for Basic Science (IBS) presso la Seoul National University e il professor Jeong-Kon Seo dell'UNIST Central Research Facilities (UCRF). I risultati dello studio sono stati pubblicati nell'edizione del 15 marzo del Giornale della Società Chimica Americana ( JACS ).
L'IMM è uno dei siti più attivi per il metabolismo cellulare ed è profondamente correlato a varie malattie metaboliche umane tra cui il cancro e le malattie neurodegenerative. Perciò, è fondamentale comprendere la corretta architettura del proteoma IMM nelle cellule vive per lo sviluppo efficace ed efficiente di terapie mirate ai mitocondri.
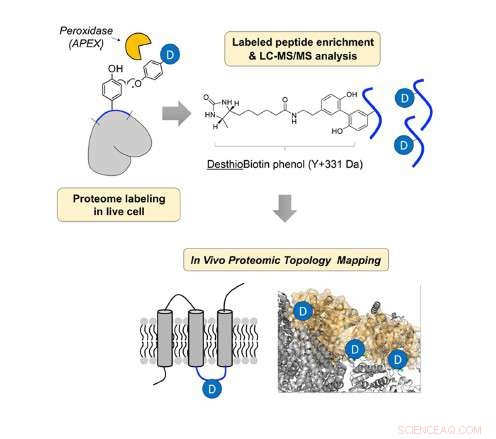
Nello studio, Il professor Lee e il suo gruppo di ricerca hanno rivelato la direzione topologica in vivo di 135 proteine IMM, utilizzando una sonda di radicali desthiobiotina-fenossile generata in situ con perossidasi geneticamente mirata (APEX).
"La determinazione della struttura delle proteine di membrana è uno dei compiti più impegnativi nell'analisi della struttura delle proteine, " afferma il professor Lee. "La nostra identificazione delle informazioni strutturali sul proteoma della membrana interna mitocondriale può fornire preziose informazioni per l'architettura e il connettoma del proteoma IMM nelle cellule vive".
Il team di ricerca ha progettato una nuova sonda chimica, destiobiotina-fenolo e lo ha applicato alle proteine IMM nelle cellule vive. Quindi, hanno identificato la struttura delle proteine di membrana tramite spettrometria di massa (MS).
La perossidasi può reagire con il perossido di idrogeno per formare il radicale fenossilico. Quindi, il radicale fenossilico può reagire con il residuo di tirosina sulla proteina prossimale formando un legame covalente. Nello studio, il team di ricerca ha ottenuto le informazioni sulla topologia analizzando il sito di tirosina marcato della proteina di membrana.
La maggior parte dell'analisi della sequenza proteica oggi utilizza la spettrometria di massa (MS), che digerisce il campione proteico in peptidi utilizzando un enzima appropriato. Analisi precedenti, che utilizzava la perossidasi di ascorbato geneticamente mirata (APEX), non è stato possibile risolvere l'identificazione strutturale perché queste analisi erano basate sul rilevamento di peptidi non etichettati. Però, solo il peptide marcato può fornire utili informazioni strutturali, secondo il gruppo di ricerca.
A differenza delle biomolecole che sono etichettate con biotina-fenolo, proteine e altri bersagli che sono etichettati con desthiobiotin-fenolo possono essere eluiti senza dure, condizioni di denaturazione. Inoltre, all'aumentare del numero di campioni di struttura proteica di membrana disponibili ottenuti tramite MS, aumenta anche l'efficienza dell'identificazione strutturale delle proteine di membrana.
A causa della breve durata dei radicali fenossilici generati in situ dall'APEX subcondriale mirato e dell'impermeabilità dell'IMM alle piccole molecole, i residui di tirosina esposti al solvente di entrambi i lati matrice e spazio intermembrana (IMS) delle proteine IMM sono stati etichettati esclusivamente con la sonda radicale in cellule vive da Matrix-APEX e IMS-APEX, rispettivamente e identificati mediante spettrometria di massa.
Attraverso questa analisi, il team di ricerca ha confermato 58 topologie di proteine IMM e ha determinato la direzione topologica di 77 proteine IMM la cui topologia all'IMM non è stata completamente caratterizzata.