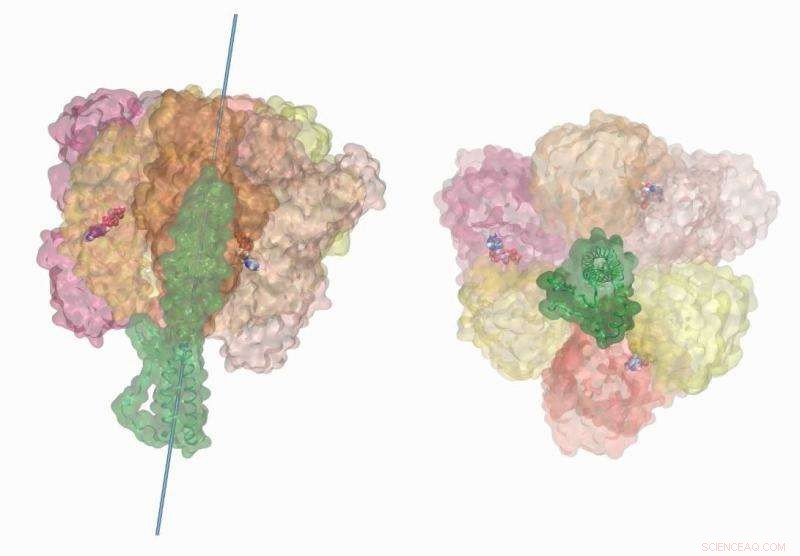
La rotazione guidata dall'idrolisi dell'ATP del gambo centrale nell'ATP sintasi di tipo V dei batteri Enterococcus hirae raffigura l'accoppiamento chemio-meccanico del motore molecolare. Queste simulazioni sono state effettuate sul supercomputer Titan utilizzando NAMD2.12. La visualizzazione e il rendering del filmato sono stati completati su VMD1.9.3. Credito:Barry Isralewitz, Università dell'Illinois a Urbana-Champaign
Nonostante la grande diversità tra gli organismi viventi, la molecola utilizzata per immagazzinare e trasmettere energia all'interno di aerobica, o che utilizzano ossigeno, cellule è notevolmente lo stesso. Dai batteri ai funghi, impianti, e animali, l'adenosina trifosfato (ATP) funge da valuta energetica universale della vita, alimentando i processi di cui le cellule hanno bisogno per sopravvivere e funzionare.
Nel corso di una giornata, un individuo userà tipicamente l'equivalente del suo peso corporeo in ATP; però, il corpo umano trasporta solo una piccola quantità della molecola alla volta. Ciò significa che le cellule devono costantemente riciclare o ricostituire la loro capacità limitata, basandosi su un motore molecolare altamente efficiente chiamato ATP sintasi per svolgere il lavoro.
Nell'ambito di un progetto dedicato alla modellazione di come i batteri unicellulari viola trasformano la luce in cibo, un team di scienziati computazionali dell'Università dell'Illinois a Urbana-Champaign (UIUC) ha simulato una sintasi di ATP completa nei dettagli di tutti gli atomi. Il lavoro si basa sulla prima fase del progetto, un organello fotosintetico da 100 milioni di atomi chiamato cromatoforo, e offre agli scienziati uno sguardo senza precedenti su una macchina biologica la cui efficienza energetica supera di gran lunga quella di qualsiasi sistema artificiale.
Proposta per la prima volta sotto la guida del compianto Klaus Schulten, pioniere nel campo della biofisica computazionale e fondatore del Theoretical and Computational Biophysics Group presso l'UIUC, la ricerca è progredita sotto la guida di Abhishek Singharoy, ricercatore co-principale e borsista post-dottorato della National Science Foundation presso il Centro per la fisica delle cellule viventi dell'UIUC.
Oltre a Singharoy, il team comprende membri dei gruppi di professori UIUC Emad Tajkhorshid, Zaida Luthey-Schulten e Aleksei Aksimentiev; ricercatore Melih Sener; e sviluppatori Barry Isralewitz, Jim Phillips, e John Stone. Al progetto ha preso parte anche il collaboratore sperimentale Neil Hunter dell'Università di Sheffield in Inghilterra.
Il team guidato dall'UIUC ha costruito e testato il suo mega-modello nell'ambito di uno stanziamento pluriennale assegnato attraverso il programma Innovative and Novel Computational Impact on Theory and Experiment sul supercomputer Titan, un Cray XK7 gestito dall'Oak Ridge Leadership Computing Facility (OLCF) del Dipartimento dell'Energia degli Stati Uniti (DOE), una struttura per gli utenti dell'Office of Science del DOE situata presso l'Oak Ridge National Laboratory del DOE.
Usando Titano, il team ha prodotto uno strumento virtuale in grado di prevedere nei minimi dettagli la produzione di energia chimica di un sistema fotosintetico in base alla quantità di luce solare assorbita. La ricerca potrebbe un giorno contribuire alla tecnologia avanzata dell'energia pulita che incorpora concetti biologici.
"La natura ha progettato il cromatoforo in modo tale da poter generare abbastanza ATP per consentire a questi batteri di sopravvivere in ambienti scarsamente illuminati come il fondo di stagni e laghi, " Singharoy ha detto. "Il nostro lavoro ha catturato questo processo di conversione dell'energia nei dettagli di tutti gli atomi e ci ha permesso di prevederne l'efficienza".
Luce in movimento
Spesso indicato come la centrale elettrica della cellula, L'ATP sintasi è un enzima complesso che accelera la sintesi dei suoi precursori molecolari, adenosina difosfato (ADP) e fosfato. Incorporato all'interno della membrana interna ed esterna del cromatoforo, il motore enzimatico è costituito da tre parti principali:un rotore a ioni, uno stelo centrale, e un anello proteico.
Simile a una ruota idraulica che viene fatta girare dalla forza di un ruscello che scorre, il rotore dell'ATP sintasi sfrutta il movimento stimolato elettrochimicamente degli ioni, come protoni o sodio, da alta concentrazione a bassa concentrazione attraverso la membrana. L'energia meccanica risultante si trasferisce allo stelo centrale, che assiste l'anello proteico nella sintesi dell'ATP.
Sorprendentemente, il processo funziona altrettanto bene al contrario. Quando troppi ioni si accumulano sul lato esterno del cromatoforo, l'anello proteico dell'ATP sintasi scompone l'ATP in ADP, un processo chiamato idrolisi, e gli ioni torneranno al lato interno.
"Normalmente, ti aspetteresti una grande perdita di energia durante questo processo, come in ogni motore artificiale, ma si scopre che l'ATP sintasi ha pochissimo spreco, " Singharoy ha detto. "Come questo motore è progettato per ridurre al minimo la perdita di energia è la domanda che abbiamo iniziato a porci".
Simile a un riparatore che smonta un motore per capire meglio come funziona, La squadra di Singharoy ha rotto i 300, enzima 000-atomo nelle sue parti costituenti. Attingendo da decenni di ricerca sull'ATP sintasi, modelli passati, e nuovi dati sperimentali forniti da un team giapponese guidato da Takeshi Murata del RIKEN Center for Life Science Technologies, il team ha costruito e simulato i pezzi del puzzle della sintasi dell'ATP indipendentemente e insieme su Titano.
Per catturare processi importanti che si svolgono su scale temporali di millisecondi, Singapore, in collaborazione con Christophe Chipot del Centro nazionale francese per la ricerca scientifica e Mahmoud Moradi dell'Università dell'Arkansas, implementato strategicamente il codice di dinamica molecolare NAMD. Il team ha eseguito una strategia d'insieme, seguendo il movimento di circa 1, 000 repliche di ATP sintasi simultanee con step temporali di 2 femtosecondi, o 2, 000 trilionesimi di secondo. In totale, il team ha accumulato 65 microsecondi (65 milionesimi di secondo) di tempo di simulazione, utilizzando queste informazioni per estrapolare i movimenti che si verificano nel corso di un millisecondo (1 millesimo di secondo).
Di conseguenza, il team ha identificato movimenti di rotazione precedentemente non documentati nell'anello proteico che aiutano a spiegare l'efficienza del motore molecolare. Allo stesso modo, le simulazioni del team hanno catturato l'elasticità simile a un elastico del gambo centrale dell'enzima. Il team di Singharoy ha stimato che quando accoppiato con l'anello proteico, il gambo assorbe circa il 75% dell'energia rilasciata durante l'idrolisi.
Inoltre, le simulazioni dell'anello proteico da solo hanno rivelato un'unità che può funzionare indipendentemente, una scoperta riportata negli esperimenti ma non nei dettagli computazionali. "Anche in assenza del gambo centrale, l'anello proteico stesso è in grado di idrolisi dell'ATP. Non è molto efficiente, ma ha la capacità, " Disse Singharoy.
La grande immagine
Dopo aver simulato il suo modello completo di ATP sintasi, il team UIUC ha incorporato l'enzima nel suo modello cromatoforo costruito in precedenza per ottenere il quadro più completo di un sistema fotosintetico fino ad oggi.
Con questo pannello solare biologico virtuale, il team ha potuto misurare ogni fase del processo di conversione dell'energia, dalla raccolta della luce, al trasferimento di elettroni e protoni, alla sintesi dell'ATP e comprenderne meglio le basi meccaniche.
Il cromatoforo della natura è progettato per l'intensità della luce scarsa, assorbendo solo tra il 3 e il 5 percento della luce solare in una giornata tipica. Il gruppo, grazie agli sforzi di Sener, scoperto che questo tasso di assorbimento si traduce in circa 300 ATP al secondo, che è la quantità di cui un batterio ha bisogno per rimanere in vita.
Dopo aver studiato il disegno della natura, il team ora voleva vedere se poteva migliorarlo. Supponendo la stessa quantità di intensità luminosa, il team ha progettato un cromatoforo artificiale con una composizione proteica decisamente innaturale, potenziando la presenza di due tipi di proteine specializzate. L'analisi del nuovo design ha previsto una triplicazione della produzione di ATP del sistema fotosintetico, aprendo la possibilità per l'ottimizzazione guidata dall'uomo del cromatoforo.
"Potresti potenzialmente modificare geneticamente un cromatoforo o cambiare la sua concentrazione di proteine, Singharoy ha detto. "Queste previsioni promettono di portare avanti nuovi sviluppi nella fotosintesi artificiale".
Nell'ambito della sua ultima allocazione INCITE, il team dell'UIUC si sta concentrando sulla conversione dell'energia in una forma di vita diversa:gli animali. Prendendo ciò che ha imparato modellando la fotosintesi nei batteri viola, il team sta modellando la respirazione cellulare, il processo utilizzato dalle cellule animali per convertire i nutrienti in ATP.
"Hai almeno due proteine in comune tra la respirazione e la fotosintesi, " disse Singharoy, che sta continuando il suo coinvolgimento nel progetto come assistente professore presso l'Arizona State University. "La domanda è quali principi di progettazione vengono trasferiti negli organismi superiori?"
La vita in situ
La simulazione del cromatoforo, completa di ATP sintasi, segna un cambiamento in corso nella biofisica computazionale dall'analisi di singole parti cellulari (ad es. singole proteine e centinaia di atomi) per analizzare interi sistemi cellulari (ad es. centinaia di proteine e milioni di atomi).
Schulten, scomparso nell'ottobre 2016, capiva meglio della maggior parte delle persone l'importanza dell'uso dei computer per simulare la natura. In un'intervista del 2015, ha esposto la sua logica per la modellazione del cromatoforo. "La motivazione è capire un passaggio fondamentale della vita sulla Terra da cui dipende tutta la vita oggi. Dal punto di vista energetico, il 95% della vita sulla Terra dipende dalla fotosintesi, compresi gli umani, " Egli ha detto.
Schulten comprese anche la pietra miliare rappresentata da un organello specializzato sulla strada per simulare un organismo unicellulare completo. "Non abbiamo niente di più piccolo di una cella che chiameremmo viva, " disse. "È il più piccolo essere vivente, e noi vogliamo capirlo".
Con i supercomputer di nuova generazione, compreso il vertice dell'OLCF, sarà online nel 2018, il gruppo di ricerca Schulten fondato nel 1989 si prepara ad affrontare la grande sfida di simulare una cellula.
Sotto la guida di Tajkhorshid, il team prevede di simulare la prima cellula da un miliardo di atomi, compresi i componenti di base di cui una cellula ha bisogno per sopravvivere e crescere. I miglioramenti alla NAMD e il lavoro svolto nell'ambito del programma Center for Accelerated Application Readiness dell'OLCF stanno contribuendo a rendere la visione di Schulten e di altri una realtà.
"Continuiamo ad andare avanti, Singharoy ha detto. "Il nostro studio esaustivo di un organello completo in tutti i dettagli atomici ha aperto la porta per una cellula piena in tutti i dettagli atomici".