La concezione di un artista del potere del design computazionale per esplorare e illuminare i peptidi strutturati attraverso il vasto panorama energetico. Credito:Vikram Mulligan/University of Washington Institute for Protein Design
Nuove strategie computazionali segnalate questa settimana in Scienza potrebbe aiutare a realizzare la promessa dei farmaci a base di peptidi. I peptidi sono simili alle molecole proteiche, ma differiscono nelle loro dimensioni più piccole, struttura e funzioni.
I peptidi macrociclici hanno suscitato l'interesse dell'industria farmaceutica, perché hanno determinate proprietà fisiche e chimiche che potrebbero diventare la base di una nuova generazione di farmaci.
I piccoli peptidi hanno i vantaggi dei farmaci a piccole molecole, come l'aspirina, e terapie con grandi anticorpi, come il rituximab, con minori inconvenienti. Sono stabili come piccole molecole e potenti e selettivi come gli anticorpi.
Un esempio di successo di un farmaco peptidico macrociclico è la ciclosporina, un immunosoppressore per i trapianti di organi e alcune malattie autoimmuni.
Prima del lavoro descritto nel Scienza carta, non c'era modo di progettare sistematicamente macrocicli peptidici ordinati come la ciclosporina.
Peptidi naturali che potrebbero servire come punti di partenza affidabili, o impalcature, sono pochi. Altrettanto frustrante è che spesso non funzionano come previsto quando vengono riproposte. Anziché, i ricercatori hanno fatto ricorso a screening di grandi dimensioni, librerie di composti generate casualmente nella speranza di trovare ciò di cui avevano bisogno.
I metodi trattati nella relazione, La "progettazione computazionale completa di macrocicli peptidici ordinati" ora risolve questi problemi.
Questa illustrazione concettuale mostra come gli eteropolimeri peptidici possono essere programmati da componenti più piccoli per esplorare un panorama energetico diversificato. Credito:Ahmad Hosseinzadeh e Khosro Khosravi
Gli autori principali sono Parisa Hossienzadeh, Gaurav Bhardwaj e Vikram Mulligan, del Dipartimento di Biochimica della School of Medicine dell'Università di Washington e dell'UW Institute of Protein Design. L'autore senior è David Baker, professore di biochimica e direttore dell'istituto. Baker è anche un investigatore dell'Howard Hughes Medical Institute.
"Nel nostro giornale, " hanno osservato i ricercatori, "descriviamo strategie computazionali per la progettazione di peptidi che adottano forme diverse con un'accuratezza molto elevata e per fornire una copertura completa delle strutture che possono essere formate da peptidi corti".
Hanno sottolineato i vantaggi di questo nuovo approccio computazionale:
Primo, sono stati in grado di progettare e compilare una libreria di molti nuovi scaffold stabili di peptidi in grado di fornire le piattaforme di base per l'architettura dei candidati farmaci. I loro metodi possono essere utilizzati anche per progettare ulteriori peptidi personalizzati con forme arbitrarie su richiesta.
"Abbiamo campionato il variegato panorama di forme che i peptidi possono formare, come guida per la progettazione della prossima generazione di farmaci, " hanno detto i ricercatori.
La chiave per il controllo della geometria e della chimica delle molecole è stata la progettazione di peptidi con amminoacidi naturali, chiamati L-amminoacidi, e i loro opposti speculari contenenti D-amminoacidi. (La L e la D stanno per parole latine per la rotazione a sinistra o a destra, come alcune strutture molecolari possono avere destrezza o chiralità).
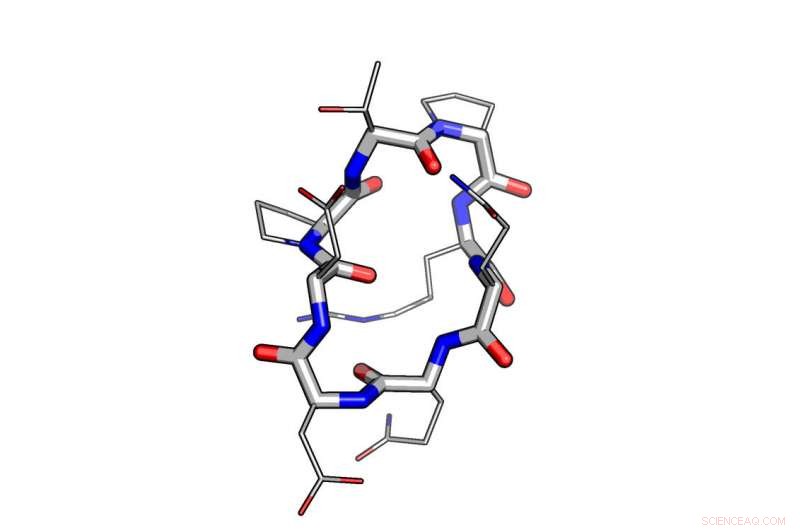
Un esempio dell'uso della progettazione computazionale nella modellazione di un peptide. Credito:University of Washington Institute for Protein Design
Gli amminoacidi D hanno migliorato le proprietà farmacologiche aumentando la resistenza agli enzimi naturali che degradano i peptidi. L'inclusione di D-amminoacidi nei design consente anche una gamma più diversificata di forme.
La progettazione di peptidi richiede un'intensa potenza del computer, con conseguenti calcoli costosi. I ricercatori hanno accreditato un gruppo di scienziati cittadini e volontari che hanno donato i loro minuti di smartphone cellulari e il tempo di utilizzo del computer. Anche il supercomputer Hyak dell'Università di Washington gestiva alcuni dei programmi.
I ricercatori hanno indicato le direzioni future per i loro approcci di progettazione computazionale dei peptidi. Sperano di progettare peptidi in grado di permeare le membrane cellulari e di entrare nelle cellule viventi.
Sotto altri aspetti, hanno in programma di aggiungere nuove funzionalità alle strutture peptidiche stabilizzando i motivi di legame alle interfacce proteina-proteina per studi scientifici di base. Per applicazioni cliniche, prevedono di utilizzare i loro metodi e scaffold per lo sviluppo di farmaci a base di peptidi.