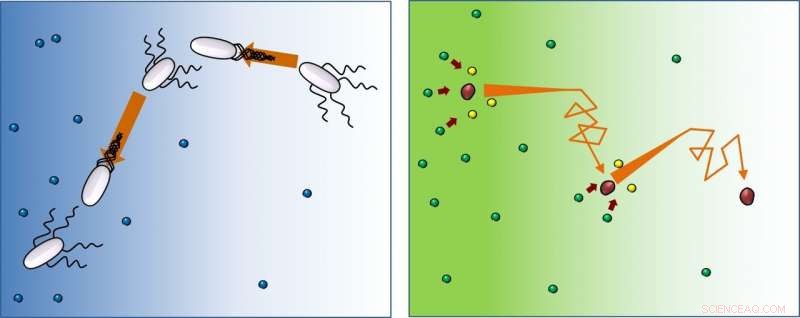
In presenza di cibo (blu), batteri (bianchi) nuotano diritti, quindi cambiare orientamento (caduta), e segui questo schema più e più volte. Questo studio mostra che gli enzimi (rossi) si muovono allo stesso modo, ma dopo aver reagito con substrati (dal verde al giallo), continuano a correre e rotolare lontano dalla maggiore concentrazione di substrati. Credito:IBS
Sebbene mettere insieme le parole "random" e "biased" possa sembrare uno scontro, questi sono gli attributi che descrivono il modo in cui i batteri si spostano e si procurano il cibo. Seguono un percorso casuale, ma sbilanciato verso la direzione della fonte dei nutrienti. Scienziati del Center for Soft and Living Matter, all'interno dell'Istituto per le Scienze di Base (IBS, Corea del Sud), osservato che gli enzimi, le molecole che aiutano le reazioni biologiche ad avvenire più rapidamente, muoversi con uno schema simile, ma nel percorso opposto, viaggiando in modo casuale verso l'area con meno substrati. Questi risultati, ottenuto con nuove tecnologie ottiche e pubblicato come articolo inaugurale in Atti dell'Accademia Nazionale delle Scienze (PNAS) , sono particolarmente intriganti in quanto comprendono un allontanamento dalla visione tradizionale.
Migliaia di enzimi promuovono una moltitudine di reazioni nel nostro corpo, ognuno dei quali sostiene la nostra vita. Gli enzimi agiscono su altre sostanze chimiche chiamate substrati e accelerano le reazioni chimiche che convertono i substrati in prodotti. Fare quello, enzimi e substrati devono incontrarsi. Originariamente si credeva che gli enzimi e i loro substrati corrispondenti si scontrassero casualmente l'uno con l'altro:"I libri di testo standard menzionano che gli enzimi e i substrati dovrebbero avvicinarsi 'in qualche modo, " interagire, ed è così che avvengono le reazioni. Abbiamo cercato di scavare nella parte "in qualche modo" di questo processo. Dopo aver raccolto migliaia di eventi, abbiamo concluso che il cammino degli enzimi è distorto in modo casuale, nel senso che sono microscopicamente direzionali, come batteri, " spiega Ah-Young Jee, primo autore di questo studio.
Il movimento dei batteri che nuotano è noto come "corri e ruzzola":corri, gira e ripeti. Procedono in una direzione, poi cambia direzione a caso, e ripetere. Però, mentre i batteri si orientano verso l'approvvigionamento alimentare, gli enzimi si muovono nella direzione di una minore concentrazione di substrato. "Le molecole mancano di capacità decisionali, ma sorprendentemente si spostano verso aree con meno substrato. Infatti, il substrato introduce la polarizzazione di direzione, e maggiore è la concentrazione di substrato che abbiamo usato nell'esperimento, più forte è questa tendenza. Ipotizziamo che possa essere un modo per rendere uniforme la concentrazione del prodotto intorno al mezzo, anche quando la concentrazione del substrato non lo è, " dice il prof. Tsvi Tlusty, che ha offerto la comprensione teorica di questa ricerca.
Poiché il passo di ciascun enzima è lungo solo circa 50 nanometri, sono necessari dispositivi estremamente precisi per misurarne la velocità e l'orientamento. Il gruppo di ricerca, guidato da Steve Granick, osservato il movimento enzimatico con una tecnologia di microscopia a super risoluzione, nota come spettroscopia di correlazione di fluorescenza a riduzione di emissione stimolata, o STED-FCS in breve.

A) Il tampone enzima-substrato (E+S+B) entra nell'ingresso sinistro, e il tampone enzimatico (E+B) viene iniettato nell'ingresso destro. In questo modo, la concentrazione dell'enzima è uniforme su tutto il chip, mentre la concentrazione del substrato è maggiore nella parte sinistra (blu) decrescendo progressivamente verso destra (bianco). Il rettangolo dei trattini indica l'area in cui il movimento dell'enzima viene studiato nei minimi dettagli utilizzando la tecnologia di microscopia STED-FCS. B) La concentrazione dell'enzima ureasi (nero) e la concentrazione di urea (il suo substrato, grigio) sono tracciati rispetto alla posizione all'interno della zona FCS, tra 0 e 2,5 millimetri dal raggio laser. La scala in ogni punto è una barra di errore, che mostra la deviazione standard su cinque misurazioni ripetute. C) Il coefficiente di diffusione (Da), che indica la velocità con cui si muove l'enzima, non cambia in assenza di substrato, ma aumenta quando il substrato è presente, il che significa che l'enzima si sta muovendo più velocemente verso destra. Credito:IBS, pubblica in PNAS
Nel metodo FCS, gli enzimi sono decorati con molecole fluorescenti e il loro passaggio attraverso un puntino viene rilevato da un raggio laser. Vengono raccolte informazioni statistiche, come il numero di enzimi che passano attraverso il punto e la loro velocità (tasso di diffusione). Il team ha integrato FCS con la tecnologia STED, uno dei punti di forza di questo centro di ricerca. STED concentra il raggio laser in un'area estremamente piccola, consentendo una misurazione più raffinata della posizione degli enzimi. Il raggio laser FCS copre un'area di circa 250 nanometri di diametro, che è circa 25 volte il diametro dell'enzima (10 nanometri). STED-FCS è più preciso poiché riduce il diametro del raggio a 50 nanometri, che è paragonabile alla distanza del passo di un enzima.
I ricercatori hanno anche progettato un nuovo chip microfluidico, formato da canali di spessore micrometrico attraverso i quali possono passare i liquidi. Studi precedenti, utilizzando FCS senza STED e un chip microfluidico diverso, suggerito che gli enzimi si spostano verso l'area con la più alta concentrazione di substrato. Però, questa nuova tecnologia permette osservazioni più profonde e mostra il contrario. Gli autori pensano che i risultati differiscano perché le informazioni dettagliate sul movimento dell'enzima sono state cancellate usando un raggio laser più grande. Sarebbe simile a localizzare qualcuno entro 50 chilometri quadrati o entro 2 chilometri quadrati, quest'ultimo è più preciso. Hanno creato condizioni in cui l'enzima è distribuito uniformemente all'interno del chip, ma la concentrazione del substrato varia da sinistra a destra:da una concentrazione minima sul lato destro del chip, a una concentrazione massima a sinistra. Quindi, hanno usato FCS per osservare la concentrazione e la velocità dell'enzima all'interno del chip, e ha scoperto che l'enzima tende ad accelerare verso l'area con minore concentrazione di substrato (cioè verso il lato destro del chip). Anziché, quando non è presente alcun substrato, gli enzimi non cambiano la loro velocità. Inoltre, le capacità di super-risoluzione di STED-FCS hanno mostrato dinamiche run-and-tumble.
Il team ha osservato lo stesso comportamento separatamente con due coppie enzima-substrato:ureasi-urea e acetilcolinesterasi (AChE)-acetilcolina. Il primo enzima viene utilizzato da alcuni microrganismi per trasformare l'urea in ammoniaca e acido carbonico, quest'ultimo svolge un ruolo essenziale nelle giunzioni neuromuscolari.
Nel futuro, il team di ricerca mira ad espandere la complessità dello studio e testare condizioni più simili alla vita reale:"Questo studio ha esaminato il comportamento di un enzima in risposta a un substrato. Successivamente, abbiamo in programma di esaminare diversi enzimi contemporaneamente. Il nostro corpo funziona con una cascata di reazioni, il che significa che una reazione innesca la successiva:l'enzima A innesca l'enzima B, quindi B innesca C, e così via. L'analisi STED-FCS potrebbe essere uno strumento chiave per far progredire la ricerca metabolica rivelando come questi molteplici sistemi enzimatici siano organicamente correlati, "conclude Jee.