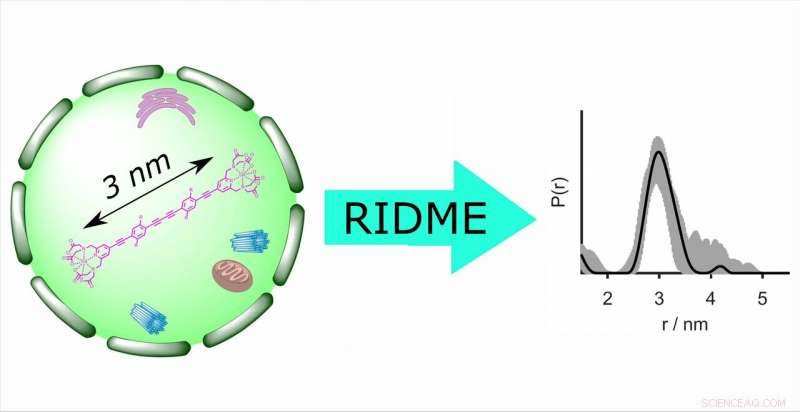
La determinazione della distanza nelle cellule mediante EPR rivela informazioni strutturali essenziali sulle biomacromolecole in condizioni native. Per la prima volta, la tecnica EPR pulsata RIDME (rilassamento indotto dipolar modulation enhancement) è stata utilizzata per misure di distanza all'interno delle cellule. Fornisce una sensibilità cinque volte migliore rispetto all'approccio della doppia risonanza elettrone-elettrone precedentemente utilizzato. Credito:gruppo di ricerca Professor Malte Drescher, Università di Costanza
In un documento congiunto, ricercatori dell'Università di Costanza, L'Università di Bielefeld e l'ETH di Zurigo dimostrano per la prima volta che la tecnica di risonanza paramagnetica elettronica (EPR) RIDME (miglioramento della modulazione dipolare indotto dal rilassamento) può essere applicata per determinare le distanze tra le etichette di spin a base di gadolinio (III) nelle cellule. La determinazione della distanza nelle cellule mediante risonanza paramagnetica elettronica (EPR) rivela informazioni strutturali essenziali sulle biomacromolecole, compresa la loro conformazione così come i processi di piegatura e spiegamento.
I metodi convenzionali per la determinazione delle distanze all'interno della cella come la doppia risonanza elettrone-elettrone (DEER o PELDOR) sono principalmente molto meno sensibili di RIDME, fornire profondità di modulazione fino a cinque volte inferiori, presentano alcune limitazioni per quanto riguarda la larghezza di banda di eccitazione e sono tecnicamente più esigenti. Come tecnica a frequenza singola che fa uso di spin flip indotti dal rilassamento per determinare la distanza tra due etichette di spin, cioè tra due elettroni spaiati, RIDME supera tutti questi svantaggi.
Questa tecnica consente ai ricercatori di lavorare con molecole in condizioni native, come il professor Malte Drescher e l'autore principale Dr Mykhailo Azarkh, entrambi dall'Università di Costanza, sottolineare:"Siamo partiti dall'analisi della conformazione di una proteina all'interno della cellula. Con tecniche meno sensibili, siamo costretti ad inserire e taggare molte proteine per poterlo osservare, che non è affatto ciò che accade in natura. Idealmente, vogliamo lavorare con concentrazioni che sono fisiologicamente rilevanti. Poiché RIDME è molto più sensibile di DEER, ci permette di fare proprio questo. Ora siamo in grado di affrontare questioni che altrimenti non saremmo in grado di affrontare".
Le prestazioni di RIDME nelle cellule sono state valutate in banda Q utilizzando righelli molecolari rigidi etichettati con Gd(III)-PyMTA e microarticolati in ovociti di Xenopus laevis (rana artigliata africana). In altre parole, i ricercatori hanno utilizzato un sistema modello in cui la distanza precisa tra le etichette di spin era già nota, consentendo loro di verificare le misurazioni RIDME. Il documento risultante intitolato "Gd(III)–Gd(III) Relaxation-Induced Dipolar Modulation Enhancement for In-Cell Electron Paramagnetic Resonance Distance Determination" è stato pubblicato online nel Journal of Physical Chemistry Letters il 13.03.2019.
La determinazione della distanza RIDME in cella è stata sviluppata e testata come parte del progetto in corso finanziato dall'ERC "SPICE – Spectroscopy in cells", per cui Malte Drescher, Heisenberg Professore di Spettroscopia di Sistemi Complessi presso l'Università di Costanza, e il suo team di ricerca hanno ricevuto un ERC Consolidator Grant del valore di circa due milioni di euro nel 2017. Il loro obiettivo è sviluppare nuovi approcci alla spettroscopia che consentano loro di esplorare strutture biologiche più grandi e complesse a livello molecolare della cellula.
Il prossimo passo in questa linea di ricerca sarà identificare altre etichette di spin adatte e sviluppare RIDME per l'applicazione in molecole in cui la distanza tra le etichette di spin è sconosciuta. Particolare attenzione sarà dedicata alle molecole associate a malattie neurodegenerative come l'Alzheimer e il Parkinson.