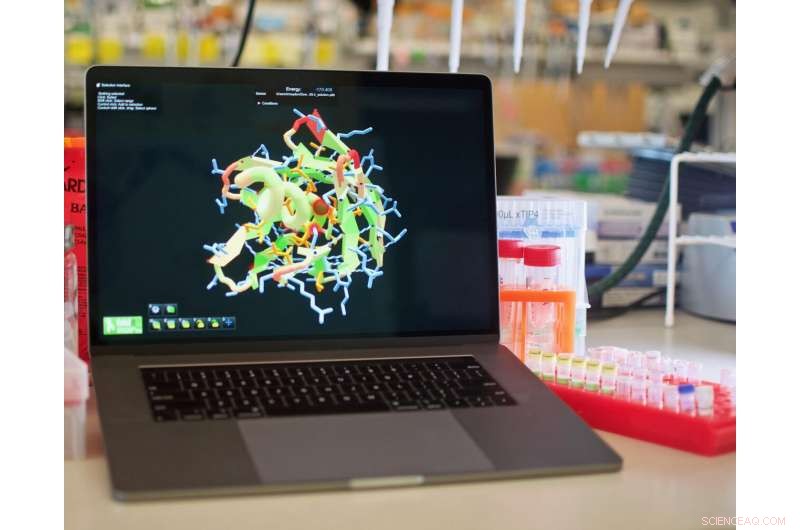
Giocando al videogioco Foldit, gli appassionati di puzzle ora possono progettare proteine che non sono mai esistite prima, e aiutare nella creazione di nuovi vaccini e trattamenti per il cancro e altre malattie gravi. Credito:Institute for Protein Design/UW Medicine
Un team di ricercatori ha codificato le proprie conoscenze specialistiche nel gioco per computer Foldit per consentire agli scienziati cittadini di progettare con successo proteine sintetiche per la prima volta.
I primi risultati di questa collaborazione appaiono nel numero del 5 giugno di Natura . L'Institute for Protein Design presso la School of Medicine dell'Università di Washington ha guidato lo sforzo multi-istituzionale.
"Ci sono più proteine possibili che atomi nell'universo. È emozionante pensare che ora chiunque possa aiutare a esplorare questo vasto spazio di possibilità, ", ha affermato il coautore senior David Baker, professore di biochimica alla UW School of Medicine e direttore dell'Institute for Protein Design.
"La diversità di molecole che questi giocatori hanno creato è sorprendente, ", ha affermato l'autore principale Brian Koepnick, ricercatore post-dottorato presso l'Institute for Protein Design. "Queste nuove proteine non sono affatto inferiori a quelle che potrebbe fare uno scienziato con un dottorato di ricerca".
Foldit è stato creato nel 2008 come un modo per "gamificare" la ricerca sulle proteine. Le proteine sono biomolecole essenziali che si trovano all'interno di ogni cellula di ogni organismo. Le loro intricate strutture tridimensionali danno origine alle loro diverse funzioni, che includono la digestione, guarigione delle ferite, autoimmunità e molto altro.
Attraverso il gioco, I giocatori di Foldit hanno contribuito a determinare la struttura di una proteina correlata all'HIV e hanno migliorato l'attività di enzimi utili. Fino ad ora, però, I giocatori di Foldit potevano interagire solo con proteine già esistenti. Non c'era modo di progettarne di nuovi.
"Progettare proteine completamente nuove che non esistevano in natura è stato il nostro obiettivo con Foldit per molto tempo, ", ha affermato il coautore senior Seth Cooper, assistente professore al Khoury College of Computer Sciences della Northeastern University. "Questa nuova serie di risultati mostra che è possibile".
Per trasformare Foldit in una piattaforma per la progettazione di proteine, i ricercatori hanno codificato la conoscenza biochimica nel gioco. Quel modo, le molecole di design che hanno ottenuto buoni risultati in Foldit avrebbero maggiori probabilità di piegarsi come previsto nel mondo reale.
"Non abbiamo tenuto lezioni [ai giocatori di Foldit] né abbiamo detto loro di leggere qualcosa. Invece, abbiamo ottimizzato il codice che ha eseguito il gioco per molti anni, ", ha affermato il coautore senior Firas Khatib, assistente professore di informatica presso l'Università del Massachusetts Dartmouth.
Gli scienziati hanno testato in laboratorio 146 proteine progettate dai giocatori di Foldit. 56 sono risultati stabili. Questa scoperta ha suggerito che i giocatori avevano prodotto alcune proteine realistiche. I ricercatori hanno raccolto dati sufficienti su quattro di queste nuove molecole per dimostrare che i progetti hanno adottato le strutture previste.
"Non avrei mai creduto che sarebbero diventati così buoni, ma i giocatori di Foldit non smettono mai di stupirci." ha detto Khatib.
Creare nuove proteine è un po' come cercare di fare nodi mai visti prima usando una corda che è un milione di volte più sottile di un capello umano. Ad oggi, solo un piccolo gruppo di esperti con una profonda conoscenza del modo in cui le biomolecole si contorcono e girano si preoccupano di questo compito estremamente complesso. La maggior parte utilizza algoritmi di progettazione molecolare automatizzata, e la maggior parte degli algoritmi di progettazione fallisce molto più spesso di quanto riesca.
"Cerchiamo sempre di migliorare gli algoritmi, ma l'elemento umano è fondamentale, " disse Khatib. "In effetti, attraverso il design Foldit, i giocatori hanno persino scoperto difetti nella funzione energetica di Rosetta, il nostro metodo all'avanguardia per la progettazione delle proteine".
Il design delle proteine è una disciplina scientifica emergente. Negli ultimi cinque anni, gli esperti dell'Institute for Protein Design e i loro colleghi hanno creato proteine che stimolano il sistema immunitario a combattere il cancro e altre che agiscono come potenti candidati al vaccino. In Aprile, l'Institute for Protein Design ha ricevuto un impegno di 45 milioni di dollari in finanziamenti attraverso The Audacious Project, una collaborazione filantropica organizzata da TED, progettare vaccini a base di proteine, medicinali e materiali.
I giocatori potrebbero creare il prossimo farmaco campione d'incassi?
"I giocatori Foldit sono una nuova aggiunta all'arsenale di ricerca, " ha detto Khatib. "Non sono un proiettile d'argento, ma sono una risorsa straordinaria".
Chiunque sia interessato ad aiutare a risolvere enigmi per la scienza può saperne di più su fold.it/portal/