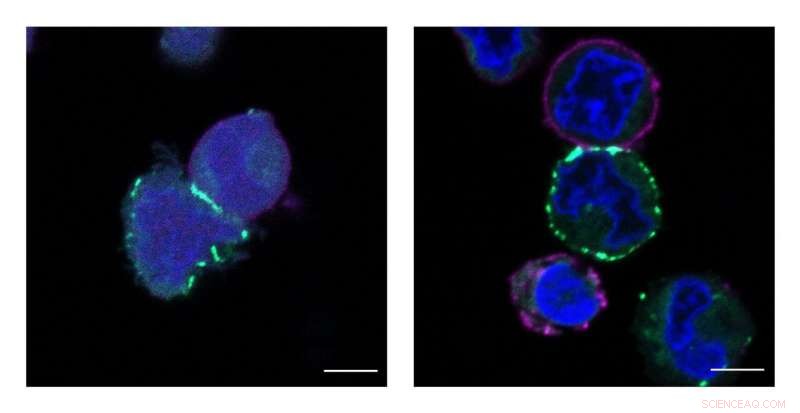
Il MacMillan Group di Princeton ha sviluppato una nuova tecnologia chiamata μMap che identifica i vicini più prossimi delle proteine sulle superfici cellulari. Queste immagini mostrano le cellule etichettate, o evidenziato in verde, dal catalizzatore μMap. Credito:Tamara Reyes-Robles/Merck Exploratory Science Center
Quando due anni fa i partner aziendali della Princeton Catalysis Initiative si sono incontrati con David MacMillan, gli hanno presentato una sfida biologica al centro di potenziali farmaci antitumorali e altre terapie:quali proteine sulla superficie di una cellula si toccano?
Quello che volevano era analogo a un riflettore che arde in una caverna buia, qualcosa per illuminare una luce metaforica su una proteina e sui suoi vicini più vicini sulla membrana cellulare. Grande, molecole complesse, le proteine sono la sostanza della vita, il vero fulcro su cui ruota tutto di noi:il modo in cui pensiamo, il modo in cui cresciamo, le malattie che riceviamo. Le proteine sono in grado di determinarlo inviando messaggi ai loro vicini. Ma mentre gli scienziati potevano dire in precedenza chi c'era all'interno della grotta, non potevano dire chi stava in piedi accanto a chi, e quindi mancavano importanti conoscenze su queste comunicazioni essenziali da proteina a proteina.
Il MacMillan Group del Dipartimento di Chimica ha annunciato nell'attuale numero di Scienza che hanno sviluppato quel proiettore.
La tecnologia rivoluzionaria, chiamato μMap dal team di ricercatori di Princeton e scienziati Merck, utilizza un fotocatalizzatore, una molecola che, quando attivato dalla luce, stimola una reazione chimica, per identificare le relazioni spaziali sulle superfici cellulari. Il catalizzatore genera un marcatore che etichetta le proteine e i loro vicini molecolari, che a sua volta consente la mappatura precisa del loro microambiente.
La tecnologia potrebbe avere un impatto sulla proteomica, genomica e neuroscienze, per citare alcuni dei campi più ovvi. Ma le applicazioni per la biologia fondamentale sono così vaste che MacMillan, chi è James S. McDonnell Distinguished Professor di Chimica della Princeton, ha fame di mettere la tecnologia "nelle mani di tutti" per vedere cosa possono inventare gli scienziati in altri campi.
"Per le tecnologie che abbiamo in questo momento, il problema non è se puoi taggare le cose, " ha detto. "Il problema è che puoi taggare migliaia di cose e quindi non puoi dire cosa c'è laggiù e cosa c'è proprio accanto. Questo risulta essere davvero, molto importante perché le molecole o le proteine o gli enzimi che si segnalano a vicenda sono di solito proprio accanto l'una all'altra. Bene, lo stato dell'arte non ti dice cosa c'è vicino."
Così hanno escogitato un approccio radicalmente nuovo.
"Abbiamo fatto alcuni esperimenti critici e immediatamente abbiamo potuto dimostrare che stavamo etichettando le cose a una distanza davvero breve, " ha detto MacMillan. "Ora sappiamo esattamente cosa c'è nel quartiere. E questo non è mai stato fatto prima. Per la biologia, sarà come accendere l'interruttore della luce e vedere improvvisamente tutto".
Gli scienziati del Merck Exploratory Science Center (MESC) Rob Oslund e Olugbeminiyi Fadeyi, coautori di articoli con sede a Cambridge, Massachusetts, ha detto che la tecnologia potrebbe ispirare vasti nuovi sviluppi in biologia. "Dato l'importante ruolo della comprensione delle interazioni proteiche all'interno dei micro-ambienti cellulari, " Oslund ha detto, "questa tecnologia ha il potenziale per essere uno strumento rivoluzionario per i laboratori di scienze biologiche sia accademici che industriali in tutto il mondo".
La μMap, micromappa pronunciata, identifica i vicini entro un raggio da 1 a 10 nanometri attorno a una particolare proteina. (Per riferimento, un capello umano è circa 100, 000 nanometri di diametro.) La risoluzione su questo livello identifica le 10 o 15 molecole più vicine.
Giacobbe Geri, un ricercatore post-dottorato presso il Merck Center for Catalysis presso la Princeton University e co-primo autore sulla Scienza documento con lo studente laureato James Oakley e la scienziata MESC Tamara Reyes-Robles, detto μMap fa questo usando la luce blu per alimentare una reazione catalitica.
Ecco come funziona:il catalizzatore, in questo caso, un composto metallico organico, è selettivamente attaccato a uno qualsiasi dei circa 40, 000 proteine sulla superficie di una cellula, dove agisce come una specie di antenna. Luce blu, che ha un'energia fotonica molto alta, funge da innesco. Quando brillava sul cellulare, questa luce blu viene captata dall'antenna, che converte la sua energia fotonica in energia chimica. Quell'energia latente non si raffredda; non si diffonde; non vaga senza meta lungo la membrana cellulare dipingendo tutto ciò che incontra. Si siede e basta.
Sulla base di un documento pubblicato circa 40 anni fa, Il gruppo di MacMillan ha avuto l'idea di impiegare l'uso di una molecola organica chiamata diazirina che è particolarmente ricettiva a questa energia latente. Quando una diazirina si avvicina molto al catalizzatore, entro 0,1 nanometri, l'energia chimica si trasferisce alla diazirina. La diazirina a sua volta reagisce così violentemente da rilasciare un sottoprodotto e diventa ciò che viene definito carbene, una specie "arrabbiata" che si attacca alle proteine vicine.
"Il catalizzatore trasferisce così tanta energia che la molecola si fa a pezzi per esporre un atomo di carbonio incredibilmente instabile, che poi si attaccherà a tutto ciò che può, " ha spiegato Geri.
Il catalizzatore può eseguire questa reazione chimica molte volte, quindi il processo si ripete per tutte le molecole localizzate, proteine ed enzimi. Poiché i carbeni hanno una vita così breve, solo un paio di nanosecondi, la loro reazione fornisce un vivido, snapshot in tempo reale di tutte le molecole contigue. Successivamente, i ricercatori possono mettere insieme una mappa precisa del microambiente, proprio la tecnologia che gli scienziati stavano cercando.
"Gran parte del meccanismo della malattia avviene attraverso il modo in cui queste cellule parlano tra loro, e possono parlare solo se si toccano, " ha detto Geri. "Ecco perché la superficie della cellula è così importante. Se si toccano, possono comunicare».
Ha aggiunto:"Ora possiamo capire cosa sta facendo accadere quella comunicazione o cosa sta facendo cambiare quella comunicazione. È stata davvero un'esperienza straordinaria, lavorando su questo».
MacMillan's group chose two categories of human cells to investigate. One was a class of proteins that had known interactions, selected as a kind of control group to prove that their interactions could be captured by μMap. The second group was "more interesting, " said Geri. It centered on proteins called PD-L1 and PD-1, which are associated with the body's immune system and its response to cancer cells.
Normalmente, sick cells like cancer cells would present as molecular interlopers that need to be cleared by the immune system. But cancer cells are deceptive, said MacMillan. They send out a "don't kill me" signal through a cloaking mechanism involving the PD-L1 and PD-1 axis. Since cancer therapies are successful partly based on their capacity to block that signal, scientists want to know more about how it is transmitted. Mapping the precise neighborhood is an essential early step. When researchers put the μMap catalyst on PD-L1 and PD-1, the molecules in their micro-environment are tagged. Protein-protein interactions that had previously been hypothesized could now be directly observed. And several correlations were detected that had never been conceived of.
"Ora, we don't do the cancer biology, " said MacMillan. "But we've invented this tool that can give you a lot of information about these cancer cells. We think that by using this information, you can start to target those proteins as a way to also remove interfering signals. And if you can remove those signals, you make your immune system better at going after these cancer cells."
Soon after MacMillan arrived at Princeton, he began driving research in harnessing blue LED light to perform previously impossible feats of chemistry. Merck became involved in 2006, with a seed donation toward MacMillan's research. The company has since donated additional monies, and in 2019 they announced a 10-year funding commitment towards the Princeton Catalysis Initiative, which fosters interdisciplinary collaborations to accelerate the discovery of new research areas.
"Our collaboration created a novel cellular chemistry approach leveraging photoredox catalysis to activate diazirines, an important class of organic molecules, in a temporal-controlled manner, " said Merck's Fadeyi. "Because of the routine use of diazirines within chemical biology and biology, this method will be in high demand not only for protein labeling, but for identifying the binding targets of other biomolecules to elucidate their functional roles."
He added:"The collaboration was successful due to the close interactions between Merck scientists and Dave's lab."
MacMillan likewise lauded the discovery as proof of the value of collaboration across academic and industry lines, like those envisioned when the Princeton Catalysis Initiative first came into being in 2018.
"Come chimici, we don't know any good questions in biology—zero, " disse. "Allora, you're taking these people who know everything about biology, and they have this problem that they're trying to solve. And it's ultimately a great problem for a chemistry group. Allo stesso tempo, it's not a problem that a chemistry group would ever think about because they don't know biology. You have these two different areas and you put them together and you start to realize there all these great things you can do.
"This is what I love about the social science of science, " he added. "It is absolutely a beautiful example of how it took a village to solve a problem."