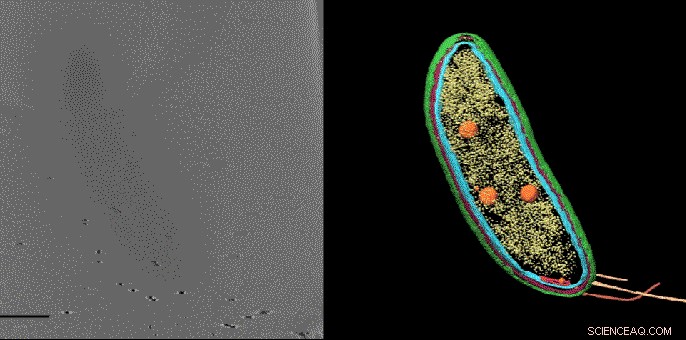
Gli scienziati della Stanford University pensavano che un'area apparentemente vuota a un'estremità del batterio Caulobacter potesse contenere due proteine coinvolte nella divisione cellulare. Etichettando le proteine con tag fluorescenti e quindi visualizzando quegli stessi campioni con la tomografia crioelettronica, sono stati in grado di confermare questa posizione e mostrare esattamente come erano disposte le proteine. Credito:Peter Dahlberg et al., PNAS , 8 giugno 2020
Gli scienziati hanno sposato due delle tecniche di microscopia più potenti di oggi per creare immagini che individuano, per la prima volta, le identità e le posizioni precise delle singole proteine nel contesto dettagliato delle cellule batteriche. Queste informazioni sono fondamentali per apprendere come le molecole proteiche lavorano insieme per organizzare la divisione cellulare e svolgere altri compiti importanti, come consentire ai microbi di fiutare il cibo e il pericolo.
Il nuovo metodo ha già portato alla luce nuove informazioni sulle proteine batteriche e sui loro quartieri cellulari vicini. I ricercatori affermano che ha anche il potenziale per rispondere a domande fondamentali sul meccanismo molecolare dei virus, parassiti, e processi come la fotosintesi.
"Questo è un grande passo avanti per la biologia, e credo che ce ne siano tanti, molti sistemi che beneficeranno di questo tipo di imaging, " ha detto la professoressa di Stanford Lucy Shapiro, il cui gruppo di ricerca ha partecipato allo studio.
Il nuovo metodo ibrido, chiamato imaging correlato mediante annotazione con singole molecole, o CIASM (pronunciato "baratro"), è stato sviluppato da Peter Dahlberg, un ricercatore post-dottorato nel laboratorio del professor W. E. Moerner alla Stanford University.
È una variazione di una tecnica chiamata microscopia a singola molecola a bassa temperatura, inventato da Moerner tre decenni fa, che attacca etichette luminose alle molecole in modo che possano essere identificate individualmente. Questo metodo è alla base della microscopia a fluorescenza a super risoluzione, il tema del Premio Nobel per la Chimica 2014 di Moerner.
Quello che ha fatto Dahlberg è stato trovare un modo per far funzionare questo tipo di imaging a fluorescenza a temperature inferiori allo zero in modo che gli stessi campioni potessero essere esaminati anche con la tomografia elettronica criogenica (CET). CET utilizza flussi di elettroni per creare immagini 3D di cellule congelate in flash e dei loro componenti con una risoluzione quasi atomica. La combinazione di CET con l'imaging fluorescente consente agli scienziati di vedere le molecole etichettate nel contesto della cellula circostante, una prospettiva cruciale per comprendere il loro ruolo nel macchinario cellulare.
"Possiamo etichettare specifiche molecole di interesse in modo che la luce che vediamo provenga solo da quelle molecole, e poi troviamo dove sono entro circa 10 nanometri, o miliardesimi di metro. Questo ci dà un'immagine molto più accurata di quello che sta succedendo, " Ha detto Dahlberg. "Abbiamo preso le istantanee ultra precise fornite da CET e abbiamo aggiunto un po' di colore".
Ha aggiunto, "È entusiasmante sviluppare nuovi metodi di imaging. Quando hai finito, puoi fare un passo indietro e guardare tutte le nuove domande che puoi attaccare."
Con CIASM, il team di ricerca è stato in grado di individuare le posizioni di tre tipi di proteine nelle immagini CET ad alta risoluzione di batteri scattate presso il National Accelerator Laboratory SLAC del Dipartimento dell'Energia. I risultati sono stati riportati nel Atti dell'Accademia Nazionale delle Scienze oggi.
"Ogni metodo ha i suoi vantaggi e svantaggi, "Moerner ha detto, "e questa è una bella situazione in cui possiamo combinare due metodi per saperne di più".
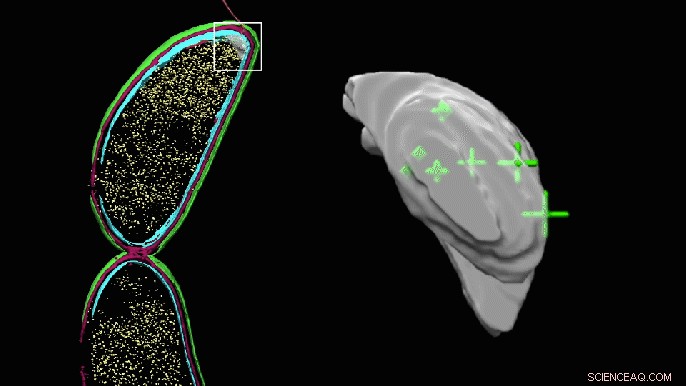
Un'immagine 3D rotante della tasca apparentemente vuota a un'estremità di una cellula di Caulobacter ora mostra le posizioni precise delle molecole PopZ. La tasca sembra grumosa perché è stata colorata manualmente per evidenziare l'area in cui i ricercatori pensavano che potessero essere le molecole, ma non è stato possibile identificare direttamente nella tomografia crioelettronica. Credito:Peter Dahlberg et al., PNAS , 8 giugno 2020
Trovare l'ordine in una zuppa cellulare
Anche in cellule batteriche relativamente semplici, la posizione è tutto, disse Saumya Saurabh, un ricercatore post-dottorato nel laboratorio di Shapiro che ha svolto un ruolo di primo piano nella ricerca.
"Le persone tendono a pensare ai batteri come a sacchi di proteine senza organizzazione, " ha detto. "Ma si scopre che non è vero, e infatti molte delle molecole nei batteri sono precisamente localizzate sia nello spazio che nel tempo. Se non sono nella posizione giusta, la cellula muore. Quello che il lavoro di Pete ci permette finalmente di fare è guardare dentro con risoluzione molecolare e scoprire quando e dove si trovano queste molecole l'una rispetto all'altra".
Caulobacter crescentus, ad esempio, una specie ben studiata di batteri d'acqua dolce, è noto per dividersi in due tipi molto diversi di cellule figlie:una nuota liberamente, mentre l'altro forma uno stelo e si attacca a una superficie. Il modo in cui ogni cellula figlia ottiene ciò di cui ha bisogno per seguire il suo percorso unico è un mistero di vecchia data.
Gli scienziati avevano precedentemente identificato piccole aree alle due estremità della cellula in divisione che potrebbero contenere proteine che svolgono ruoli chiave in questa divisione cellulare asimmetrica. Una delle proteine, PopZ, si trova ad entrambe le estremità della cellula divisoria, mentre l'altro, SpmX ("Spam-X") si trova solo nella metà che svilupperà un gambo.
Per questo studio, Saurabh e lo studente laureato Jiarui Wang hanno etichettato le proteine in Caulobacter con etichette fluorescenti. Poi Dahlberg ha congelato questi campioni, ha eseguito su di essi l'imaging a fluorescenza a singola molecola con l'aiuto della studentessa laureata Annina Sartor, e li ha portati alle strutture Cryo-EM di Stanford-SLAC per l'imaging CET diretto da Wah Chiu, un professore a Stanford e SLAC.
Mappare un ritrovo proteico
Le immagini combinate non solo hanno confermato che entrambe le proteine erano nelle aree sospettate dagli scienziati, ma ha anche rivelato esattamente come erano disposti:SpmX era incorporato nella membrana interna della cellula e sporgeva all'interno della cellula, dove è entrato in contatto diretto con PopZ.
"L'esatto orientamento di questo complesso proteico è stato discusso negli ultimi 12 anni, "Sarabh ha detto. "Siamo stati in grado di osservare i partner proteici con una risoluzione squisita. Ora abbiamo un quadro molto preciso di come queste proteine comunicano tra loro nella cellula".
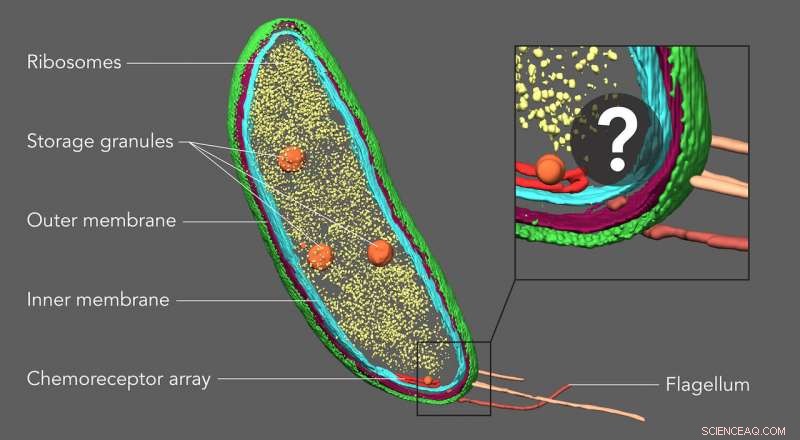
Con una tecnica chiamata tomografia crioelettronica, gli scienziati possono creare immagini 3D dettagliate delle cellule, come questo batterio Caulobacter, ed evidenziare i loro componenti - in questo caso, le membrane cellulari (rosso e blu), guscio proteico (verde), fabbriche di proteine note come ribosomi (giallo) e granuli di stoccaggio (arancione). Ma fino ad ora, strutture più piccole e singole molecole non possono essere identificate e localizzate con precisione all'interno di queste immagini. Una nuova tecnica di imaging sviluppata a Stanford colma questa lacuna, rivelando piccole molecole che non sono visibili qui. Credito:Peter Dahlberg et al., PNAS , 8 giugno 2020
Il team ha testato l'accuratezza del CIASM utilizzandolo per confermare la posizione di una proteina chiamata McpA che era nota per essere parte di una serie di chemocettori nei batteri. "Le proteine squisitamente sensibili in questa matrice fungono da naso di Caulobacter, "Sarabh ha detto, "percependo la chimica dell'ambiente circostante in modo che possano allontanarsi da cose spiacevoli e avvicinarsi al glucosio che mangiano".
L'array appare come linee nere parallele nelle immagini CET, e l'etichettatura fluorescente delle stesse immagini ha individuato le posizioni delle singole proteine McpA entro circa 10 nanometri.
Uno sguardo dettagliato ai punti quantici
In un separato, studio parallelo, pubblicato il 24 aprile in Angewandte Chemie , i ricercatori hanno utilizzato una tecnica simile per esaminare singoli punti quantici, con alcuni risultati sorprendenti.
I punti quantici sono cristalli su scala nanometrica di materiale semiconduttore che emettono naturalmente fluorescenza in colori determinati dalla loro dimensione, forma e composizione. Questi punti vengono utilizzati nella ricerca per etichettare e tracciare proteine e altri materiali biologici, e hanno potenziali applicazioni nell'elettronica futura, illuminazione, informatica quantistica, imaging medico e altre aree.
In questo studio, l'obiettivo era vedere come i dettagli strutturali più fini dei singoli punti fossero correlati a dettagli specifici delle loro proprietà ottiche, disse Davis Perez, un dottorato di ricerca studente nel laboratorio di Moerner.
"Siamo stati in grado di vedere alcuni comportamenti sorprendenti dei singoli punti quantici, ad esempio, nella loro risposta all'eccitazione con luce laser, " ha detto. "Ma l'aspetto più eccitante per me è che il metodo che abbiamo sviluppato per studiare i punti quantici può essere utilizzato anche per studiare sistemi biologici come le proteine fotosintetiche, dove l'energia viene trasferita tra gruppi di proteine, e vedere come funziona il macchinario fotosintetico."
Moerner ha detto che il suo laboratorio sta lavorando con Chiu per perseguire queste sfide.
"Sono i primi giorni di combinazione dei due metodi, e siamo entusiasti di esplorare più collaborazioni che collegano luce ed elettroni, " Ha detto Chiu. "Questo approccio di imaging ibrido ha il potenziale per scoprire strutture di componenti molecolari coinvolti in processi biologici chiave nelle cellule che abbracciano tutti i domini della vita".