Codifica elettrochimica delle basi del DNA. Crediti:Istituto di Chimica Organica e Biochimica del CAS
Un team di ricerca internazionale guidato da Michal Hocek dell'Istituto di chimica organica e biochimica dell'Accademia ceca delle scienze (IOCB Praga) e della Charles University e Ciara K. O'Sullivan dell'Università Rovira i Virgili (URV) in Spagna hanno sviluppato un romanzo metodo per etichettare il DNA, che in futuro potrà essere utilizzato per il sequenziamento del DNA mediante rivelazione elettrochimica. I ricercatori hanno presentato i loro risultati nel Giornale della Società Chimica Americana .
Una molecola di DNA comprende quattro elementi costitutivi di base, nucleotidi. L'informazione genetica trasportata all'interno della molecola è determinata dall'ordine dei nucleotidi. Conoscenza dell'ordine di questi elementi costitutivi, nota come sequenza del DNA, è necessario per la diagnostica delle malattie e l'analisi forense del DNA, Per esempio. Nonostante i grandi progressi degli ultimi anni, gli attuali metodi di sequenziamento del DNA, tipicamente basato su etichettatura fluorescente, sono ancora tecniche lunghe e relativamente costose, che hanno alcune limitazioni. Perciò, gli scienziati sono alla ricerca intensiva di nuovi approcci per semplificare e accelerare il sequenziamento.
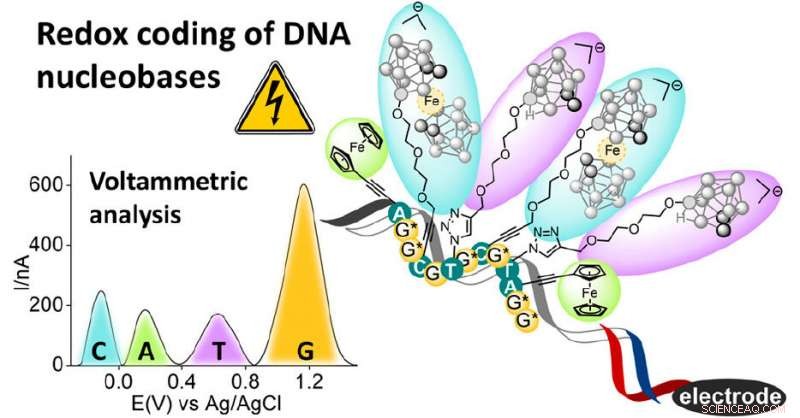
Un approccio promettente è l'uso del rilevamento elettrochimico e delle cosiddette etichette redox, che sono composti che possono essere ossidati o ridotti sugli elettrodi. Un team multidisciplinare di ricercatori dell'IOCB Praga, URV, la Facoltà di Scienze della Charles University, l'Accademia polacca delle scienze, e l'Istituto di biofisica dell'Accademia ceca delle scienze, con gli studenti David Kodr e Cansu Pinar Yenice come primi autori, è ora riuscita a progettare e sintetizzare nucleotidi artificiali con speciali etichette redox attaccate che possono essere ossidate su un elettrodo d'oro o di carbonio a un potenziale specifico per produrre un segnale misurabile e analiticamente utile. Queste etichette sono carborani, strutture a gabbia composte da boro e atomi di carbonio, in cui possono essere incorporati altri atomi di metallo, come ferro o cobalto, influenzando le loro proprietà elettrochimiche risultanti.

Rappresentazione artistica della codifica elettrochimica delle basi del DNA. Credito:Tomáš Belloň / IOCB Praga
I nucleotidi modificati sono stati ingegnerizzati in modo che l'enzima DNA polimerasi, che utilizza i blocchi costitutivi dei nucleotidi disponibili per costruire il DNA all'interno di una cellula, possono incorporarli in un filamento di DNA di nuova sintesi. Così, i ricercatori sono riusciti a preparare un filamento di DNA comprendente nucleotidi modificati. Allo stesso tempo, ciascuno dei quattro tipi di nucleotide porta la propria etichetta univoca che consente la sua successiva identificazione. E qui stava la trappola principale del progetto; fino ad ora, i ricercatori erano sempre riusciti solo a etichettare e misurare uno, al massimo due, tipi di nucleotidi marcati con redox in un singolo filamento di DNA.
Poiché ciascuno dei nucleotidi modificati porta la propria etichetta, che durante il rilevamento elettrochimico fornisce un segnale di ossidazione specifico a potenziali variabili, si possono distinguere i singoli tipi di nucleotidi. Inoltre, la dimensione di ciascun segnale dipende dal numero di copie del dato nucleotide nel DNA, che consente quindi di determinare rapidamente la rappresentazione relativa dei singoli nucleotidi nel DNA misurato.
La codifica elettrochimica di recente sviluppo delle basi del DNA offre una serie di vantaggi, come strumentazione più semplice ed economica e analisi più rapide. Il metodo è promettente per il sequenziamento del DNA e le applicazioni diagnostiche, nonché per lo sviluppo di nuovi chip di DNA.