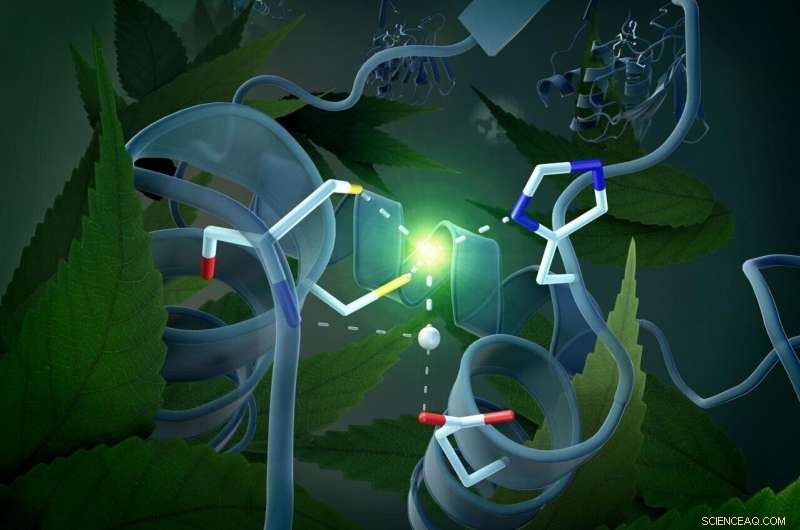
Intorno al centro catalitico c'è un gruppo di molecole, il dominio di accesso, che può occupare due diverse posizioni. Credito:M. Künsting / HZB
Tutte le cellule vegetali ottengono la loro energia principalmente da due organelli che contengono:i cloroplasti (responsabili della fotosintesi) e i mitocondri (responsabili del ciclo biochimico della respirazione che converte gli zuccheri in energia). Però, un gran numero di geni di una cellula vegetale nei suoi mitocondri e cloroplasti può sviluppare difetti, compromettendo la loro funzione. Tuttavia, le cellule vegetali hanno sviluppato uno strumento straordinario chiamato editosoma dell'RNA (un grande complesso proteico) per riparare questo tipo di errori. Può modificare l'RNA messaggero difettoso che risulta da DNA difettoso trasformando (deaminazione) di alcuni nucleotidi di mRNA.
Correzione automatica degli errori nelle cellule vegetali
La correzione automatica degli errori nelle piante è stata scoperta circa 30 anni fa da un team guidato dal fisiologo vegetale Axel Brennicke e da altri due gruppi contemporaneamente. Questo meccanismo converte alcuni nucleotidi citidinici nell'RNA messaggero in uridina per correggere errori nel DNA dei cloroplasti o nel DNA mitocondriale. L'editing dell'RNA è quindi essenziale per processi come la fotosintesi e la respirazione cellulare nelle piante. Anni dopo, ulteriori studi hanno dimostrato che un gruppo di proteine denominate proteine PPR con domini DYW svolge un ruolo centrale nell'editing dell'RNA delle piante. Queste proteine PPR con domini DYW sono trascritte nel nucleo cellulare e migrano attraverso le cellule verso i cloroplasti e i mitocondri. Però, sono inattivi nel loro cammino verso questi organelli. Solo una volta che sono all'interno degli organelli diventano attivi ed eseguono la loro funzione in uno specifico sito di mRNA. Come funziona questa attivazione, però, è stato un mistero fino ad ora.
Non funziona in provetta
Per molti anni, non è stato possibile produrre sinteticamente queste proteine PPR di tipo DYW in laboratorio per studiarne la funzione e la struttura più da vicino. Solo ora un team tedesco-giapponese guidato dal biologo strutturale e biochimico Dr. Gert Weber del Joint Protein Crystallography Group dell'Helmholtz-Zentrum Berlin e della Freie Universität Berlin è riuscito a farlo.
Ora:struttura 3D della proteina chiave decodificata
Il gruppo del prof. Mizuki Takenaka era stato precedentemente in grado di produrre il dominio DYW nei batteri. Takenaka conduce ricerche presso l'Università di Kyoto dal 2018 e in precedenza ha lavorato nel laboratorio di Axel Brennicke a Ulm, Germania. Tatiana Barthel (Università di Greifswald e ora presso HZB) è stata quindi in grado di far crescere i primi cristalli proteici del dominio DYW. Un gran numero di questi delicati cristalli è stato ora analizzato alle linee di luce MX di BESSY II in modo da poter decodificare l'architettura tridimensionale del dominio DYW. "Grazie al Joint Research Group con sede presso HZB e FU Berlin, abbiamo la capacità di tempo del fascio per misurazioni molto rapidamente quando necessario, che è stato fondamentale, "dice il dottor Manfred Weiss, che è responsabile delle linee di luce MX presso BESSY II e coautore dello studio.
Scoperto meccanismo di attivazione
Questa architettura tridimensionale ha effettivamente fornito l'indizio cruciale per il meccanismo di attivazione del dominio DYW che si applica a tutte le piante. È dovuto a un atomo di zinco situato al centro del dominio DYW che può accelerare la deaminazione della citidina in uridina come un catalizzatore. Perché ciò accada, però, lo zinco deve essere posizionato in modo ottimale. L'interruttore di attivazione è fornito da un dominio di gating molto insolito nelle immediate vicinanze del centro catalitico:l'analisi strutturale mostra che questo dominio di gating può assumere due posizioni diverse, attivando o disattivando l'enzima. "Il movimento del dominio di gating regola la misura in cui lo ione zinco è disponibile per la reazione catalitica, "Spiega Weber.
Una molecola come le forbici
Ora è diventato chiaro perché finora è stato difficile far reagire le proteine PPR di tipo DYW con l'RNA nella provetta:queste proteine PPR sono nominalmente inattive e richiedono l'attivazione. Nelle cellule vegetali, sono prima prodotti nel nucleo cellulare e poi molto probabilmente migrano in uno stato inattivato verso gli organelli, dove si attivano. "Questo è l'ideale, perché altrimenti queste molecole sarebbero attive lungo il percorso, alterando in modo incontrollato varie molecole di RNA dannose per la cellula, "dice Weber.
Strumento di riparazione universale
Questo lavoro è una svolta per la biologia molecolare delle piante perché descrive un ulteriore livello di regolazione sofisticata nei cloroplasti e nei mitocondri. I risultati sono fondamentali per la scienza delle piante, ma potrebbero anche svolgere un ruolo nella nostra vita quotidiana un giorno. Il dominio DYW potrebbe fornire uno strumento utile per l'editing dell'RNA C-to-U e U-to-C controllabile e specifico del sito. Questo potrebbe aprire nuove applicazioni mediche e di bioingegneria, come la riprogrammazione di alcuni geni mitocondriali senza modificare il DNA nucleare di una cellula.