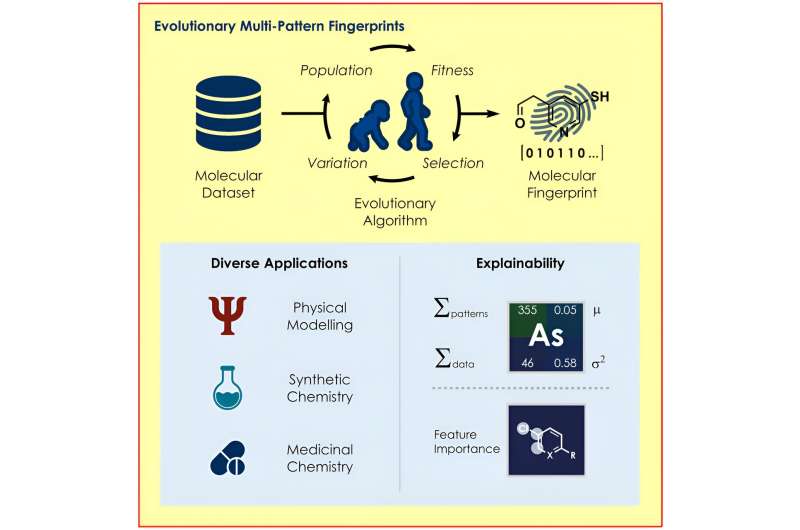
Un team guidato dal professor Frank Glorius dell'Istituto di chimica organica dell'Università di Münster ha sviluppato un algoritmo evolutivo che identifica le strutture di una molecola particolarmente rilevanti per una determinata questione e le utilizza per codificare le proprietà delle molecole per vari modelli di apprendimento automatico.
Il metodo è adatto anche per la previsione automatica delle proprietà chimiche quantistiche e della tossicità delle molecole. Può essere applicato a qualsiasi set di dati molecolari e non richiede una conoscenza approfondita delle relazioni sottostanti.
L’intelligenza artificiale e l’apprendimento automatico stanno diventando sempre più rilevanti nella vita di tutti i giorni, e lo stesso vale per la chimica. I chimici organici, ad esempio, sono interessati a come l'apprendimento automatico possa aiutare a scoprire e sintetizzare nuove molecole efficaci contro le malattie o utili in altri modi.
Il nuovo algoritmo sviluppato dal team di Glorius ricerca rappresentazioni molecolari ottimali basate sui principi dell'evoluzione, utilizzando meccanismi come la riproduzione, la mutazione e la selezione. A seconda del modello e della domanda posta, vengono create "impronte molecolari" personalizzate, che i chimici hanno utilizzato nel loro studio per prevedere le reazioni chimiche con sorprendente precisione.
Il metodo, pubblicato sulla rivista Chem , è adatto anche per prevedere le proprietà chimiche quantistiche e la tossicità delle molecole.
Per utilizzare l’apprendimento automatico, i ricercatori devono prima convertire le molecole in una forma leggibile dal computer. Molti gruppi di ricerca hanno già affrontato questo problema e, di conseguenza, esistono vari modi per svolgere questo compito. Tuttavia, è difficile prevedere quale dei metodi disponibili sia più adatto a rispondere a una domanda specifica, ad esempio per determinare se un composto chimico è dannoso per l'uomo.
Il nuovo algoritmo è progettato per aiutare a trovare l’impronta molecolare ottimale in ciascun caso. Per fare ciò, l'algoritmo seleziona gradualmente le impronte molecolari che ottengono i migliori risultati nella previsione da molte impronte molecolari generate casualmente.
"Seguendo l'esempio della natura, utilizziamo mutazioni, cioè modifiche casuali di singoli componenti delle impronte digitali, o ricombiniamo componenti di due impronte digitali", spiega il dottorando Felix Katzenburg.
"In altri studi, le molecole sono spesso descritte da proprietà quantificabili che sono state selezionate e calcolate dagli esseri umani", aggiunge Glorius.
"Poiché l'algoritmo che abbiamo sviluppato identifica automaticamente le strutture molecolari rilevanti, non ci sono errori sistematici causati da esperti umani."
Un altro vantaggio è che il metodo di codifica permette di capire perché un modello fa una certa previsione. Ad esempio, è possibile trarre conclusioni su quali parti di una molecola influiscono positivamente o negativamente sulla previsione di come si svolgerebbe una reazione, consentendo ai ricercatori di modificare le strutture rilevanti in modo mirato.
Il team di Münster ha scoperto che il loro nuovo metodo non sempre raggiungeva i risultati ottimali.
"Quando è stata impiegata una notevole esperienza umana nella selezione di proprietà molecolari particolarmente rilevanti o quando sono disponibili quantità molto grandi di dati, altri metodi come le reti neurali a volte hanno un vantaggio", afferma Katzenburg.
Tuttavia, uno degli obiettivi principali dello studio era sviluppare un metodo per codificare le molecole che possa essere applicato a qualsiasi set di dati molecolari e non richieda una conoscenza approfondita delle relazioni sottostanti.
Ulteriori informazioni: Philipp M. Pflüger et al, Un algoritmo evolutivo per rappresentazioni molecolari interpretabili, Chem (2024). DOI:10.1016/j.chempr.2024.02.004
Informazioni sul giornale: Chimica
Fornito dall'Università di Münster