
Uno screenshot del sito web DIVE. Credito:Gupta et al.
I documenti accademici contengono spesso resoconti di nuove scoperte e teorie interessanti relative a una varietà di campi. Però, la maggior parte di questi articoli sono scritti utilizzando un gergo e un linguaggio tecnico che possono essere compresi solo da lettori che hanno familiarità con quella particolare area di studio.
I lettori non esperti sono quindi in genere incapaci di comprendere articoli scientifici, a meno che non siano curati e resi più accessibili da terze parti che comprendano i concetti e le idee in essi contenuti. Con questo in testa, un team di ricercatori del Texas Advanced Computing Center dell'Università del Texas ad Austin (TACC), L'Oregon State University (OSU) e l'American Society of Plant Biologists (ASPB) hanno deciso di sviluppare uno strumento in grado di estrarre automaticamente frasi e terminologia importanti da documenti di ricerca al fine di fornire definizioni utili e migliorarne la leggibilità.
"Il nostro progetto è motivato dalla necessità di migliorare la leggibilità degli articoli di riviste, "Weijia Xu, che guidano la squadra di TACC, ha detto a TechXplore. "È uno sforzo congiunto tra curatori biologici, editori di riviste e scienziati informatici miravano a sviluppare un servizio web in grado di riconoscere e consentire la cura dell'autore di importanti terminologia utilizzata nelle pubblicazioni di riviste. La terminologia e le parole vengono poi allegate alla fine dell'articolo di giornale per aumentarne l'accessibilità per i lettori."
Xu e i suoi colleghi hanno sviluppato un framework estensibile che può essere utilizzato per estrarre informazioni dai documenti. Hanno quindi implementato questo framework all'interno di un servizio web chiamato DIVE (Domain Information Vocabulary Extraction), integrandolo con la pipeline di pubblicazioni di riviste dell'ASPB. A differenza degli strumenti esistenti per l'estrazione di informazioni sul dominio, il loro quadro combina diversi approcci, compresa l'estrazione guidata dall'ontologia, estrazione basata su regole, tecniche di elaborazione del linguaggio naturale (PNL) e deep learning.

La panoramica dell'architettura del sistema proposta dai ricercatori. Credito:Gupta et al.
"I risultati ottenuti dai diversi modelli vengono poi archiviati in un database centralizzato, " Xu ha spiegato. "Abbiamo anche progettato un servizio web che consente agli utenti di curare i risultati dell'estrazione. Il servizio web è integrato con la pipeline di pubblicazione di produzione in ASPB."
Una volta che la versione di anteprima di un articolo di giornale è stata inviata ed è entrata nella pipeline di ASPB, il manoscritto viene automaticamente inviato a DIVE, che lo elabora e produce un URL con il quale l'autore potrà accedere ai risultati dell'elaborazione di DIVE. L'autore del documento è invitato a visitare il collegamento fornito e rivedere le informazioni estratte prima di poter inviare ufficialmente il documento.
"L'autore deve visitare il sito DIVE per rivedere i risultati dell'estrazione e fare l'approvazione finale dell'elenco delle informazioni da includere alla fine del loro articolo, " ha detto Xu. "DIVE tiene traccia anche delle correzioni dell'autore per migliorare le future attività di estrazione. Attualmente, nessun altro editore di riviste ha adottato un approccio simile integrandolo con la propria pipeline di pubblicazioni".
Durante le sue analisi e durante l'estrazione dei dati chiave dai documenti, il framework sviluppato dai ricercatori utilizza diverse tecniche. Ciò consente di acquisire più informazioni rispetto ad altri metodi, come ABNER (A Biomedical Named Entity Recognizer), che è uno strumento software open source per il text mining di biologia molecolare che può estrarre solo termini generali (ad esempio geni e proteine). Contrariamente a DIVE, ABNER si basa solo su campi casuali condizionali (CRF), un metodo di modellazione statistica comunemente utilizzato nelle applicazioni di riconoscimento di modelli e di apprendimento automatico.
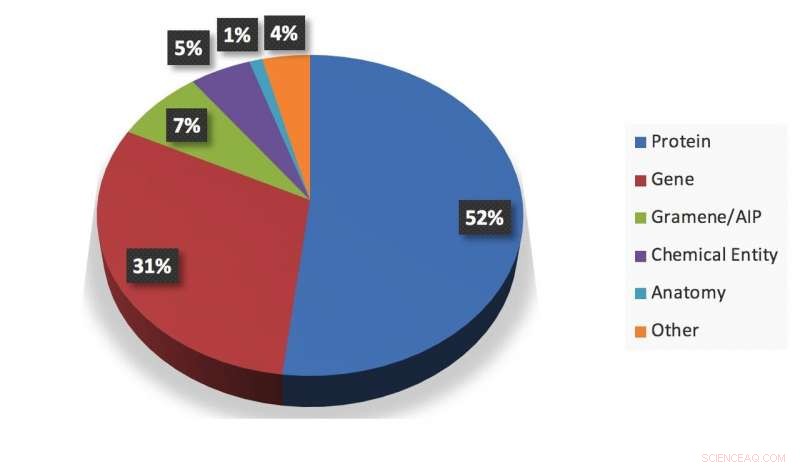
Un riepilogo visivo di un'istantanea delle informazioni estratte dal sistema. Credito:Gupta et al.
"Un importante contributo del nostro progetto è che aiuta a costruire set di dati e modelli che possono dedurre gli interessi di ricerca degli autori dalle loro pubblicazioni, " Xu ha detto. "Il nostro progetto può avvantaggiare comunità più ampie di ricercatori biologici. Per gli autori, le estrazioni e l'inclusione delle informazioni chiave possono aumentare l'accessibilità dei loro articoli."
Xu e il suo collega Amit Gupta hanno valutato il loro framework e confrontato le sue prestazioni con quelle di altri strumenti di estrazione delle informazioni, compreso ABNER. I loro risultati hanno rivelato che utilizzando più approcci, compreso l'apprendimento profondo, DIVE ottiene punteggi di precisione più elevati rispetto ad altri modelli pre-addestrati basati esclusivamente su CRF. interessante, il framework DIVE può anche essere continuamente aggiornato, poiché è possibile aggiungere in qualsiasi momento ulteriori modelli di estrazione.
L'applicazione web DIVE non solo consente ai lettori non esperti di comprendere meglio i documenti accademici, può anche aiutarli a identificare i documenti in linea con i loro interessi. Ricercatori, d'altra parte, può utilizzare DIVE per rimanere informato su particolari aree di ricerca, nonché per conoscere nuova terminologia e tendenze relative al loro campo di interesse. Finalmente, le informazioni generate dall'applicazione possono anche guidare i curatori di biologia nelle loro decisioni e nei processi di raccolta dei dati.
"Stiamo continuando il nostro progetto esplorando due direzioni, " Xu ha detto. "Da un lato, stiamo studiando nuovi metodi da incorporare con i nostri modelli di estrazione delle informazioni per migliorare le prestazioni. D'altra parte, stiamo anche cercando di espandere il nostro servizio offrendolo a ulteriori comunità di utenti ed editori di riviste".
© 2019 Scienza X Rete