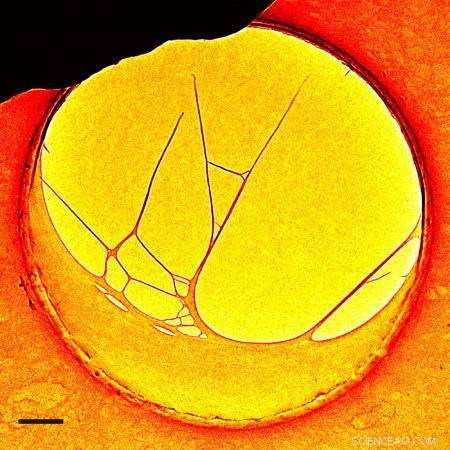
Una struttura del DNA vista attraverso il microscopio elettronico 4D inventato al Caltech. Credito:Zewail &Lorenz/Caltech
(Phys.org)—Ogni grande struttura, dall'Empire State Building al Golden Gate Bridge, dipende da specifiche proprietà meccaniche per rimanere forte e affidabile. La rigidità, la rigidità di un materiale, è di particolare importanza per mantenere la robusta funzionalità di qualsiasi cosa, dagli edifici colossali alla più piccola delle strutture su scala nanometrica. Nelle nanostrutture biologiche, come reti di DNA, è stato difficile misurare questa rigidità, che è essenziale per le loro proprietà e funzioni. Ma gli scienziati del California Institute of Technology (Caltech) hanno recentemente sviluppato tecniche per visualizzare il comportamento delle nanostrutture biologiche sia nello spazio che nel tempo, consentendo loro di misurare direttamente la rigidità e mappare la sua variazione in tutta la rete.
Il nuovo metodo è delineato nella prima edizione del 4 febbraio del Atti dell'Accademia Nazionale delle Scienze ( PNAS ).
"Questo tipo di visualizzazione ci sta portando in domini delle scienze biologiche che non abbiamo esplorato prima, "dice il premio Nobel Ahmed Zewail, il Linus Pauling Professor di Chimica e professore di fisica al Caltech, che è stato coautore del documento con Ulrich Lorenz, uno studioso postdottorato nel laboratorio di Zewail. "Stiamo fornendo la metodologia per scoprire, direttamente, la rigidità di una rete biologica che ha proprietà su scala nanometrica".
Conoscere le proprietà meccaniche delle strutture del DNA è fondamentale per costruire reti biologiche robuste, tra le altre applicazioni. Secondo Zewail, questo tipo di visualizzazione della biomeccanica nello spazio e nel tempo dovrebbe essere applicabile allo studio di altri nanomateriali biologici, compresi gli assemblaggi proteici anomali che sono alla base di malattie come l'Alzheimer e il Parkinson.
Zewail e Lorenz potevano vedere, per la prima volta, il movimento delle nanostrutture di DNA sia nello spazio che nel tempo utilizzando il microscopio elettronico quadridimensionale (4D) sviluppato presso il Centro di biologia fisica per la scienza e la tecnologia ultraveloce di Caltech. Il centro è diretto da Zewail, che lo ha creato nel 2005 per far progredire la comprensione della fisica fondamentale del comportamento chimico e biologico.
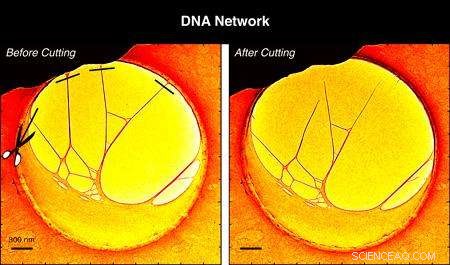
Usando gli elettroni nel microscopio, diversi filamenti di DNA sono stati tagliati via dal film di carbonio (a sinistra) in modo che un tridimensionale, la struttura autoportante è stata ottenuta al microscopio 4D (a destra).
"In natura, il comportamento della materia è determinato dalla sua struttura - la disposizione dei suoi atomi nelle tre dimensioni dello spazio - e da come la struttura cambia nel tempo, la quarta dimensione, " spiega Zewail. "Se guardi un cavallo galoppare al rallentatore, puoi seguire il tempo delle galoppate, e puoi vedere in dettaglio cosa, Per esempio, ogni gamba sta facendo nel tempo. Quando arriviamo alla scala nanometrica, questa è una storia diversa:dobbiamo migliorare la risoluzione spaziale fino a un miliardo di volte quella del cavallo per visualizzare ciò che sta accadendo".
Zewail è stato insignito del Premio Nobel per la Chimica 1999 per il suo sviluppo della femtochimica, che utilizza lampi laser ultracorti per osservare le reazioni chimiche fondamentali che si verificano nella scala temporale del femtosecondo (un milionesimo di miliardesimo di secondo). Sebbene la femtochimica possa catturare atomi e molecole in movimento, dando la dimensione temporale, non può mostrare contemporaneamente le dimensioni dello spazio, e quindi la struttura del materiale. Questo perché utilizza la luce laser con lunghezze d'onda che superano di gran lunga la dimensione di una nanostruttura, making it impossible to resolve and image nanoscale details in tiny physical structures such as DNA .
To overcome this major hurdle, the 4D electron microscope employs a stream of individual electrons that scatter off objects to produce an image. The electrons are accelerated to wavelengths of picometers, or trillionths of a meter, providing the capability for visualizing the structure in space with a resolution a thousand times higher than that of a nanostructure, and with a time resolution of femtoseconds or longer.
The experiments reported in PNAS began with a structure created by stretching DNA over a hole embedded in a thin carbon film. Using the electrons in the microscope, several DNA filaments were cut away from the carbon film so that a three-dimensional, free-standing structure was achieved under the 4D microscope.
Prossimo, the scientists employed laser heat to excite oscillations in the DNA structure, which were imaged using the electron pulses as a function of time—the fourth dimension. By observing the frequency and amplitude of these oscillations, a direct measure of stiffness was made.
"It was surprising that we could do this with a complex network, " says Zewail. "And yet by cutting and probing, we could go into a selective area of the network and find out about its behavior and properties."
Using 4D electron microscopy, Zewail's group has begun to visualize protein assemblies called amyloids, which are believed to play a role in many neurodegenerative diseases, and they are continuing their investigation of the biomechanical properties of these networks. He says that this technique has the potential for broad applications not only to biological assemblies, but also in the materials science of nanostructures.