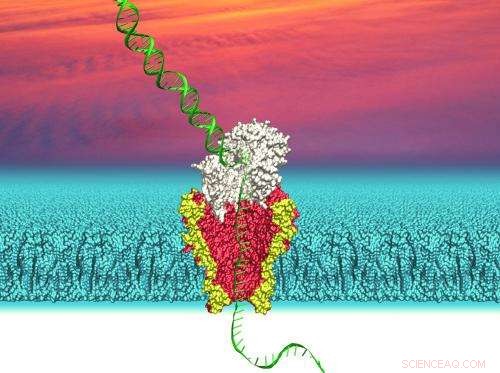
Illustrazione di un nanoporo derivato da un canale di membrana batterica geneticamente modificato utilizzato per sequenziare il DNA. Credito:Ian Derrington
(Phys.org) —Una tecnologia a basso costo può rendere possibile leggere lunghe sequenze di DNA molto più rapidamente delle tecniche attuali.
La ricerca fa avanzare una tecnologia, chiamato sequenziamento del DNA a nanopori. Se perfezionato, potrebbe un giorno essere utilizzato per creare dispositivi portatili in grado di identificare rapidamente sequenze di DNA da campioni di tessuto e dall'ambiente, hanno detto i ricercatori dell'Università di Washington che hanno sviluppato e testato l'approccio.
"Uno dei motivi per cui le persone sono così entusiaste del sequenziamento del DNA dei nanopori è che la tecnologia potrebbe essere utilizzata per creare dispositivi simili a "tricorder" per rilevare agenti patogeni o diagnosticare malattie genetiche rapidamente e sul posto, " ha detto Andrew Laszlo, autore principale e studente laureato nel laboratorio di Jen Gundlach, un professore di fisica della UW che ha guidato il progetto.
Il documento "Decoding long nanopore sequencing reads of natural DNA" descrive la nuova tecnica. Appare il 25 giugno nell'edizione online avanzata della rivista Biotecnologie naturali .
La maggior parte delle attuali tecnologie di sequenziamento genico richiedono di lavorare con brevi frammenti di DNA, tipicamente da 50 a 100 nucleotidi. Questi devono essere elaborati da grandi sequenziatori in un laboratorio. Il processo ingombrante può richiedere giorni o settimane per essere completato.
La tecnologia Nanopore sfrutta il piccolo, strutture a tunnel che si trovano nelle membrane batteriche. In natura, tali pori consentono ai batteri di controllare il flusso di nutrienti attraverso le loro membrane.
Il ricercatore UW ha utilizzato il nanoporo Mycobacterium smegmatis porina A (MspA). Questo poro batterico è stato geneticamente modificato in modo che la parte più stretta del canale abbia un diametro di circa un nanometro, o 1 miliardesimo di metro. Questo è abbastanza grande da consentire il passaggio di un singolo filamento di DNA. Il nanoporo modificato viene quindi inserito in una membrana che separa due soluzioni saline per creare un canale che collega le due soluzioni.
Per leggere una sequenza di DNA con questo sistema, una piccola tensione viene applicata attraverso la membrana per far fluire gli ioni della soluzione salina attraverso il nanoporo. Il flusso di ioni crea una corrente misurabile. Se un filamento di DNA viene aggiunto alla soluzione su un lato della membrana e poi entra in un poro, le ingombranti molecole di DNA ostacoleranno il flusso dello ione molto più piccolo e quindi altereranno la corrente. Quanto i cambiamenti attuali dipendono da quali nucleotidi:le singole molecole adenina, guanina, citosina e timina che costituiscono la catena del DNA, sono all'interno del poro. Rilevare i cambiamenti nella corrente può rivelare quali nucleotidi stanno passando attraverso il canale del nanoporo in un dato istante.
Poiché la tecnica è stata proposta per la prima volta negli anni '90, i ricercatori speravano che il sequenziamento del DNA dei nanopori offrisse una soluzione economica, rapida alternativa all'attuale sequenziamento genico. Ma i loro tentativi sono stati frustrati da diverse sfide. È difficile identificare ogni nucleotide uno per uno mentre passano attraverso il nanoporo. Anziché, i ricercatori devono lavorare con i cambiamenti di corrente associati a quattro nucleotidi alla volta. Inoltre, alcuni nucleotidi possono essere persi o letti più di una volta. Di conseguenza, L'attuale tecnologia di sequenziamento dei nanopori fornisce una lettura imprecisa di una sequenza di DNA.
I ricercatori UW descrivono come hanno aggirato questi problemi. I ricercatori hanno prima identificato le firme elettroniche di tutte le combinazioni di nucleotidi possibili con i quattro nucleotidi che compongono il DNA, per un totale di 256 combinazioni in tutto (4 x 4 x 4 x 4).
Hanno quindi creato algoritmi informatici per abbinare i cambiamenti attuali generati quando un segmento di DNA passa attraverso il poro con i cambiamenti attuali attesi dalle sequenze di DNA di geni noti e genomi archiviati in un database informatico. Una corrispondenza mostrerebbe che la sequenza del DNA che passa attraverso il poro era identica o vicina alla sequenza del DNA memorizzata nel database. L'intero processo richiederebbe da pochi minuti ad alcune ore, invece di settimane.
Per testare questo approccio, i ricercatori hanno usato il loro sistema a nanopori per leggere la sequenza del batteriofago Phi X 174, un virus che infetta i batteri e che è comunemente usato per valutare nuove tecnologie di sequenziamento del genoma. Hanno scoperto che l'approccio leggeva in modo affidabile le sequenze di DNA del batteriofago e poteva leggere sequenze fino a 4, 500 nucleotidi.
"Questa è la prima volta che qualcuno ha dimostrato che i nanopori possono essere utilizzati per generare firme interpretabili corrispondenti a sequenze di DNA molto lunghe da genomi del mondo reale, ", ha affermato il co-autore Jay Shendure, un professore associato UW di scienze del genoma il cui laboratorio sviluppa applicazioni di tecnologie di sequenziamento del genoma. "È un grande passo avanti".
Poiché la tecnica si basa sulla corrispondenza delle letture con i database di geni e genomi precedentemente sequenziati, non può ancora essere utilizzato per sequenziare un gene o un genoma appena scoperto, i ricercatori hanno detto, ma con qualche affinamento, hanno aggiunto, dovrebbe essere possibile migliorare le prestazioni in questo settore. Per accelerare la ricerca su questa nuova tecnologia, gli scienziati stanno facendo i loro metodi, dati e algoritmi informatici a completa disposizione di tutti.
"Nonostante gli ostacoli rimasti, la nostra dimostrazione che un dispositivo a basso costo può leggere in modo affidabile le sequenze di DNA naturale e può interpretare segmenti di DNA fino a 4, 500 nucleotidi di lunghezza rappresentano un importante progresso nel sequenziamento del DNA dei nanopori, " disse Gundlach.