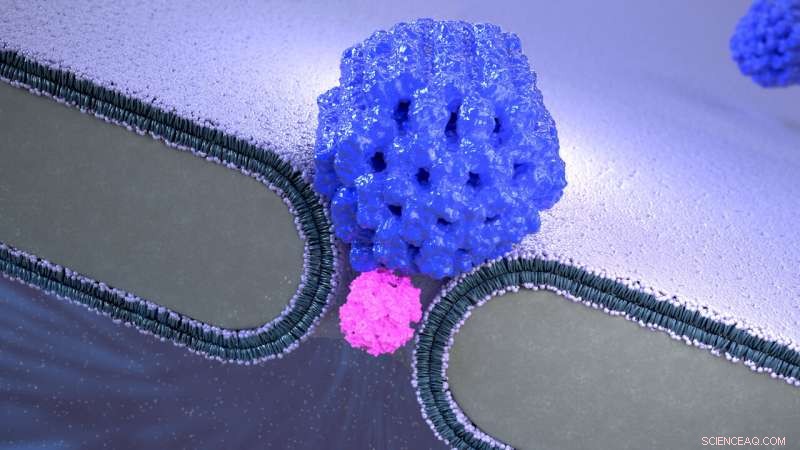
Rappresentazione artistica della trappola elettro-osmotica Nanopore (NEOtrap):un nuovo metodo per l'analisi delle singole molecole. Una nanosfera di DNA-origami copre un nanoforo solido, permettendogli di intrappolare singole molecole proteiche non modificate, consentendo analisi sensibili alla conformazione della durata di un'ora. Credito:Cees Dekker Lab / SciXel
I ricercatori delle università tecniche di Delft e Monaco hanno inventato un nuovo tipo di trappola molecolare in grado di trattenere una singola proteina per ore per studiarne il comportamento naturale, un milione di volte in più rispetto a prima. La nuova tecnica NEOtrap consente agli scienziati di utilizzare le correnti elettriche per studiare la natura vibrante delle proteine, che possono stimolare l'innovazione nella biomedicina, biotecnologia, e altro ancora.
Anche se le proteine sono fondamentali per la vita, fornendoti visione e connessioni neurali per leggere questo testo, per esempio, il modo in cui cambiano forma è ancora poco compreso. Come pubblicato lunedì 30 agosto in Nanotecnologia della natura , un team guidato da Cees Dekker della Delft University of Technology ha sviluppato una nuova tecnica, chiamata trappola elettro-osmotica Nanopore (NEOtrap), studiare le singole molecole proteiche per molto più tempo di quanto fosse possibile prima. Il NEOtrap consente ai ricercatori di misurare come le singole proteine cambiano forma nel tempo.
Come un tappo su una bottiglia
Il NEOtrap combina due nanotecnologie:nanopori a stato solido e origami di DNA. I nanopori sono piccoli fori che gli scienziati usano come sensori per singole molecole come le proteine. Poiché le proteine normalmente passano attraverso il piccolo foro in microsecondi, possono essere registrati solo brevemente. Sigillando il nanoforo con una sfera su scala nanometrica costruita interamente da DNA (!), i ricercatori possono bloccare la proteina in posizione per ore, proprio come un tappo di sughero sigilla una bottiglia di vino. Hendrik Dietz e il suo gruppo presso l'Università tecnica di Monaco hanno costruito questa nano-sfera utilizzando un approccio chiamato "DNA origami", una tecnica che imita la piegatura degli origami su macroscala, utilizzando filamenti di DNA su nanoscala anziché carta.
Autrice principale dell'articolo Sonja Schmid, che ha sviluppato il NEOtrap come postdoc nel laboratorio di Dekker, spiega:"Questa nanosfera di DNA-origami si comporta come una spugna che succhia l'acqua attraverso il nanoporo, attirando una singola proteina nel nanoporo e intrappolandola lì. Ciò significa che possiamo studiare quella proteina per periodi di tempo molto lunghi. In questo lavoro dimostriamo già che possiamo distinguere tra diversi tipi di proteine, e anche diverse forme funzionali di una stessa proteina."
Progresso radicale nel campo
Cees Dekker aggiunge:"Questa nuova tecnica è davvero un importante passo avanti:un revisore anonimo del nostro articolo l'ha definita "uno dei progressi più radicali nel campo del rilevamento dei nanopori". il NEOtrap ci permette di intrappolare una singola proteina nativa senza la necessità di modificare la molecola di interesse, rispetto alle tecniche precedenti. Questa tecnica può, Per esempio, aiutare i ricercatori a scoprire il meccanismo alla base degli enzimi e di altre importanti proteine che cambiano la loro forma per facilitare le reazioni chimiche".
Il NEOtrap consente agli scienziati di tutto il mondo di eseguire esperimenti completamente nuovi, con il potenziale per rivelare caratteristiche funzionali precedentemente trascurate delle proteine e quindi stimolare l'innovazione in biomedicina, biotecnologia, e altro ancora. Schmid (che ora ha avviato il proprio laboratorio a Wageningen) e Dekker stanno pianificando molti studi successivi sulla dinamica delle singole proteine negli anni a venire.