Un team di scienziati guidati dall'Università di Oxford ha ottenuto un risultato significativo nel rilevamento delle modifiche sulle strutture delle proteine. Il metodo, pubblicato su Nature Nanotechnology , utilizza l'innovativa tecnologia dei nanopori per identificare le variazioni strutturali a livello di singola molecola, anche in profondità all'interno delle lunghe catene proteiche.
Le cellule umane contengono circa 20.000 geni che codificano proteine. Tuttavia, il numero effettivo di proteine osservate nelle cellule è molto maggiore, con oltre 1.000.000 di strutture diverse conosciute. Queste varianti vengono generate attraverso un processo noto come modificazione post-traduzionale (PTM), che avviene dopo che una proteina è stata trascritta dal DNA.
La PTM introduce cambiamenti strutturali come l'aggiunta di gruppi chimici o catene di carboidrati ai singoli aminoacidi che compongono le proteine. Ciò si traduce in centinaia di possibili variazioni per la stessa catena proteica.
Queste varianti svolgono un ruolo fondamentale in biologia, consentendo una regolazione precisa di processi biologici complessi all’interno delle singole cellule. La mappatura di questa variazione rivelerebbe una ricchezza di informazioni preziose che potrebbero rivoluzionare la nostra comprensione delle funzioni cellulari. Ma fino ad oggi, la capacità di produrre inventari proteici completi è rimasta un obiettivo irraggiungibile.
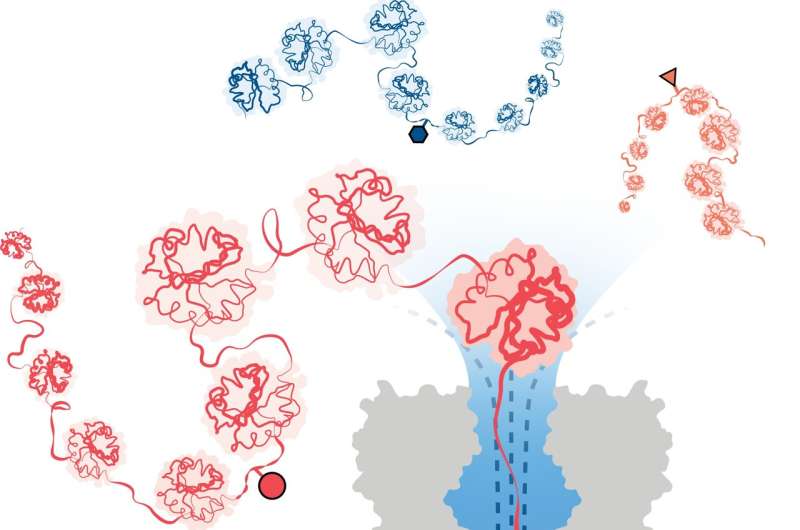
Per superare questo problema, un team guidato da ricercatori del Dipartimento di Chimica dell'Università di Oxford ha sviluppato con successo un metodo per l'analisi delle proteine basato sulla tecnologia di sequenziamento del DNA/RNA tramite nanopori. In questo approccio, un flusso direzionale di acqua cattura e dispiega le proteine 3D in catene lineari che vengono alimentate attraverso minuscoli pori, appena sufficientemente larghi da consentire il passaggio di una singola molecola di amminoacido.
Le variazioni strutturali vengono identificate misurando i cambiamenti in una corrente elettrica applicata attraverso il nanoporo. Molecole diverse causano interruzioni diverse nella corrente, conferendo loro una firma unica.
Il team ha dimostrato con successo l'efficacia del metodo nel rilevare tre diverse modifiche PTM (fosforilazione, glutationilazione e glicosilazione) a livello di singola molecola per catene proteiche lunghe oltre 1.200 residui. Queste includevano modifiche profonde nella sequenza della proteina. È importante sottolineare che il metodo non richiede l'uso di etichette, enzimi o reagenti aggiuntivi.
Secondo il gruppo di ricerca, il nuovo metodo di caratterizzazione delle proteine potrebbe essere facilmente integrato nei dispositivi portatili di sequenziamento dei nanopori esistenti per consentire ai ricercatori di costruire rapidamente inventari proteici di singole cellule e tessuti. Ciò potrebbe facilitare la diagnostica presso il punto di cura, consentendo il rilevamento personalizzato di specifiche varianti proteiche associate a malattie tra cui cancro e disturbi neurodegenerativi.
Il professor Yujia Qing (Dipartimento di Chimica, Università di Oxford), autore dello studio, ha dichiarato:"Questo metodo semplice ma potente apre numerose possibilità. Inizialmente, consente l'esame di singole proteine, come quelle coinvolte in malattie specifiche . A lungo termine, il metodo ha il potenziale per creare inventari estesi di varianti proteiche all'interno delle cellule, sbloccando conoscenze più approfondite sui processi cellulari e sui meccanismi delle malattie."
Il professor Hagan Bayley (Dipartimento di Chimica, Università di Oxford), autore collaboratore e co-fondatore di Oxford Nanopore Technologies, ha aggiunto:"La capacità di individuare e identificare le modifiche post-traduzionali e altre variazioni proteiche a livello di singola molecola rappresenta un'enorme promessa per far progredire la nostra comprensione delle funzioni cellulari e delle interazioni molecolari. Potrebbe anche aprire nuove strade per la medicina personalizzata, la diagnostica e gli interventi terapeutici."
Questo lavoro è stato svolto in collaborazione con il gruppo di ricerca del meccanobiologo Sergi Garcia-Maynes del King's College di Londra e del Francis Crick Institute.
Ulteriori informazioni: Pablo Martin-Baniandres et al, Rilevazione di nanopori senza enzima di modifiche post-traduzionali all'interno di polipeptidi lunghi, Nature Nanotechnology (2023). DOI:10.1038/s41565-023-01462-8
Informazioni sul giornale: Nanotecnologia naturale
Fornito dall'Università di Oxford