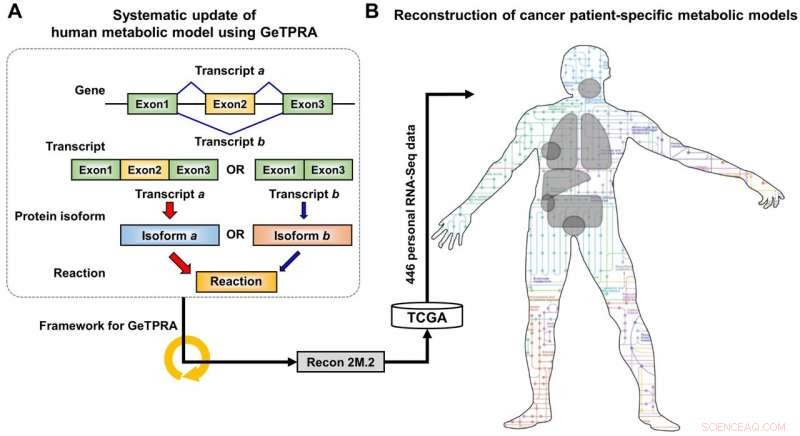
Schema di sviluppo di Recon 2M.2 e suo utilizzo nella ricostruzione di modelli metabolici personali su scala genomica (GEM). (A) Nello sviluppo di Recon 2M.2, è stato considerato un concetto di splicing alternativo di geni umani, e incorporati nella modellazione attraverso il framework computazionale per le associazioni di reazione tra geni e proteine (GeTPRA). (B) Dopo lo sviluppo di Recon 2M.2, i GEM specifici del paziente oncologico potrebbero essere ricostruiti utilizzando dati biologici personali (dati RNA-Seq). In questo studio, I dati RNA-Seq specifici del paziente sono stati ottenuti da The Cancer Genome Atlas (TCGA; https://cancergenome.nih.gov/). Attestazione:KAIST
Un team di ricerca coreano di KAIST ha sviluppato un framework computazionale che consente la ricostruzione di un modello computazionale completo del metabolismo umano, che consente una previsione accurata delle caratteristiche metaboliche personali (o fenotipi).
La comprensione dei fenotipi metabolici personali ci consente di progettare strategie terapeutiche efficaci per varie malattie croniche e infettive. Un modello computazionale umano chiamato modello metabolico su scala del genoma (GEM) contiene informazioni su migliaia di geni metabolici e le loro reazioni e metaboliti corrispondenti, e ha svolto un ruolo importante nella predizione dei fenotipi metabolici. Sebbene siano state rilasciate diverse versioni di GEM umane, avevano spazio per ulteriori sviluppi, soprattutto per quanto riguarda l'incorporazione di informazioni biologiche provenienti da un meccanismo genetico umano chiamato "splicing alternativo". Lo splicing alternativo è un meccanismo genetico che consente a un gene di dare origine a reazioni multiple, ed è fortemente associato alla patologia.
Per affrontare questo problema, Jae Yong Ryu (uno studente di dottorato), Dr. Hyun Uk Kim (Ricercatore), e illustre professor Sang Yup Lee, tutti dal Dipartimento di Ingegneria Chimica e Biomolecolare del KAIST, sviluppato un quadro computazionale che genera sistematicamente reazioni metaboliche, e li aggiunge al GEM umano. È stato dimostrato che il GEM umano risultante è in grado di prevedere con precisione i fenotipi metabolici in varie condizioni ambientali. I risultati della ricerca sono stati pubblicati online in Atti dell'Accademia Nazionale delle Scienze ( PNAS ) il 24 ottobre, 2017, sotto il titolo "Quadro e risorse per più di 11, 000 associazioni di reazione genica-trascrizione-proteina nel metabolismo umano."
Il team di ricerca ha prima aggiornato i contenuti biologici di una versione precedente del GEM umano. I contenuti biologici aggiornati includono i geni metabolici e i loro corrispondenti metaboliti e reazioni. In particolare, reazioni metaboliche catalizzate da isoforme proteiche già note sono state inoltre incorporate nel GEM umano; le isoforme proteiche sono varianti multiple di proteine generate da singoli geni attraverso il processo di splicing alternativo. Ogni isoforma proteica è spesso responsabile del funzionamento di una reazione metabolica. Sebbene più isoforme proteiche generate da un gene possano svolgere funzioni diverse avendo diversi insiemi di domini proteici e/o localizzazioni subcellulari, tali informazioni non erano adeguatamente considerate nelle versioni precedenti delle gemme umane.
Dopo l'aggiornamento iniziale del GEM umano, denominato Recon 2M.1, il team di ricerca ha successivamente implementato un framework computazionale che genera sistematicamente informazioni sulle associazioni di reazione tra geni e proteine (GeTPRA) al fine di identificare le isoforme proteiche che in precedenza non erano state identificate. Questo quadro è stato sviluppato in questo studio. A seguito dell'implementazione del framework per GeTPRA, più di 11, 000 GeTPRA sono stati previsti automaticamente, e completamente convalidato. Ulteriori reazioni metaboliche sono state quindi aggiunte a Recon 2M.1 in base al GeTPRA previsto per le isoforme proteiche precedentemente non caratterizzate; Recon 2M.1 è stato rinominato Recon 2M.2 da questo aggiornamento. Finalmente, Recon 2M.2 è stato integrato con 446 set di dati biologici personali (dati RNA-Seq) al fine di costruire modelli di cancro specifici per il paziente. Questi modelli di cancro specifici per il paziente sono stati utilizzati per prevedere le attività del metabolismo del cancro e gli obiettivi antitumorali.
Si prevede che lo sviluppo di una nuova versione di GEM umani insieme al quadro computazionale per GeTPRA darà impulso agli studi sulla genetica umana fondamentale e sulla medicina. File modello delle gemme umane Recon 2M.1 e 2M.2, un elenco completo del GeTPRA e il codice sorgente per il framework computazionale per prevedere il GeTPRA sono tutti disponibili come parte della pubblicazione di questo studio.
Il distinto professor Lee ha detto, "Il previsto GeTPRA dal quadro computazionale dovrebbe servire come linea guida per futuri esperimenti sulla genetica umana e la biochimica, considerando che il Recon 2M.2 risultante può essere utilizzato per prevedere i bersagli farmacologici per varie malattie umane."