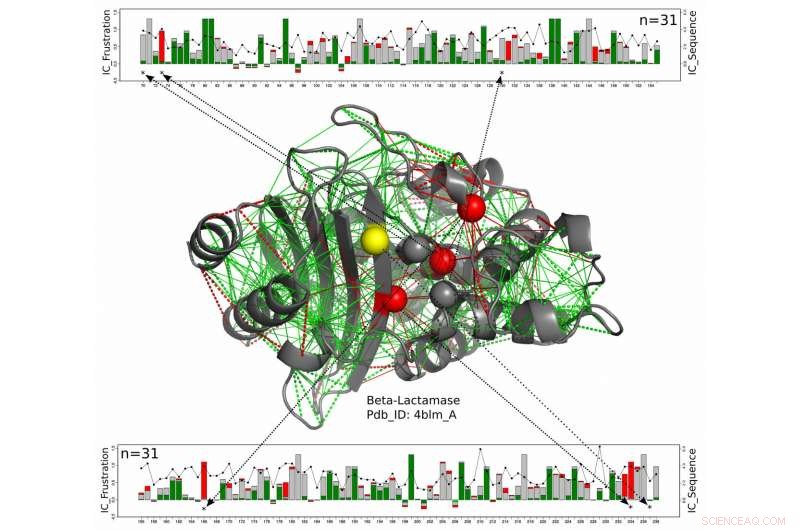
In questo modello strutturale per una proteina beta-lattamasi, i residui catalitici sono mostrati come sfere con quelli che sono costantemente altamente frustrati lungo l'intera famiglia delle beta-lattamasi mostrati in rosso e quelli per i quali lo stato frustrato non è conservato in giallo. Il nuovo lavoro degli scienziati della Rice University, l'Università di Buenos Aires e il Laboratorio Europeo di Biologia Molecolare suggeriscono questi enzimi, che devono restare esposti per reagire con i loro bersagli, ottenere una misura di protezione dagli amminoacidi un po' frustrati che li circondano. Le linee verdi rappresentano una frustrazione minima, le linee rosse rappresentano un'elevata frustrazione. I grafici lineari in alto e in basso mostrano le posizioni degli amminoacidi rilevanti sulla catena proteica e una misura della conservazione dell'energia sull'intera famiglia di proteine beta-lattamasi. Credito:Maria Feiberger/Università di Buenos Aires
Le proteine si ripiegano secondo uno script scritto nel modello dei loro amminoacidi. Ma per funzionare correttamente, alcune parti devono ad-lib.
I siti attivi degli enzimi, proteine che accelerano e guidano le reazioni biochimiche, rendere possibile la vita. Per fare questo, le loro istruzioni devono essere in qualche modo in conflitto con il piano strutturale generale della proteina, secondo gli scienziati della Rice University, l'Università di Buenos Aires e il Laboratorio europeo di biologia molecolare di Heidelberg, Germania.
Secondo i ricercatori, l'evoluzione ha selezionato parti di enzimi come abbastanza importanti da poter deviare energeticamente dalle istruzioni che portano a un imbuto pieghevole, che guida le proteine nei loro stati funzionali a bassa energia. Esaminando essenzialmente tutti gli enzimi conosciuti, i ricercatori hanno anche confermato il sospetto che livelli aggiuntivi di interazioni più debolmente frustrate circondano e supportano gli stessi siti catalitici.
La teoria dettagliata negli Atti della National Academy of Sciences estende il principio accettato di minima frustrazione che spiega perché le proteine possono progredire senza problemi dalle sequenze lineari originariamente codificate nel loro DNA al loro funzionamento, forme globulari tridimensionali. Questo principio riconosce il fatto che la frustrazione spesso rimane irrisolta quando il conflitto energetico rimanente serve in qualche modo alla funzione della proteina.
Secondo Peter Wolynes di Rice, che ha enunciato il principio in forma matematica più di 20 anni fa, i paesaggi energetici delle proteine con parti catalitiche meritano un'analisi extra, data l'importanza che l'evoluzione ha loro assegnato.
"Una molecola perfettamente piegata, non importa quanto sia bella come scultura, non posso fare molto, " ha detto. "Devi avere una proteina che, pur essendo per lo più piegato, ha ancora alcune cerniere frustrate che sono incompatibili con la piegatura ma sono necessarie per consentire il movimento necessario per le reazioni chimiche."
La frustrazione a livello atomico è semplice da provare:basta unire i poli positivi di due magneti e sentire come si respingono. Come magneti, gli amminoacidi di una proteina ripiegabile si attraggono e si respingono l'un l'altro mentre (per lo più) risolvono i conflitti e alla fine arrivano a formare una disposizione stabile di contatti.
"Ora aggiungi un terzo magnete, e puoi incontrare una frustrazione di cui non puoi liberarti, " ha detto il co-autore Wolynes, che ha lavorato con il team di Buenos Aires guidato dal chimico biologico Diego Ferreiro, un ex ricercatore post-dottorato nel suo gruppo.
Il nuovo studio mostra che la frustrazione tra gli amminoacidi vicini dell'enzima, come nella situazione con il terzo magnete, agisce per aiutare l'enzima frustrato a mantenere la sua posizione. Rimane alquanto instabile ma ha regioni disponibili per legarsi e modificare le molecole bersaglio.
Chiaramente, Wolynes ha detto, evoluzione ha priorità che superano l'estetica di ottenere una piega perfetta.
Il team di ricerca ha esaminato in modo computazionale centinaia di strutture proteiche note e ha scoperto che intere famiglie di enzimi condividono le firme caratteristiche della frustrazione del sito attivo che si estendono al secondo e al terzo strato di amminoacidi, o conchiglie, intorno all'enzima.
"La cosa interessante che abbiamo scoperto è che la frustrazione generalmente si estende oltre il primo guscio assoluto, " ha detto. "Ciò significa che c'è una sottigliezza imbarazzante ma necessaria ai vincoli funzionali che richiede di ottenere questi tre gusci giusti".
Wolynes ha notato che il premio Nobel Frances Arnold e altri che studiano l'evoluzione artificiale in laboratorio hanno visto prove di una prolungata frustrazione nei loro sforzi per modificare gli enzimi per nuove reazioni.
"A volte cambiavano specificamente il sito attivo per catalizzare reazioni chimiche diverse da quelle naturali, ma ha scoperto che anche gli amminoacidi distanti dal sito attivo dovevano cambiare per far funzionare l'enzima in modo efficiente, " ha detto. "Non era ovvio perché, ma i modelli mostrano che i cambiamenti nel secondo e nel terzo guscio migliorano la capacità catalitica degli enzimi.
"Quindi i nostri risultati non sono stati uno shock completo, ma è bello vedere che la frustrazione estesa è abbastanza prevalente in natura ed è un tema comune a tutte le classi di enzimi conosciute, " Egli ha detto.