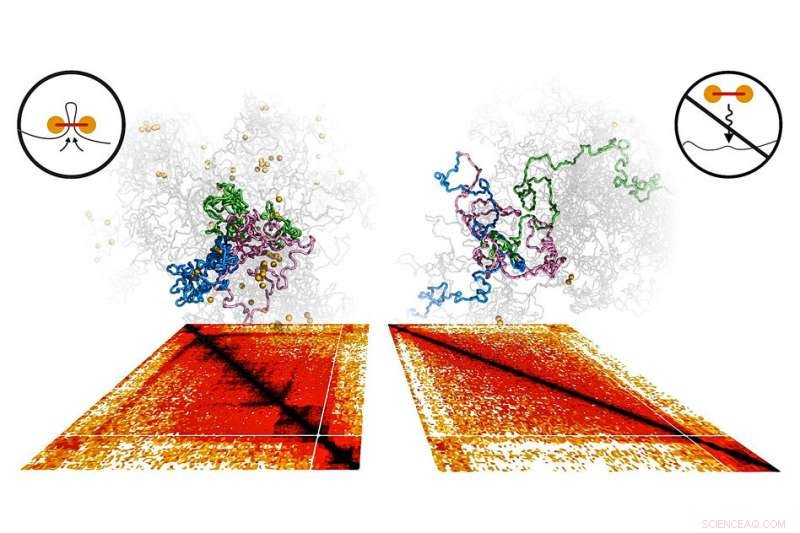
I modelli computerizzati dei cromosomi e le mappe sperimentali Hi-C mostrate sotto di essi rivelano il ruolo centrale della coesione nel piegare il genoma in domini distinti (evidenziati in blu, verde, e rosa). Quando la coesione è esaurita sperimentalmente e nelle simulazioni, i domini si dispiegano e si mescolano (a destra). Credito:Massachusetts Institute of Technology
Un "motore" molecolare che organizza il genoma in quartieri distinti formando anelli di DNA è stato caratterizzato dai ricercatori del MIT e dell'Istituto Pasteur in Francia.
In uno studio pubblicato nel 2016, una squadra guidata da Leonid Mirny, un professore di fisica presso l'Istituto per l'ingegneria e le scienze mediche del MIT, proposto che i motori molecolari trasformano i cromosomi da uno stato vagamente aggrovigliato in una serie dinamica di anelli in espansione.
Il processo, noto come estrusione ad anello, si pensa che riunisca gli elementi regolatori con i geni che controllano. Il team ha anche suggerito che il DNA è decorato con barriere, simili a segnali di stop, che limitano il processo di estrusione.
In questo modo, l'estrusione ad anello divide i cromosomi in quartieri regolatori separati, noti come domini ad associazione topologica (TAD).
Però, mentre i ricercatori hanno suggerito che un complesso proteico simile ad un anello chiamato coesina era un probabile candidato per questi motori molecolari, questo doveva ancora essere dimostrato.
Ora, in un articolo pubblicato sulla rivista Natura , un team guidato da Mirny e Francois Spitz presso l'Istituto Pasteur, hanno dimostrato che la coesione svolge effettivamente il ruolo di motore nel processo di estrusione dell'anello.
"Ognuna di queste macchine atterra sul DNA e inizia a estrudere anelli, ma ci sono limiti sul DNA che questi motori non possono superare, " dice Mirny. "Quindi come risultato di questa attività motoria, il genoma è organizzato in molti loop dinamici che non ne attraversano i confini, così il genoma si divide in una serie di quartieri."
I ricercatori hanno anche scoperto che un meccanismo diverso, che non usa la coesione, è al lavoro organizzando le regioni attive e inattive del DNA in compartimenti separati nel nucleo della cellula.
Per determinare il ruolo della coesione nella formazione del genoma, il team ha prima eliminato una molecola nota come Nipbl, che è responsabile del caricamento della coesione sul DNA.
Hanno quindi utilizzato una tecnica sperimentale nota come Hi-C, in cui vengono catturate e sequenziate parti di DNA vicine l'una all'altra nello spazio 3D, nel tentativo di misurare la frequenza delle interazioni fisiche tra i diversi punti lungo i cromosomi.
Questa tecnica, che è stato introdotto da Job Dekker, professore di biochimica e farmacologia molecolare presso il Medical Center dell'Università del Massachusetts a Worcester, è stato precedentemente utilizzato per dimostrare l'esistenza di DAT.
Il team ha utilizzato per la prima volta la tecnica Hi-C per valutare l'organizzazione dei cromosomi prima di rimuovere la molecola Nipbl dai topi. Hanno quindi rimosso la molecola ed eseguito nuovamente la stessa misurazione.
Hanno scoperto che i quartieri erano praticamente scomparsi.
Però, la compartimentazione tra regioni attive e inattive del genoma era diventata ancora più marcata.
Il team ritiene che i motori della coesione consentano a ciascun gene di raggiungere i suoi elementi regolatori, che controllano se i geni devono essere attivati o disattivati.
Cosa c'è di più, sembra che i motori della coesione siano fermati da un'altra proteina, CTCF, che delimita i confini di ogni quartiere. In un recente studio sulla rivista Cellula , il laboratorio Mirny, in collaborazione con ricercatori dell'Università della California a San Francisco e della University of Massachusetts Medical School ha dimostrato che se questa proteina di demarcazione viene rimossa, scompaiono i confini tra i quartieri, permettendo ai geni in un quartiere di parlare con elementi regolatori con cui non dovrebbero parlare in un altro quartiere, e portando a un'errata regolazione dei geni nella cellula.
"La coesione è fondamentale per la regolazione genica, e sottolineiamo che questa è una funzione motoria, quindi non è solo che loro (i geni e i loro elementi regolatori) si trovano da qualche parte casualmente nello spazio, ma erano accomunati da questa attività motoria, "dice Mirne.
Questo articolo fornisce importanti nuove conoscenze molecolari sui meccanismi con cui le cellule ripiegano i loro cromosomi, secondo Dekker, che non è stato coinvolto nello studio in corso.
"In questo lavoro i laboratori Mirny e Spitz combinano modelli murini con approcci genomici per studiare il ripiegamento dei cromosomi per rivelare che la macchina che carica il complesso della coesione è fondamentale per la formazione di TAD, " dice Dekker. "Da questo e da un altro studio precedente, sta emergendo un meccanismo molecolare in cui i TAD si formano per estrusione del ciclo della cromatina dipendente dalla coesione e da Nipbl, che è bloccato dai siti vincolati da CTCF."
I ricercatori stanno ora tentando di caratterizzare come l'assenza del motore molecolare influenzerebbe la regolazione genica. Stanno anche effettuando simulazioni al computer nel tentativo di determinare come avviene l'estrusione del ciclo a base di coesione mentre il genoma sta subendo il processo indipendente di segregazione in compartimenti attivi e inattivi.
"È come due pianisti che suonano sullo stesso pianoforte, "dice Nezar Abdennur, uno studente di dottorato nel laboratorio di Mirny, che ha preso parte allo studio insieme al collega dottorando Anton Goloborodko. "Essi interferiscono e si limitano a vicenda, ma insieme possono produrre un bel pezzo di musica."
Questa storia è stata ripubblicata per gentile concessione di MIT News (web.mit.edu/newsoffice/), un popolare sito che copre notizie sulla ricerca del MIT, innovazione e didattica.