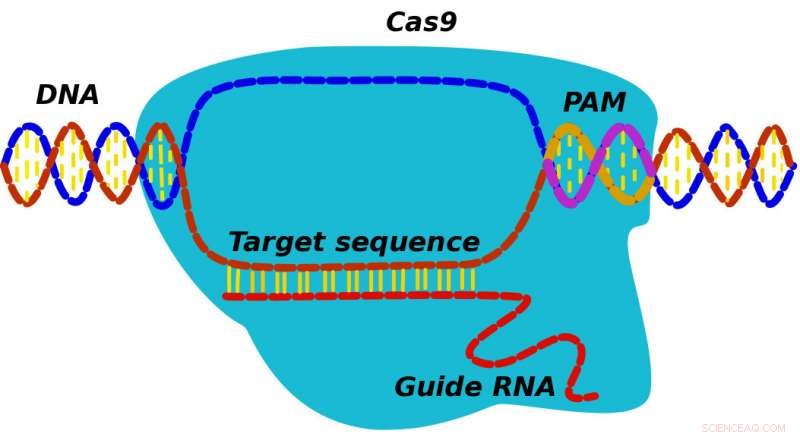
Quando una proteina CRISPR-Cas9 trova il suo bersaglio, prima trova una sequenza spaziatrice nota come PAM e quindi cerca il DNA adiacente per vedere se corrisponde all'RNA guida di Cas9. Un nuovo modello sviluppato dai ricercatori della Rice University potrebbe aiutare a scoprire dettagli sul meccanismo con cui CRISPR-Cas9 può sostituire le mutazioni con il DNA corretto. Credito:Alexey Shvets/Rice University
I ricercatori della Rice University hanno sviluppato un modello computazionale per quantificare il meccanismo con cui le proteine CRISPR-Cas9 trovano i loro obiettivi di modifica del genoma.
Anatoly Kolomeisky, un professore di chimica e ingegneria chimica e biomolecolare della Rice, e l'alunno Alexey Shvets ha adattato un sistema sviluppato in precedenza per mostrare come le proteine generalmente trovano i loro bersagli biologici. Sperano che il modello rivisto aiuti a svelare i restanti misteri di CRISPR.
Nel suo stato naturale, CRISPR, che sta per "brevi ripetizioni palindromiche a grappolo regolarmente interspaziate, " è il meccanismo biologico mediante il quale i batteri si proteggono dalle infezioni virali. I batteri incorporano una copia del DNA estraneo e costruiscono un registro di tutti quelli che invadono. Si riferiscono a quel registro quando vengono rilevati nuovi invasori e lo usano per distruggerli.
Negli ultimi anni, i ricercatori hanno iniziato ad adattare il meccanismo per l'uso nell'editing del genoma, che ha il potenziale per curare le malattie e migliorare gli organismi, compresi gli umani. Ma un ostacolo è stato il rischio che le proteine CRISPR-Cas9, uno dei sistemi che utilizzano l'approccio CRISPR, taglierà e sostituirà le sequenze target sbagliate, introduzione di mutazioni.
Il modello di riso descritto nel Giornale Biofisico ha ritenuto probabile che CRISPR-Cas9 localizzi buoni bersagli in modo più efficiente quando queste modifiche fuori bersaglio possono verificarsi, perché le proteine non perdono tempo a dissociarsi dai target fuori bersaglio per continuare la ricerca.
Potrebbe o non potrebbe essere una buona cosa, ma è certamente degno di studio, disse Kolomeisky.
"Il tasso di errore (taglio fuori bersaglio) a volte è del 10-20 percento, " ha detto. "Abbiamo due idee su questo:una è che i virus mutano molto rapidamente e forse i batteri stanno cercando di tagliare obiettivi che sono solo leggermente mutati come un modo per essere più flessibili. L'altro è che ci sono proteine che possono correggere gli errori, quindi se non ci sono molti tagli sbagliati, il sistema li può tollerare.
Kolomeisky ha affermato che il suo modello è un semplice passo verso la comprensione delle dinamiche dell'editing CRISPR. "CRISPR-Cas9 è la variante più popolare perché ha una sola proteina ed è più facile, biologicamente, lavorare con, " Egli ha detto.
Il laboratorio Rice ha sviluppato il suo modello originale per imparare come le proteine scivolano lungo il DNA per trovare bersagli e innescare processi come la trascrizione genica. Kolomeisky ha notato che la pioniera di CRISPR Jennifer Doudna ha scoperto che CRISPR-Cas9 non cerca allo stesso modo. "Ha scoperto che non scorre da nessuna parte sul DNA, " Egli ha detto.
Anziché, secondo Doudna e il suo team, la proteina riconosce inizialmente sequenze PAM a tre nucleotidi (per motivo adiacente protospacer) che segnano la posizione di potenziali bersagli. "CRISPR trova e si lega al PAM e quindi il suo RNA associato esplora il DNA adiacente per vedere se questo è il bersaglio, " disse Kolomeisky. "Se lo è, la proteina inizia a tagliare. Altrimenti, si dissocia e guarda altrove."
Nei successivi esperimenti di Doudna con le sequenze PAM rimosse, Le proteine CRISPR-Cas9 non sono riuscite a trovare i loro bersagli. Quindi i PAM hanno un ruolo importante e non sono solo un generico distanziatore, Egli ha detto. "Appena ho letto questo, Ho capito che anche qui potevamo usare il nostro modello".
Il modello teorico esamina i processi di primo passaggio, quelli che si verificano quando un sistema supera una soglia fisica o chimica, come trovare un PAM rilevante, per tracciare le proteine CRISPR-Cas9 inserite in una cellula mentre prima esaminano le sequenze PAM e poi, mentre legato ai PAM, cerca il bersaglio del DNA che corrisponda all'RNA di Cas9.
Hanno scoperto che i CRISPR che evitano tagli fuori bersaglio dissociandosi dal DNA "sbagliato" richiedono più tempo per stabilirsi rispetto a uno che semplicemente taglia fuori bersaglio. "Andare al PAM sbagliato richiede tempo, " ha detto Kolomeisky. "Il nostro calcolo mostra che CRISPR può trovare obiettivi reali più velocemente quando a volte taglia nei posti sbagliati. La frazione che va ai bersagli giusti può essere più piccola, ma alla fine li taglierai.
"È un modello semplice ed esattamente risolvibile, " ha detto Kolomeisky. "Se qualcuno vuole testare, il modello può fornire previsioni specifiche e in alcuni casi offrire tendenze per ciò che dovrebbe essere osservato." Ciò che manca al modello è la capacità di vedere se la chiave dell'RNA riconosce il suo bersaglio simultaneamente, legandosi al DNA tutto in una volta, o sequenzialmente, nucleotide per nucleotide.
"La cosa più impressionante di CRISPR non è la scoperta di un sistema immunitario nei batteri, ma il fatto che questo abbia creato una rivoluzione nella biotecnologia, perché significa che in ogni cellula possiamo tagliare qualsiasi DNA in una posizione specifica, molto preciso, " ha detto Kolomeisky. "Spero che il nostro lavoro stimolerà studi più fondamentali, perché mi piace molto il metodo CRISPR. Ma non sono felice quando le persone lo applicano senza capire come funziona a livello molecolare".
Shvets è ora ricercatore post-dottorato presso il Massachusetts Institute of Technology. Kolomeisky è professore di chimica e di ingegneria chimica e biomolecolare.