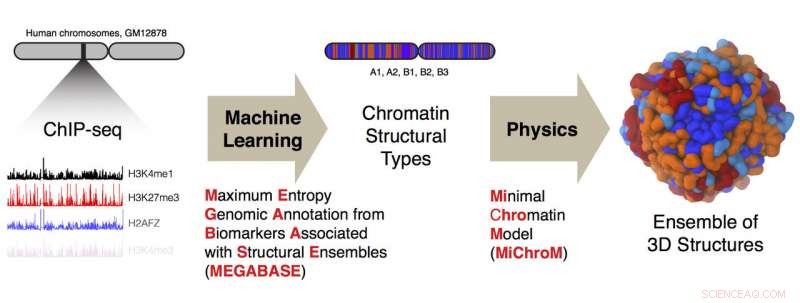
I ricercatori della Rice University e del Baylor College of Medicine hanno sviluppato una pipeline computazionale per convertire i dati di sequenziamento ChIP unidimensionali sul DNA in strutture tridimensionali dei cromosomi umani. Credito:Ryan Cheng/Michele Di Pierro
Il DNA in una cellula umana è lungo 2 iarde (1,83 metri) e avvolge milioni di proteine istoniche simili a sferette per adattarsi all'interno del nucleo della cellula. I ricercatori della Rice University e del Baylor College of Medicine hanno dimostrato che l'esame dello stato chimico di queste proteine consente di prevedere come si piegherà un intero cromosoma del DNA.
I ricercatori del Rice's Center for Theoretical Biological Physics (CTBP) hanno costruito modelli al computer per analizzare i segni epigenetici, che includono proteine legate al DNA e modificazioni chimiche alle proteine istoniche. Hanno raccolto le informazioni codificate in questi segni per prevedere come i cromosomi si piegano in tre dimensioni.
Le loro scoperte avvicinano il campo della genetica alla capacità di prevedere la struttura ripiegata di interi genomi, che un giorno potrebbe aiutare a identificare le malattie genetiche legate al misfolding.
Il lavoro appare questa settimana in Atti dell'Accademia Nazionale delle Scienze .
Impacchettato nel nucleo, Il DNA si piega in una forma funzionale che differisce in vari tipi di cellule. Poiché ogni cellula di un organismo contiene lo stesso DNA, i segni epigenetici lo aiutano a trovare la forma giusta per il tipo di cellula in cui vive.
"Qualcosa in cima al codice genetico dice alla cellula cosa dovrebbe essere e determina quali parti del cromosoma verranno lette in un dato momento, " disse il biofisico Peter Wolynes, un coautore del documento. "Questi sono i cosiddetti segni epigenetici".
Collettivamente, i segni epigenetici aiutano a impacchettare il genoma nei compartimenti sciolti ma altamente organizzati che adotta durante l'interfase, la "mezza età" lavorativa nella vita di una cellula. Questi comparti portano i geni correlati alla trascrizione nelle immediate vicinanze e consentono loro di comunicare e funzionare.
I segni epigenetici possono essere rivelati da una tecnica consolidata chiamata ChIP-sequencing, che mappa i siti di legame alle proteine lungo il DNA.
"Non capiamo esattamente come viene marcato il genoma, ma possiamo misurarlo attraverso il sequenziamento ChIP, che è diventato un esperimento abbastanza semplice, " Wolynes ha detto. "Nello stesso modo in cui possiamo visualizzare il codice genetico (il DNA), possiamo anche misurare questi segni direttamente in molte celle diverse. Sono diventati lo strato successivo di sequenza sul genoma".
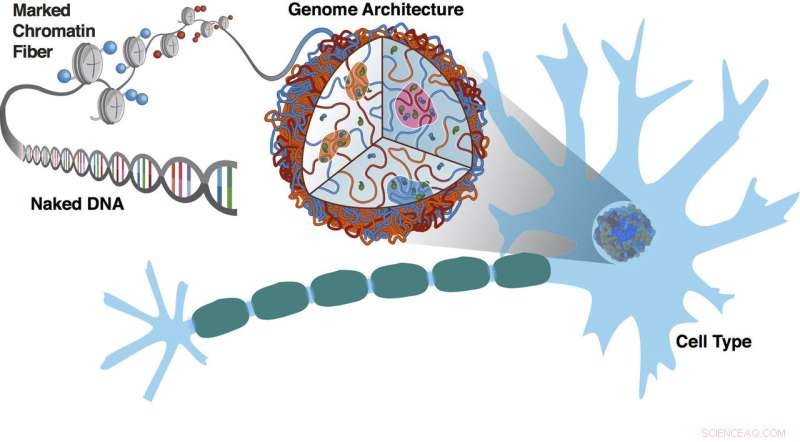
Esperimenti presso la Rice University e il Baylor College of Medicine mostrano come segmenti di cromatina con gli stessi modelli di marcatura epigenetica si localizzino insieme in un processo correlato alla separazione di fase. Il DNA nudo è decorato da segni epigenetici che codificano la disposizione tridimensionale dei cromosomi. L'architettura del genoma e i modelli di marcatura sono caratteristiche del tipo cellulare, in questo caso una cellula nervosa con la sua caratteristica guaina mielinica. Credito:Sigrid Knemeyer/Centro di fisica biologica teorica presso la Rice University
"È un altro livello di informazioni, " ha detto il co-autore e biofisico José Onuchic. "Il DNA di ogni cellula è lo stesso. Però, diversi tipi di cellule hanno epigenetica diversa, quindi i loro modelli di espressione sono diversi."
Gli autori co-leader e i borsisti post-dottorato Rice Michele Di Pierro e Ryan Cheng hanno utilizzato i dati di sequenziamento ChIP per una cellula linfoblastica umana che sonda 84 diverse proteine leganti il DNA e 11 modificazioni chimiche degli istoni. Le proteine istoniche aiutano a organizzare il genoma agendo come bobine attorno alle quali si avvolge il DNA.
Utilizzando i dati di solo alcuni dei cromosomi, hanno addestrato una rete neurale personalizzata chiamata MEGABASE (Maximum Entropy Genomic Annotation from Biomarkers Associated with Structural Ensembles) per produrre una sequenza di tipi di cromatina. Ciò ha rivelato come i segni epigenetici fossero correlati ai compartimenti, loro hanno detto. Una volta addestrato, hanno convalidato il modello MEGABASE fornendogli dati dai cromosomi rimanenti. Ciò ha prodotto una nuova serie di tipi strutturali per l'analisi da parte del programma MiChroM del team Rice, un cugino dell'algoritmo del paesaggio energetico AWSEM del laboratorio che prevede le strutture delle proteine. L'algoritmo MiChroM ha previsto le strutture 3-D dei cromosomi.
"I nostri risultati supportano l'idea che la compartimentazione nei cromosomi derivi dalla separazione di fase di diversi tipi di cromatina nel nucleo, come la separazione di olio e acqua, " Ha detto Cheng.
Quando i ricercatori hanno ridotto il set di dati originale a soli 11 segni istonici e hanno eseguito nuovamente i calcoli, i risultati sono stati solo marginalmente diversi. In definitiva, hanno determinato che i soli dati sugli istoni sono sufficienti per predire la forma di un cromosoma. "C'è un codice ben definito che collega le marcature istoniche alla struttura, " Di Pierro ha detto. "È ben conservato, quindi è probabile che abbia una funzione."
Per convalidare la loro teoria, i ricercatori hanno confrontato i loro risultati con le mappe di contatto delle cellule linfoblastiche generate da Hi-C. Questa tecnica sperimentale, che utilizza il sequenziamento ad alto rendimento per identificare i modelli di piegatura nel DNA, è stato sviluppato dal co-autore Erez Lieberman Aiden, direttore del Center for Genome Architecture di Baylor e ricercatore senior presso il CTBP.
"Questo documento dice che possiamo prendere informazioni unidimensionali sugli istoni e usarle con i nostri strumenti di big data per prevedere la struttura tridimensionale, "Ha detto Wolynes.
Il loro successo avvicina il team all'obiettivo finale di una teoria che predice l'architettura di un intero genoma. Però, rimane un problema della gallina o dell'uovo:la cromatina si piega a causa dei marcatori, o i pennarelli appaiono a causa della piegatura?
"Fa tutto parte del nostro fascino per come funziona la vita, " Di Pierro ha detto. "È un bel problema".