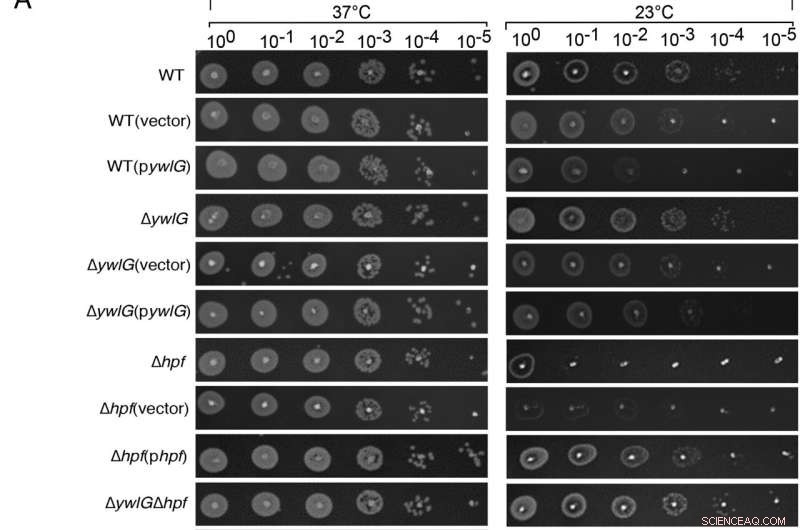
L'inattivazione di ywlG sopprime la sensibilità al freddo di un mutante Δhpf-null in modo medio-dipendente. La complementazione con ywlG e hpf codificati da plasmidi ha confermato la soppressione mediata da YwlG e la risposta al freddo dipendente da HPF. I saggi spot di diluizione seriale sono stati eseguiti su piastre di agar a base di brodo di lisogenia (LB) o (B) a base di brodo di soia triptico (TSB). Cellule a crescita esponenziale (OD600 =0,6) in TSB sono stati adeguati a OD600 =0,2 dopo due lavaggi in 1xPBS. Dopo diluizioni seriali, sulle piastre di agar sono stati individuati 2 µl di ciascuna diluizione. I risultati sono stati registrati dopo 24 ore e 48 ore di incubazione rispettivamente a 37 °C e 23 °C. Le immagini sono rappresentative di tre esperimenti indipendenti. Credito:Atti dell'Accademia Nazionale delle Scienze (2022). DOI:10.1073/pnas.2207257119
I ricercatori della Northwestern Medicine hanno scoperto nuovi meccanismi regolatori che promuovono la sopravvivenza batterica, secondo i risultati pubblicati negli Proceedings of National Academy of Sciences .
Lo studio, guidato da M.-N. Frances Yap, Ph.D., professore associato di Microbiologia-Immunologia, pone anche le basi per l'identificazione di nuovi bersagli terapeutici per il trattamento delle infezioni batteriche.
L'ibernazione dei ribosomi è un meccanismo svolto da batteri e altri organismi, inclusi gli eucarioti, per garantire la sopravvivenza a lungo termine. I ribosomi 100S in letargo nei batteri sono costituiti da due complessi 70S uniti tra loro da una proteina del fattore che promuove l'ibernazione (HPF).
Questo complesso 100S ha due scopi principali:prevenire la degradazione dei macchinari per la biosintesi delle proteine (ribosomi) e conservare l'energia nella cellula interrompendo la traduzione dell'mRNA.
Nel presente studio, il team di Yap ha utilizzato la spettrometria di massa per studiare le interazioni proteiche nello Staphylococcus aureus resistente alla meticillina, un batterio comunemente noto per causare infezioni da stafilococco. Da queste analisi, hanno scoperto che l'HPF interagisce con un partner di legame precedentemente non identificato situato al di fuori del ribosoma, una proteina chiamata YwlG.
Inoltre, risolvendo la struttura atomica di YwlG, hanno scoperto che questa proteina è coinvolta nell'attività della glutammato idrogenasi, che è una via metabolica essenziale richiesta per la sopravvivenza dei batteri.
"In condizioni normali, una frazione della proteina YwlG è legata all'HPF. Legandosi insieme, l'YwlG non può stimolare l'attività dell'idrogenasi del glutammato e l'HPF non può legarsi al ribosoma, quindi è un 'sequestro reciproco' delle due proteine per garantire la giusta quantità di Vengono prodotti complessi 100S e glutammato deidrogenasi cellulare", ha affermato Yap.
I risultati indicano sia HPF che YwlG come fattori che influenzano la colonizzazione batterica e la gravità dell'infezione. L'obiettivo finale, secondo Yap, è identificare un composto in grado di interrompere la formazione di questi complessi e, a sua volta, inibire la sopravvivenza a lungo termine dei batteri.
"Un vantaggio chiave è che la proteina simile a HPF o YwlG non esiste negli esseri umani, quindi potresti sviluppare un antibatterico mirato a HPF o YwlG che inibisce la crescita batterica ma non danneggia gli ospiti", ha detto Yap. + Esplora ulteriormente