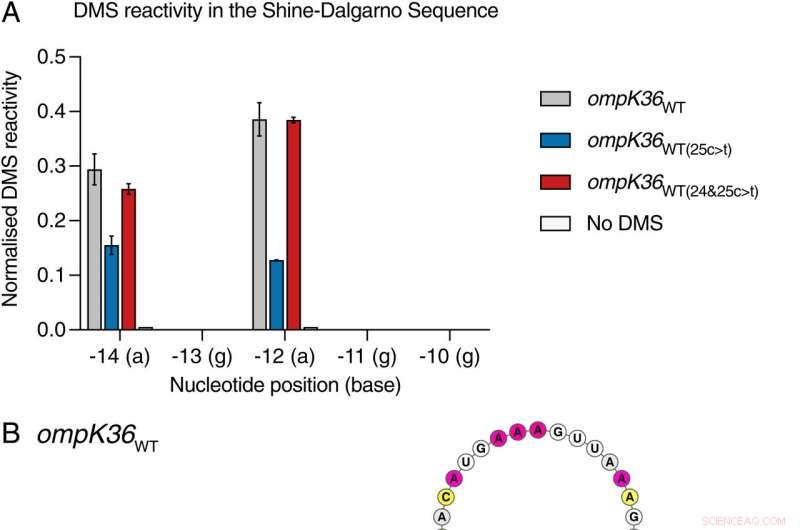
Posizione 25 nell'ompK36WT(25c> t) L'RNA induce uno stelo che coinvolge l'SDS. (A) Segnale DMS normalizzato affiancato per nucleotide nell'SDS da ompK36 trascritto e ripiegato a tutta lunghezza in vitroWT , ompK36WT(25c> t) , ompk36WT(24&25c> t) e ompK36WT non trattato con DMS . Valori più elevati corrispondono a una maggiore accessibilità di base. Viene mostrato il segnale DMS (± SD) di 2 ripetizioni biologiche per nucleotidi da -14a a -10g. (B–D). Modelli di struttura vincolati da DMS dell'estremità 5′ di ompK36WT (B), ompK36WT(24&25c> t) (C) e ompK36WT(25c> t) (D). I nucleotidi sono colorati dal segnale DMS normalizzato. Le SDS nelle strutture dell'RNA sono evidenziate in grigio. Le frecce indicano la posizione -14 e la posizione 25c> t in ompK36WT(25c> t) . Credito:Atti dell'Accademia Nazionale delle Scienze (2022). DOI:10.1073/pnas.2203593119
I ricercatori hanno scoperto un nuovo modo in cui le infezioni nosocomiali resistono agli antibiotici, attraverso una mutazione genetica "silenziosa".
I batteri possono acquisire resistenza agli antibiotici attraverso mutazioni casuali nel loro DNA che forniscono loro un vantaggio che li aiuta a sopravvivere. Trovare mutazioni genetiche e scoprire come aiutano i batteri a sopravvivere all'attacco di antibiotici è fondamentale per aiutarci a combattere con nuovi farmaci.
I ricercatori hanno ora scoperto una mutazione "silenziosa" nel codice genetico che porta alla resistenza agli antibiotici. Tipicamente, mutazioni di questo tipo verrebbero trascurate e potrebbero essere già presenti in altri batteri infettivi.
Il team, guidato da ricercatori dell'Imperial College London e che include collaboratori internazionali, ha pubblicato oggi i risultati sulla rivista Proceedings of the National Academy of Sciences .
Resistenza in aumento
I ricercatori hanno esaminato il batterio Klebsiella pneumoniae, che causa infezioni ai polmoni, al sangue e alle ferite degli ospedali, con i pazienti che hanno un sistema immunitario compromesso, come quelli nelle unità di terapia intensiva, particolarmente vulnerabili.
Come molti batteri, K. pneumoniae sta diventando sempre più resistente agli antibiotici, in particolare a una famiglia di farmaci chiamati carbapenemi. Questi importanti farmaci di ultima istanza vengono utilizzati negli ospedali quando altri antibiotici hanno già fallito.
Poiché l'aumento della resistenza ai carbapenemi potrebbe influenzare notevolmente la nostra capacità di trattare le infezioni, K. pneumoniae resistente ai carbapenemi è classificato come organismi "critici" di Priorità 1 dell'Organizzazione Mondiale della Sanità.
Per essere efficaci, gli antibiotici devono penetrare all'interno dei batteri e in K. pneumoniae ciò avviene attraverso un canale nella membrana esterna del batterio, formato da una proteina chiamata OmpK36. Il team ha scoperto una mutazione genetica che fa sì che i batteri producano meno proteine, chiudendo efficacemente alcuni di questi canali e tenendo lontani gli antibiotici carbapenemici.
Mutazioni "silenziose"
Questa mutazione, tuttavia, funziona in modo diverso rispetto alle mutazioni standard che provocano resistenza agli antibiotici. Solitamente, le mutazioni modificano il codice genetico in modo che quando viene "letto" dai ribosomi e convertito in una proteina, produca una diversa catena di aminoacidi con diverse funzioni.
Questa mutazione produce ancora la stessa catena di amminoacidi, ma altera la struttura di un importante mRNA intermedio, impedendo ai ribosomi di leggere il codice e di produrre proteine da esso.
Quando si cercano le mutazioni, le tecniche genomiche di solito cercano cambiamenti nella sequenza di amminoacidi. Tuttavia, poiché questa mutazione altera una struttura, piuttosto che la sequenza stessa, potrebbe essere pensata come una mutazione "silenziosa".
Il primo autore, il dottor Joshua Wong, del Dipartimento di scienze della vita dell'Imperial, ha affermato:"Nell'era dei big data e della genomica, le mutazioni che abbiamo scoperto possono essere considerate" silenziose "in quanto il codice genetico risulta nella stessa sequenza proteica .
"Questa scoperta dovrebbe cambiare il modo in cui vediamo il codice genetico nei batteri e potenzialmente indica che nella comunità scientifica abbiamo trascurato altre mutazioni simili che potrebbero avere effetti importanti. Il nostro lavoro si concentra su una singola mutazione ma cambia fondamentalmente il modo in cui interpretiamo le mutazioni, in particolare quelle che si pensava tacessero."
Guidato dall'uso di antibiotici
Il team dell'Imperial, che ha caratterizzato la mutazione, ha lavorato con i team dell'Università di Oxford, dell'Università di Firenze e dell'Università di Harvard per identificare la distribuzione della mutazione a livello globale, valutare i livelli di resistenza e determinare in che modo la mutazione ha influenzato l'mRNA intermedio struttura.
Utilizzando i dati provenienti da campioni di batteri resistenti raccolti a livello globale, il team ha mostrato che la mutazione si era manifestata diverse volte indipendentemente. Questo suggerisce che non è casuale, ed è invece guidato dalla necessità dei batteri di difendersi nuovamente dagli antibiotici.
Il ricercatore capo, il professor Gad Frankel, del Dipartimento di scienze della vita dell'Imperial, ha dichiarato:"La mutazione si è evoluta in diverse occasioni in modo indipendente, e questo ci dice che questo nuovo meccanismo non è un caso fortuito, ma guidato dal consumo di antibiotici. Questo suggerisce che la mutazione si verifica sotto pressione antibiotica e mette in evidenza gli effetti collaterali dell'uso eccessivo di antibiotici negli ospedali e in altri contesti."
Il team ora spera che la loro scoperta venga incorporata in strumenti bioinformatici che analizzino le sequenze genetiche per identificare la presenza della mutazione, come è stato fatto con un meccanismo precedente scoperto dal team.
Continueranno inoltre a lavorare con i loro collaboratori per cercare altre importanti mutazioni in questo patogeno chiave. + Esplora ulteriormente