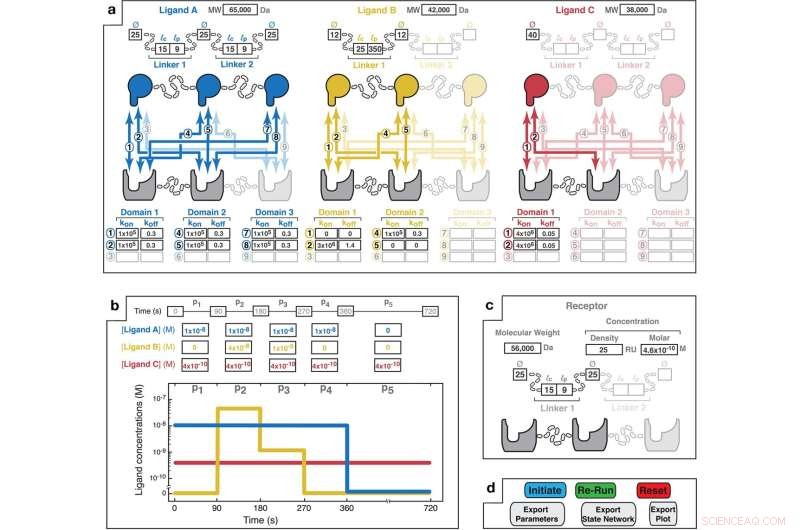
L'interfaccia di progettazione degli input di MVsim fornisce la specifica dei parametri interattivi per i sistemi di interazione multivalente e multimolecolare. un Un'interfaccia point-and-click consente all'utente di selezionare il numero di ligandi (fino a tre) e le valenze dei ligandi e dei recettori (fino a trivalenti) che compongono il sistema multivalente. Sulla base del disegno scelto, l'utente specifica la struttura di ciascuno dei leganti inserendo il peso molecolare (MW) applicabile; i diametri dei domini di legame (Ø); le lunghezze del contorno (lc dei linker (cioè la distanza massima end-to-end; es., 3,5 Å e 1,5 Å per amminoacido rispettivamente per una bobina casuale e un'alfa-elica); e le lunghezze di persistenza (lp) del linker. Inoltre, sono evidenziate le interazioni combinatorie applicabili (numerate da 1 a 9) uniche per ciascun accoppiamento recettore-ligando. I campi dei parametri consentono l'immissione di costanti di velocità monovalenti per ciascuna interazione a coppie. Le interazioni non vincolanti possono essere indicate con k acceso e k disattivato valori pari a zero (ad esempio, come illustrato con il ligando B in giallo per le interazioni "1" e "5"). b Un campo di input consente all'utente di specificare i modelli delle concentrazioni totali di ligando di massa. Una fase di associazione si verifica durante periodi di concentrazione di ligando di massa diversa da zero (ad esempio, 90–270 s per il ligando B). Le fasi di dissociazione si verificano quando il ligando viene rimosso dalla soluzione sfusa (ad esempio, 360–720 s per il ligando A). Qui, il Ligand C è specificato come continuamente presente in soluzione durante i 720 s del corso del tempo di interazione. Il display grafico consente la visualizzazione del pattern di impulso di concentrazione di massa specificato. c Parametri di input dell'utente per il recettore. La concentrazione del recettore può essere specificata come densità superficiale che imita l'SPR (misurata in UR; dove 1 UR equivale a ~1 pg/mm 2 ) o una concentrazione molare. La topologia del recettore è specificata nella stessa forma descritta sopra per i ligandi. d Il MVsim La scheda controller consente l'avvio, l'iterazione e l'esportazione di simulazioni di associazione. "Inizia" esegue una simulazione. "Re-run" esegue una simulazione abbreviata utilizzata quando non sono state apportate modifiche alla valenza o alla topologia del sistema. "Reset" riavvia l'app e cancella i parametri di input dell'utente da tutti i campi. Credito:Comunicazioni sulla natura (2022). DOI:10.1038/s41467-022-32496-6
Un team guidato dagli ingegneri biomedici dell'Università del Minnesota Twin Cities ha sviluppato un'applicazione universalmente accessibile in grado di simulare complesse interazioni molecolari, che consentirà ai ricercatori di progettare trattamenti migliori per malattie come il cancro e il COVID-19.
Il documento si basa su uno studio pubblicato dai ricercatori nel 2019. Ora hanno ampliato la tecnologia per simulare interazioni molecolari ancora più complesse, reso l'applicazione facile da usare per i non esperti e applicato le loro scoperte per far luce su come la SARS -Il virus CoV-2 infetta il corpo.
Lo studio è pubblicato su Nature Communications e l'app, chiamata MVsim, è disponibile gratuitamente per altri ricercatori su GitHub.
Il simulatore prevede la forza, la velocità e la selettività delle interazioni multivalenti, che coinvolgono molecole che hanno più siti di legame e possono essere utilizzate per sviluppare farmaci per malattie, in particolare cancro e COVID-19.
"Le interazioni multivalenti sono davvero importanti nei sistemi biologici naturali e ora stanno iniziando a essere sfruttate in modo creativo per creare nuovi farmaci terapeutici che sfruttano le loro proprietà leganti uniche", ha affermato Casim Sarkar, autore senior dell'articolo e professore all'Università del Minnesota Dipartimento di Ingegneria Biomedica.
"Con i farmaci multivalenti, in linea di principio, è possibile prendere di mira le cellule in modo molto specifico in un modo che non è possibile con i farmaci monovalenti standard, ma ci sono molte variabili da considerare nella loro progettazione e gran parte del lavoro sul campo fino ad oggi è stato svolto attraverso tentativi ed errori sperimentali", ha aggiunto Sarkar. "Ora, usando MVsim, siamo in grado di fare buone previsioni che possono essere utilizzate per progettare in modo più razionale tali terapie."
Molti farmaci antitumorali non solo si legano alle cellule tumorali ma anche a quelle che non sono destinate a colpire, il che spesso crea effetti collaterali indesiderati per il paziente. Ottimizzando la specificità delle interazioni multivalenti utilizzando MVsim, i ricercatori possono progettare farmaci che prendono di mira in modo più specifico le cellule di un tumore riducendo al minimo il legame con altre cellule del corpo.
Un altro esempio è il virus SARS-CoV-2. Gli scienziati sanno che il virus si sta evolvendo per infettare meglio le nostre cellule ed eludere il nostro sistema immunitario, ma i meccanismi molecolari alla base del modo in cui il virus lo fa sono relativamente sconosciuti. Utilizzando la loro tecnologia MVsim, i ricercatori dell'Università del Minnesota sono stati in grado di esplorare questo processo in modo più approfondito, scoprendo le velocità con cui i singoli domini di legame all'interno della proteina spike multivalente del virus passano da uno stato di infezione cellulare a uno stato di elusione immunitaria.
"Essenzialmente abbiamo un microscopio computazionale che ci permette di guardare sotto il cofano e vedere cosa stanno facendo proteine multivalenti come la proteina spike SARS-CoV-2 a livello molecolare", ha spiegato Sarkar. "This level of molecular detail is hard to capture with a physical experiment. One of the real powers of MVsim is that we can not only learn more about how these systems work but we can also use this tool to design new multivalent interactions for diseases like cancer and COVID-19."
The researchers have already identified potential ways to limit the infectivity of current and future SARS-CoV-2 variants, which they plan to test soon. + Esplora ulteriormente