Astratto grafico. Credito:Cellula molecolare (2022). DOI:10.1016/j.molcel.2022.06.023
Utilizzando una nuova tecnica innovativa, gli scienziati della Duke-NUS Medical School e i loro collaboratori hanno identificato migliaia di sequenze di DNA precedentemente sconosciute nel genoma umano che codificano per microproteine e peptidi potenzialmente critici per la salute e le malattie umane.
"Gran parte di ciò che capiamo sul noto 2% del genoma che codifica per le proteine deriva dalla ricerca di lunghi filamenti di sequenze nucleotidiche codificanti proteine, o lunghi frame di lettura aperti", ha spiegato la biologa computazionale Dr. Sonia Chothani, ricercatrice con Programma Duke-NUS sui disturbi cardiovascolari e metabolici (CVMD) e primo autore dello studio. "Recentemente, tuttavia, gli scienziati hanno scoperto piccoli frame di lettura aperti (smORF) che possono anche essere tradotti dall'RNA in piccoli peptidi, che hanno ruoli nella riparazione del DNA, nella formazione muscolare e nella regolazione genetica".
Gli scienziati hanno cercato di identificare gli smORF e i piccoli peptidi per i quali codificano, poiché l'interruzione di questi smORF può causare malattie. Tuttavia, gli approcci attualmente disponibili sono molto limitati.
"Gran parte degli attuali set di dati non forniscono informazioni sufficientemente dettagliate per identificare gli smORF nell'RNA", ha aggiunto il dott. Chothani. "La maggior parte proviene anche da analisi di cellule umane immortalate che vengono propagate, a volte per decenni, per studiare la fisiologia, la funzione e la malattia cellulare. Tuttavia, queste linee cellulari non sono sempre rappresentazioni accurate della fisiologia umana".
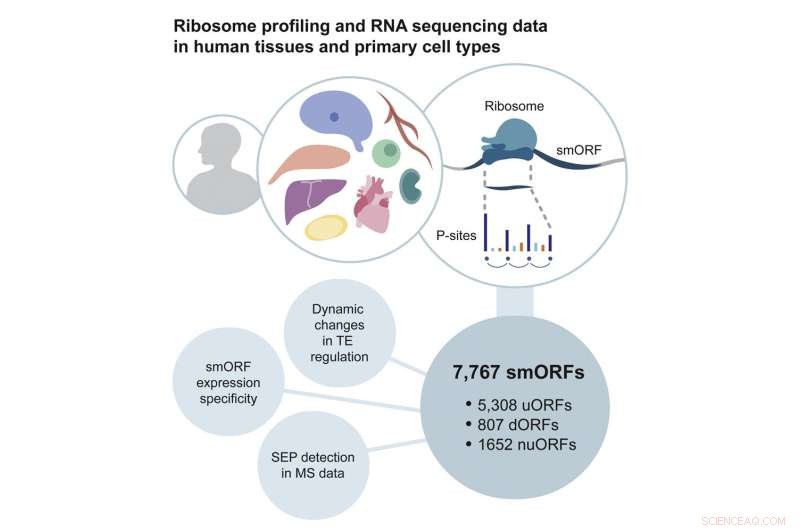
Pubblicazione in Cella molecolare , Chothani e i suoi colleghi a Singapore, in Germania, nel Regno Unito e in Australia descrivono una metodologia che hanno sviluppato per affrontare questi problemi. Hanno esaminato i set di dati di profilazione del ribosoma attualmente disponibili per brevi filamenti di RNA con sezioni periodiche a tre basi, che coprono oltre il 60% della lunghezza dell'RNA. Hanno quindi condotto il proprio sequenziamento dell'RNA e la profilazione del ribosoma per generare una risorsa di dati combinata di sei tipi di cellule e cinque tipi di tessuti, come il cuore e il cervello, derivati da centinaia di pazienti.
L'analisi di questi dati ha identificato quasi 8.000 smORF. È interessante notare che erano altamente specifici per i tessuti in cui sono stati trovati, il che significa che questi smORF possono svolgere una funzione specifica per il loro ambiente. Il team ha anche identificato 603 microproteine codificate da alcuni di questi smORF.
"Il genoma è disseminato di smORF", ha affermato il professore assistente Owen Rackham, autore senior dello studio del programma CVMD. "La nostra mappa completa e risolta nello spazio delle smORF umane evidenzia i componenti funzionali trascurati del genoma, individua nuovi attori nella salute e nelle malattie e fornisce una risorsa per la comunità scientifica come piattaforma per accelerare le scoperte".
Il professor Patrick Casey, vice-decano senior della ricerca presso Duke-NUS, ha dichiarato:"Con il sistema sanitario in evoluzione non solo per curare le malattie ma anche per prevenirle, l'identificazione di potenziali nuovi obiettivi per la ricerca sulle malattie e lo sviluppo di farmaci potrebbe aprire strade a nuove soluzioni. Questa ricerca della dott.ssa Chothani e del suo team, pubblicata come risorsa per la comunità scientifica, porta importanti spunti sul campo". + Esplora ulteriormente