Un gruppo di ricerca ha costruito strumenti genetici versatili per Saccharolobus islandicus REY15A, uno dei pochissimi modelli archeologici per la biologia degli archaea e la ricerca biologica CRISPR.
Tali strumenti includono un efficiente editing del genoma, robusti sistemi di espressione proteica, analisi di plasmidi di interferenza, silenziamento genico e editing genetico basato su CRISPR. Tuttavia, i vettori plasmidici costruiti finora per questo crenarchaeon si basano esclusivamente sul plasmide criptico pRN2.
Lo studio, apparso su mLife , è stato guidato dal Prof. Qunxin She e dal Dr. Guanhua Yuan, entrambi dell'Università Shandong di Qingdao, Cina.
"Per arricchire gli strumenti genetici di questo archeone modello è necessario un doppio sistema ospite-vettore", afferma la prof.ssa She.
Infatti, in una fase iniziale dello sviluppo del vettore archaeale, sia i plasmidi pRN1 che quelli pRN2 che coesistono nel Sa. islandicus REN1H1 sono stati impiegati per costruire vettori navetta per Sa. islandicus REY15A basato su questi due plasmidi; I plasmidi derivati da pRN2 hanno ottenuto un alto tasso di trasformazione e hanno prodotto veri trasformanti, mentre i vettori basati su pRN1 hanno prodotto solo pochissime colonie da cui i plasmidi erano apparentemente assenti.
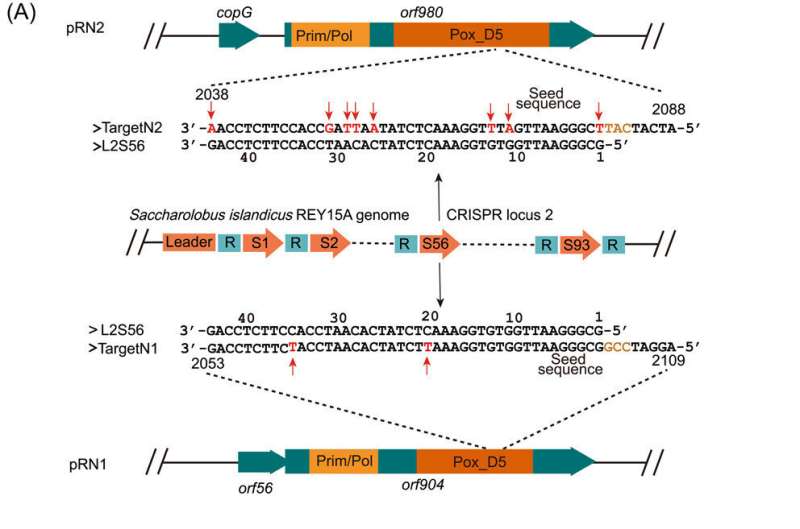
"Poiché gli array CRISPR spesso portano spaziatori che corrispondono a vari plasmidi in Sulfolobales, sospettavamo che il genoma di Sa. islandicus REY15A potesse portare uno spaziatore che corrisponde a una sequenza in pRN1 ma non in pRN2," dice il dottor Yuan.
Dopo aver determinato la sequenza completa del genoma di Sa. islandicus REY15A4, il genoma ospite porta uno spaziatore (L2S56) che mostra solo due disallineamenti con un segmento di DNA (Target N1) nella sequenza codificante del gene della replicasi pRN1. Esperimenti sull’efficienza della trasformazione hanno dimostrato che i crRNA L2S56 erano espressi a un livello che potrebbe essere sufficiente per suscitare l’immunità I-A ma insufficiente per innescare l’immunità III-B per l’eliminazione del plasmide in Sa. islandico REY15A.
Per ottenere un derivato N1 bersaglio funzionale che eluda l'immunità CRISPR dell'ospite, il team ha progettato tre segmenti di DNA (N1a, N1b e N1c) basati sul bersaglio pRN1, mentre le mutazioni progettate in N1a erano sinonimi, N1b e N1c avevano mutazioni missenso. I risultati hanno mostrato che nessuno dei tre bersagli mutati è stato preso di mira dal sistema CRISPR nell'ospite archeologico. Tuttavia, esperimenti successivi hanno dimostrato che N1c porta le mutazioni missenso che potrebbero aver inattivato la proteina di replicazione.
Attraverso la costruzione di una serie di vettori, il Saccharolobus–E. coli shuttle pN1dAA con le mutazioni N1a, il marcatore di selezione argD, l'origine di replicazione p15A e un marcatore resistente alla kanamicina sono stati progettati sulla base del backbone pRN1, che può raggiungere una coesistenza stabile con il plasmide pSeSD derivato da pRN2 in Sa. cellule islandicus REY15A. Ciò ha prodotto un sistema a doppio plasmide per lo studio genetico con questo importante modello archeologico.
Poiché l'esame dei conflitti ospite-plasmide fornisce un mezzo utile per l'identificazione di sistemi plasmide-vettore compatibili, una volta risolto sperimentalmente il conflitto, i plasmidi ingegnerizzati sono quindi molto utili per lo sviluppo di sistemi vettore-ospite, come riportato in questo articolo.
Ulteriori informazioni: Pengpeng Zhao et al, Progettazione razionale di derivati pRN1 illimitati e loro applicazione nella costruzione di un sistema vettoriale a doppio plasmide per Saccharolobus islandicus, mLife (2024). DOI:10.1002/mlf2.12107
Fornito dalla Tsinghua University Press