Un nuovo studio presso l’Università Ebraica rivela approfondimenti sulla regolazione dell’mRNA durante lo sviluppo embrionale. Lo studio fa luce sull'intricato processo di regolazione dell'mRNA durante lo sviluppo embrionale, fornendo nuove informazioni su come le cellule pluripotenti adottano identità specializzate attraverso l'espressione genica.
Il lavoro è pubblicato sulla rivista Nature Communications .La ricerca è stata condotta dal Ph.D. Lo studente Lior Fishman e il suo team sotto la guida del ricercatore Dr. Michal Rabani dell'Istituto Alexander Silberman di Scienze della Vita dell'Università Ebraica e in collaborazione con ricercatori del National Institutes of Health negli Stati Uniti
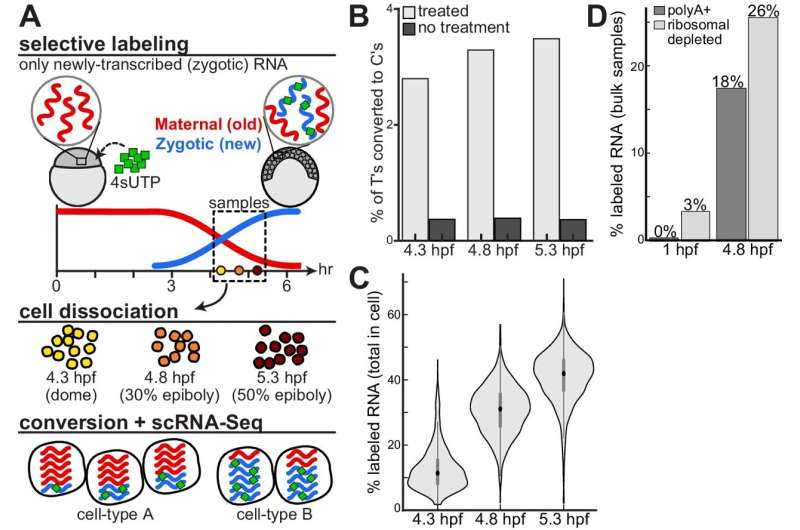
Lo sviluppo embrionale coinvolge cellule pluripotenti che assumono identità specializzate adottando particolari profili di espressione genica. Tuttavia, comprendere i contributi relativi della trascrizione e della degradazione dell'mRNA alla formazione di questi profili è stato impegnativo, in particolare all'interno di embrioni con identità cellulari diverse.
Nello studio, i ricercatori hanno utilizzato una tecnica chiamata sequenziamento dell’RNA a cellula singola insieme all’etichettatura metabolica per monitorare il modo in cui i geni vengono attivati e disattivati nel tempo negli embrioni di pesce zebra. Potevano distinguere l'mRNA che era stato creato nuovo (dall'embrione stesso) e l'mRNA che era già presente (dalla madre). Utilizzando modelli matematici, hanno misurato la velocità con cui i geni venivano attivati e disattivati in diversi tipi di cellule durante il loro sviluppo.
I risultati dello studio rivelano tassi regolatori molto diversi tra migliaia di geni. I ricercatori hanno osservato tassi di trascrizione e distruzione coordinati per molte trascrizioni e hanno collegato le differenze di degradazione a specifici elementi della sequenza. È importante sottolineare che hanno identificato differenze specifiche del tipo di cellula nella degradazione, inclusa la ritenzione selettiva delle trascrizioni materne all'interno delle cellule germinali primordiali e delle cellule dello strato avvolgente, due dei primi tipi di cellule specificati.
Il dottor Rabani, autore senior dello studio, ha commentato:"Il nostro studio fornisce un approccio quantitativo allo studio della regolazione dell'mRNA durante una risposta spazio-temporale dinamica. Questo lavoro apre nuove strade per comprendere i meccanismi molecolari alla base della determinazione del destino cellulare durante lo sviluppo embrionale. "
I risultati di questo studio contribuiscono a una comprensione più profonda della regolazione dell'mRNA e del suo ruolo nel modellare le identità cellulari durante lo sviluppo embrionale. Il gruppo di ricerca spera che il loro lavoro possa aprire la strada a studi futuri volti a svelare le complessità della regolazione dell'espressione genica in vari contesti biologici.
Ulteriori informazioni: Lior Fishman et al, Trascrizione dell'mRNA specifica per tipo cellulare e cinetica di degradazione nell'embriogenesi del pesce zebra da RNA-seq a singola cellula metabolicamente etichettato, Nature Communications (2024). DOI:10.1038/s41467-024-47290-9
Informazioni sul giornale: Comunicazioni sulla natura
Fornito dall'Università Ebraica di Gerusalemme