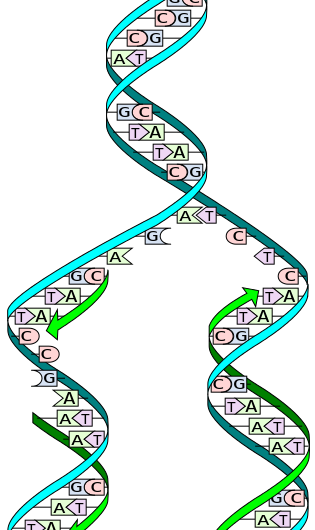
Durante la replica, i due fili sono separati. Ogni filamento della molecola di DNA originale funge quindi da stampo per la produzione della sua controparte complementare. Credito:Wikimedia Commons
L'analisi del DNA è utile per una serie di applicazioni vitali. Ciò include la diagnosi e il monitoraggio delle malattie, identificazione dei criminali, e studiare la funzione di un segmento mirato di DNA. Però, i metodi utilizzati per le analisi spesso richiedono più DNA di quanto potrebbe essere disponibile in un campione tipico. 'Perciò, l'amplificazione è necessaria, ma non sempre diretto. Il metodo di amplificazione o fotocopia più utilizzato è la reazione a catena della polimerasi (PCR). Un nuovo metodo PCR potrebbe aiutare il processo di amplificazione, e quindi sviluppare saggi robusti che in precedenza non sarebbero stati possibili.
L'acido desossiribonucleico (DNA) è una molecola che si trova nel nucleo delle cellule e porta le 'istruzioni' per lo sviluppo e il funzionamento degli organismi viventi. Viene spesso paragonato a una serie di progetti poiché contiene le istruzioni necessarie per costruire le celle. Queste istruzioni sono divise in segmenti lungo un filamento di DNA.
replica
Il DNA è costituito da una doppia elica di due filamenti complementari costituiti da nucleotidi, una struttura spesso paragonata a una scala. Quando è il momento di replicare, i due filamenti di DNA, o "lati" della scala, si srotolano e si separano. Un enzima chiamato DNA polimerasi legge i singoli filamenti e abbina basi complementari - i "pioli" della scala - al filamento originale. Nuovi filamenti sono prodotti su entrambi i lati del DNA originale, formando due doppie eliche di DNA identiche composte da un filamento originale e uno nuovo. Questo processo si verifica in tutti gli organismi viventi ed è la base dell'eredità biologica.
PCR
La replica può anche essere elaborata artificialmente. A volte chiamato "fotocopia molecolare, "la reazione a catena della polimerasi (PCR) è una tecnica veloce e relativamente poco costosa utilizzata per amplificare, o fare molte copie di, piccoli segmenti di DNA. Ciò è necessario perché i metodi utilizzati per analizzare il DNA o determinare la sequenza della coppia di basi del DNA richiedono più DNA di quello che potrebbe essere in un campione tipico. Quindi, l'obiettivo con la PCR è amplificare una regione specifica di un filamento di DNA.
Per eseguire la PCR, i due filamenti della doppia elica del DNA sono fisicamente separati applicando una temperatura elevata (93-98 °C) in un processo chiamato fusione del DNA. Nella seconda fase, la temperatura si abbassa e i due filamenti di DNA, ora separato, diventano modelli per i cosiddetti primer. I primer sono brevi molecole di DNA a singolo filamento, complementare alla regione del DNA mirata per l'amplificazione. Dopo aver abbassato la temperatura, i primer aggiunti al processo PCR trovano e si legano al suo bersaglio specifico. Un enzima chiamato DNA polimerasi allungherà - o costruirà - un nuovo filamento di DNA dal primer utilizzando la molecola di DNA a singolo filamento sottostante come modello.
Costruire una scala
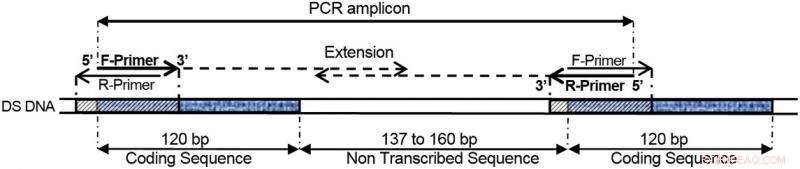
COMPAS-PCR che utilizza primer altamente complementari applicati a geni a ripetizione diretta del DNA ribosomiale 5S, come mostrato in questo esempio. Credito:Istituto norvegese per la ricerca sull'acqua (NIVA)
Come menzionato sopra, i primer sono brevi molecole di DNA a singolo filamento. Questi sono solitamente lunghi circa 20 nucleotidi. Questa stringa di nucleotidi, si attacca specificamente all'inizio del filamento modello mediante accoppiamento di basi - trovando le basi complementari. La DNA polimerasi è quindi in grado di aggiungere il successivo nucleotide complementare. La polimerasi continua ad aggiungere altri nucleotidi complementari al DNA stampo fino al completamento di un nuovo doppio filamento di DNA, o per usare la metafora sopra; viene costruita una nuova scala, con un lato originale e un lato nuovo.
I doppi primer consentono di definire con precisione l'area di una molecola di DNA da amplificare mediante la PCR. Questi due primer fiancheggianti specificano dove dovrebbe iniziare e finire la nuova catena.
Un piccolo cambio di paradigma
Una regola centrale fortemente ancorata nella mente dello scienziato molecolare è che i primer dovrebbero essere complementari solo alla sequenza bersaglio, e non tra di loro. Questo per evitare che i primer si utilizzino l'un l'altro come modelli e disabilitando così un ulteriore possibile coinvolgimento nell'amplificazione tradizionale del bersaglio.
In un recente studio, pubblicato sulla rivista scientifica PLOS One , Il ricercatore senior Marc Anglès d´Auriac con l'Istituto norvegese per la ricerca sull'acqua (NIVA) mostra che è possibile utilizzare primer altamente complementari e tuttavia evitare le sfortunate conseguenze sopra menzionate. Il nuovo metodo è stato denominato COMPAS-PCR, abbreviazione di COMPlementary Primer Asymmetric PCR.
In breve, Anglès d'Auriac ha osservato che i primer complementari tra loro e un bersaglio (tripla sovrapposizione) potrebbero ancora definire un prodotto di amplificazione quando fanno parte di un motivo o di una struttura di DNA ripetuto. Questo è mostrato nella figura sottostante. Ulteriore, l'ostacolo fisico dell'amplificazione del bersaglio dovuto alla complementarità dei primer è stato alleviato introducendo concentrazioni di primer asimmetriche. Asimmetrico in questo senso significa un numero dispari di molecole tra i due primer. Un primer è costituito da un basso numero di molecole, l'altro con un numero alto.
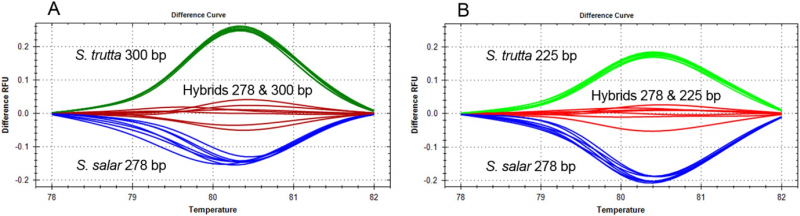
Analisi del melt ad alta risoluzione di due COMPAS-PCR duplex a tre primer per la differenziazione di S. salar, S. trutta e ibridi. Credito:Istituto norvegese per la ricerca sull'acqua (NIVA)
"Se si utilizzano concentrazioni uguali, come fai di solito per la PCR, i primer, presente in egual misura, si attaccheranno l'uno all'altro, " dice Anglès d'Auriac.
"Con concentrazioni asimmetriche, il primer in eccesso o ad alta concentrazione ha un numero di molecole non bloccato con il primer limitante, e quindi è disponibile per l'amplificazione del bersaglio."
Lavorare da soli
"Questo approccio controintuitivo isolerà, ma anche proteggere, il primer a bassa concentrazione, " dice Anglès d'Auriac.
Con il primer limitante bloccato, e quindi protetto, è possibile che il primer ad alta concentrazione agisca da solo, questo è, fare una copia a filamento singolo del DNA-bersaglio. Quando vengono prodotti sufficienti ampliconi a singolo filamento, possono servire come materiale modello per il primer a bassa concentrazione. Il primer limitante viene rilasciato, e la reazione passa alla classica amplificazione PCR esponenziale.
"Questo amplia le possibilità di applicazione della PCR per lo scienziato, Anglès d'Auriac chiarisce."
Strutture ripetitive
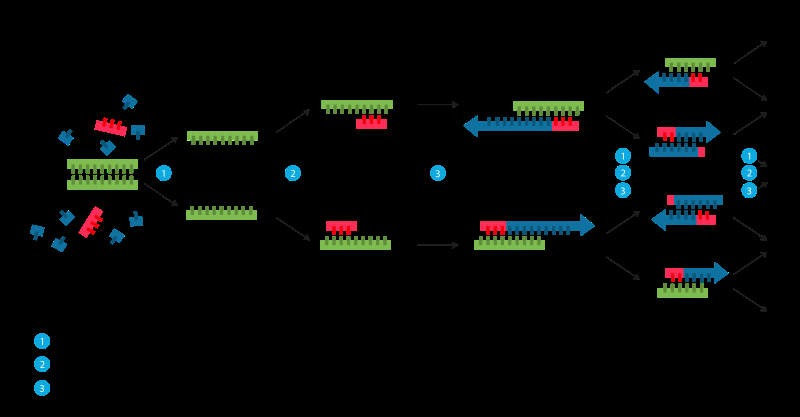
Reazione a catena della polimerasi (PCR) . Credito:Wikimedia
In molti organismi, una frazione significativa del DNA genomico è altamente ripetitiva, con oltre la metà della sequenza costituita da elementi ripetitivi negli esseri umani.
È stata infatti una struttura ripetitiva del DNA che ha indotto Anglès d'Auriac a pensare fuori dagli schemi e a proporre il principio COMPAS-PCR, un piccolo cambiamento di paradigma nel campo della fotocopia molecolare.
Nel processo di esecuzione della diagnosi basata sul DNA per i salmonidi, in particolare l'identificazione della trota fario (Salmo trutta) e del salmone atlantico (Salmo salar) strettamente imparentati, Anglès d'Auriac ha lottato per separare queste specie, compresi gli ibridi tra di loro. Un'identificazione rapida e accurata aiuterebbe a migliorare il monitoraggio e gli studi dell'ecosistema fluviale, poiché l'identificazione degli ibridi è importante per stimare la salute ecologica dei bacini idrografici. Dopo aver utilizzato COMPAS-PCR, Anglès d'Auriac è stato in grado di applicare un metodo di analisi del prodotto PCR chiamato analisi del fuso ad alta risoluzione nell'identificazione della trota fario, Salmone atlantico e loro ibridi in un unico test.
Anglès d'Auriac sottolinea tuttavia che il principio COMPAS-PCR non si limita all'identificazione delle specie ittiche. L'utilizzo di primer quasi completamente complementari mirati alla stessa sequenza può essere applicato a qualsiasi copia adiacente l'una all'altra, in motivi di DNA di interesse come sequenze bersaglio.
"Si dice che le sequenze ripetute del DNA comprendano più del 50% del genoma umano e sono presenti in molte famiglie di geni "domestici" come il gene ribosomiale 5S utilizzato in questo studio. Quindi, i principi generali COMPAS-PCR aiuteranno a sviluppare nuove strategie di amplificazione del DNA sfruttando queste strutture ripetute del DNA, "Conclude Marc Anglès d'Auriac.