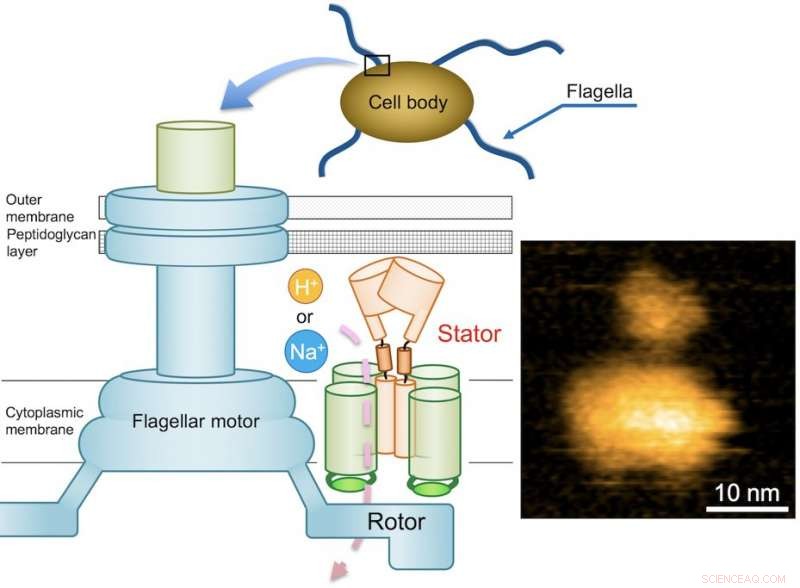
Fig. 1. Schema schematico del motore flagellare batterico e una tipica immagine AFM del complesso MotPS purificato. Credito:Università di Osaka
I batteri nuotano in molti modi diversi, e i motori che guidano il loro nuoto sono molto vari, implicando una risposta adattativa a un ambiente. Uno dei più comunemente identificati di tali motori è il flagello. Sebbene fornire motilità sia una caratteristica primaria dei flagelli, ad essi sono state attribuite anche proprietà adesive e altre proprietà.
Il motore flagellare è costituito da un rotore e da una dozzina di unità statoriche, ed è guidato dall'energia creata dagli ioni quando migrano attraverso la membrana cellulare. Mentre il motore flagellare di alcuni batteri d'acqua dolce ed enterici, come Salmonella, è alimentato da protoni (ioni idrogeno, h + ), che consiste di quattro copie della proteina MotA e due copie di MotB, i motori di altri batteri che vivono in ambienti di acqua salata sono alimentati da ioni sodio (Na + ).
Nel caso di Bacillus subtilis (B. subtilis), un batterio che si trova comunemente nel terreno, il motore flagellare ha due distinti tipi di unità statoriche:MotAB di tipo H+ e MotPS di tipo Na+. Mentre i componenti sono identificati, il meccanismo che alimenta il motore flagellare rimane sconosciuto. Questo mistero è ulteriormente aggravato dal fatto che i flagelli non sono chiaramente visibili al microscopio ottico. Queste sfide hanno spinto un team di ricercatori dell'Università di Osaka a cercare di chiarire il meccanismo sconosciuto. Hanno riportato i loro risultati in Progressi scientifici .
"Analisi precise della dinamica strutturale del motore flagellare utilizzando la microscopia a forza atomica ad alta velocità (HS-AFM) combinata con l'analisi mutazionale hanno fornito approfondimenti sul meccanismo di generazione della coppia del motore flagellare, ", afferma la prima autrice dello studio Naoya Terahara. "Abbiamo fatto osservazioni in tempo reale dei cambiamenti strutturali indotti dal Na+ al dominio di MotS che lega il peptidoglicano, un polimero costituito da zuccheri e amminoacidi nei batteri".
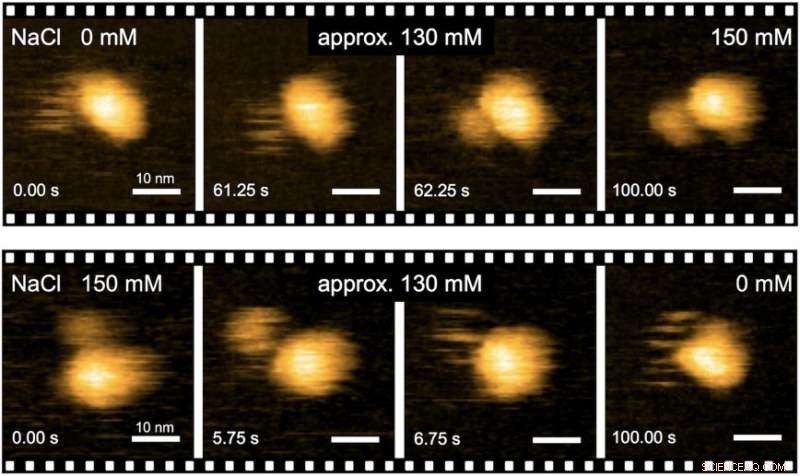
Fig. 2. Imaging in tempo reale delle transizioni strutturali indotte da Na+ del complesso MotPS mediante HS-AFM. Credito:Università di Osaka
Per capire come il complesso proteico MotPS risponde ai cambiamenti nell'ambiente, i ricercatori hanno prima caratterizzato la relazione coppia-velocità del motore B. subtilis su un'ampia gamma di concentrazioni di Na+. La velocità massima del motore è stata ridotta da circa 200 a 80 Hz quando la concentrazione esterna di Na+ è stata aumentata da 0 a 200 mM, anche se la coppia di stallo non è stata cambiata affatto.
In particolare, quando il dominio di MotB che si lega al peptidoglicano è stato sostituito da quello di MotS, il motore chimerico ha mantenuto la stessa curva coppia-velocità del motore MotAB in presenza di ioni Na+ ma non in assenza. Ciò significa che Na+ è il driver principale dell'assemblaggio e dell'attivazione del motore flagellare, indipendentemente dalla sua composizione.
"In assenza di ioni sodio, MotPS esiste come forma inattiva nella membrana citoplasmatica perché MotSc, il dominio periplasmatico C-terminale di MotS, adotta una conformazione spiegata. Quando la concentrazione di ioni sodio aumenta, il legame degli ioni a MotSc induce il ripiegamento del dominio PGB e ne facilita la dimerizzazione in modo altamente cooperativo, " dice l'autore corrispondente Tohru Minamino.
Attraverso questo studio, il team ha anche dimostrato la fattibilità dell'uso di HS-AFM combinato con l'analisi mutazionale per studiare strutture batteriche complesse, che possono fornire preziose informazioni per la produzione industriale di prodotti commercialmente validi, come proteasi e terapeutici, come nel caso del B. subtilis.
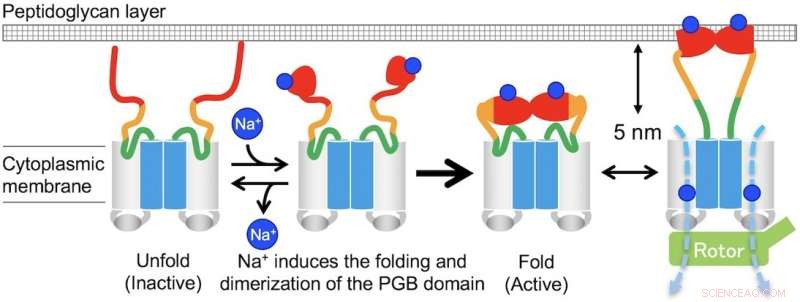
Fig. 3. Modello per il montaggio e lo smontaggio indotto da Na+ del complesso statorico MotPS. Credito:Università di Osaka