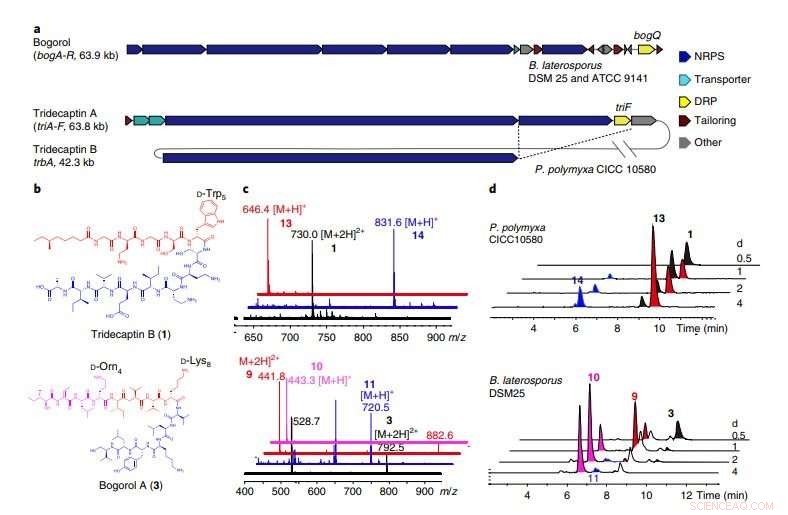
un, I BGC di bogorol da B. laterosporus DSM 25 e ATCC 9141, così come tridecaptina A e tridecaptina B da P. polymyxa CICC 10580. BogQ dal ceppo DSM 25 e quello dal ceppo ATCC 9141 condividono l'85% di identità di sequenza di amminoacidi. Le linee tratteggiate che si intersecano indicano geni condivisi da due cluster di geni all'interno dello stesso ospite (Figura 10 supplementare). B, Strutture dei DNRP tridecaptina B e bogorol A, con i motivi di riconoscimento DRP evidenziati. C, Sovrapposizione impilata degli spettri di massa (ionizzazione elettrospray) dei composti progenitori (nero), frammenti C-terminali (blu), e frammenti N-terminali (rosso). I dati sono rappresentativi di due esperimenti indipendenti. Superiore, tridecaptina B; parte inferiore, bogorol A. d, Analisi nel tempo dei corrispondenti composti prodotti da P. polymyxa CICC 10580 (in alto) e B. laterosporus DSM 25 (in basso) a diversi tempi di fermentazione (rappresentativo di tre esperimenti indipendenti). Credito:Divisione di scienze della vita, HKUST
Antibiotici peptidici non ribosomiali, compresa la polimixina, vancomicina, e teixobactina, la maggior parte dei quali contiene D-amminoacidi, sono altamente efficaci contro i batteri multiresistenti. Però, l'uso eccessivo di antibiotici ignorando il rischio di insorgenza di resistenza ha inesorabilmente portato alla diffusione diffusa di batteri resistenti. Chiarire i meccanismi poco conosciuti di resistenza agli antibiotici peptidici è fondamentale quando si implementano gli antibiotici peptidici e migliorerebbe efficacemente l'efficienza.
In un recente studio, un gruppo di scienziati della Hong Kong University of Science and Technology rivela sia la distribuzione diffusa che il potenziale di resistenza ad ampio spettro delle peptidasi D-stereospecifiche, fornendo un potenziale indicatore precoce della resistenza agli antibiotici peptidici non ribosomiali.
I loro risultati sono stati pubblicati sulla rivista Natura chimica biologia il 26 febbraio, 2018.
"Abbiamo applicato un approccio a 5, 585 genomi batterici completi che coprono l'intero dominio dei batteri, " disse Pei-Yuan Qian, professore a contratto della Divisione di Scienze della Vita, HKUST, e autore principale dell'articolo. "Con successive analisi chimiche ed enzimatiche, abbiamo dimostrato un meccanismo di resistenza agli antibiotici peptidici non ribosomiali che si basa sulla scissione idrolitica da parte delle peptidasi D-stereo specifiche".
Il team ha identificato una famiglia di peptidasi di resistenza D-stereospecifica (DRP) che sono filogeneticamente ampiamente distribuite in natura. La scoperta dei DRP è risultata essere coinvolta nella lotta agli antibiotici ampiamente distribuiti contenenti D-aa per la sopravvivenza del loro ospite, che è stato convalidato sperimentalmente da una combinazione di editing genetico basato su CRISPR/Cas9, analisi chimiche ed enzimatiche.
"Dato il potenziale dei DRP per la resistenza ad ampio spettro e il loro potenziale per colpire antibiotici clinicamente importanti contenenti D-aa, è probabile che questi geni di resistenza ampiamente distribuiti siano particolarmente pericolosi se vengono trasferiti a patogeni opportunisti, " ha detto il Prof. Qian. "I DRP trovati in natura costituiscono solo la punta dell'iceberg, che porterà a un'intensa ricerca sull'uso e lo sviluppo di antibiotici peptidici per combattere la resistenza agli antibiotici".
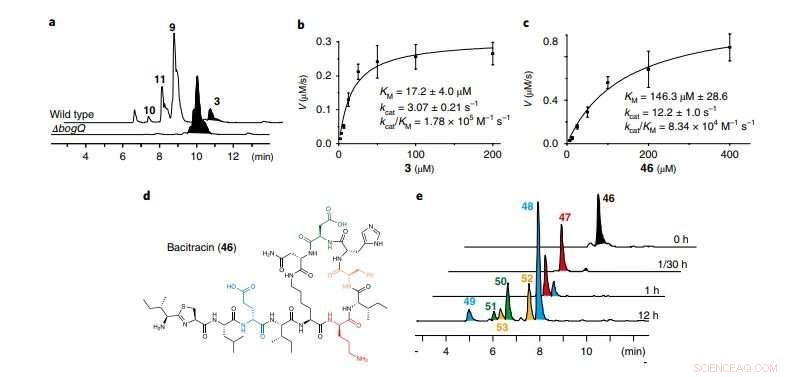
un, Tracce LC-MS che confrontano B. laterosporus ATCC 9141 wild-type e il mutante ΔbogQ (rappresentante di tre esperimenti indipendenti). B, C, Analisi cinetiche dell'idrolisi catalizzata da BogQ di bogorol A (b; 3) e bacitracina (c; 46) v, velocità di reazione I dati sono significa?±?s.d.; n=?3 esperimenti indipendenti. D, Struttura dell'antibiotico bacitracina DNRP; i colori evidenziano i siti di scollatura di BogQ. e, Tracce LC-MS di saggi in vitro di BogQ (2.0?μM) contro 46 (200?μM; rappresentativo di due esperimenti indipendenti). I prodotti di scissione nel corso del tempo (47-53) di 46 sono etichettati utilizzando lo stesso codice colore dei loro siti di scissione d-aa in d. Per i modelli di scissione enzimatica, vedere la Fig. 22 supplementare. Credito:Divisione di scienze della vita, HKUST